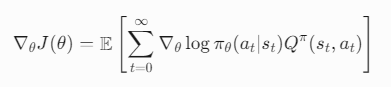The panel of normals is applied by replacing the germline read depth of the sample with the median read depth of samples with the same genotype in our panel.
1.解释:
panel of normal 正常组织,用于识别技术噪音
germline read depth: 胚系测序深度。根据上下文,是血液或者唾液的DNA。
same genotype: 相同基因型,比如AA, AB, BB
2.步骤
(1)识别肿瘤样本的胚系位点(和血液一致的位点,这些位点的深度代表 正常细胞 的测序覆盖情况)
(2)在正常样本中匹配相同的基因型的样本
肿瘤中某个SNP位点的基因型是AB,就找也是AB的正常样本。
(3)计算正常样本的中位数深度
正常样品中5个AB基因型样本的测序深度分别为: 100x, 105x, 110x, 115x, 120x, 中位数为 110x
(4)替换肿瘤样本的原始深度
肿瘤样本的原始深度: 95x(可能因技术偏差偏低)
校正后深度: 110x (来自同基因型正常样品的深度中位数)
(5)重复所有位点,生成校正后的深度数据
对所有胚系位点执行上述操作,最终得到 技术偏差校正后的测序深度。
3.为什么这么做?技术原理
消除个体间自然变异:消除测序深度、建库效率的差异波动
提高肿瘤纯度计算准确性:校正后更接近真实生物学状态,避免技术因素干扰肿瘤细胞比例(purity)计算。
优化拷贝数变异(CNV)检测:深度标准化后,CNV信号(如扩增/缺失)更易识别。
4.实际应用示例
(1)场景:检测肿瘤样本的LOH(杂合性缺失)
原始数据: 肿瘤样本在某个位点 基因型AB,原始深度=80x(A=60x, B=20x); B-allele频率(BAF)=20/(60+20)=0.25(偏离预期的0.5,可能提示LOH)
正常样本校正:正常样本中AB基因型的样本测序深度中位数=100x;校正后肿瘤样本深度=100x(假设A=75x,B=25x, BAF=0.25任保留)
结果解读:校正后的深度拍出来技术偏差,确认BAF异常(0.25)是真实的LOH信号,而非测序噪音。
5.注意事项
- PoN样本量要求:通常需要 ≥50个正常样本,以保证中位值的代表性
- 基因型匹配严格性:需确保匹配的基因型可靠(如通过GATK等工具高置信度call)。
- 肿瘤纯度影响:高纯度肿瘤样本中,胚系位点的深度可能已被肿瘤细胞稀释,需结合B-allele频率(BAF)综合分析。
总结
这句话描述的是 通过PoN标准化肿瘤样本测序深度 的关键步骤:
- 用PoN中 相同基因型正常样本的中位深度 替换原始深度。
- 消除技术噪声,突出真实的生物学变异(如CNV、LOH)。
- 该方法在GATK、CNVkit、VarScan2等分析流程中广泛应用。
Ref
- https://pmc.ncbi.nlm.nih.gov/articles/PMC11300791/