Ambitious goal !!
任务:molecule captioning and text-guided de novo molecule generation.
论文链接:https://arxiv.org/abs/2204.11817
代码链接:GitHub - blender-nlp/MolT5: Associated Repository for "Translation between Molecules and Natural Language"
huggingface:laituan245 (Tuan Lai)
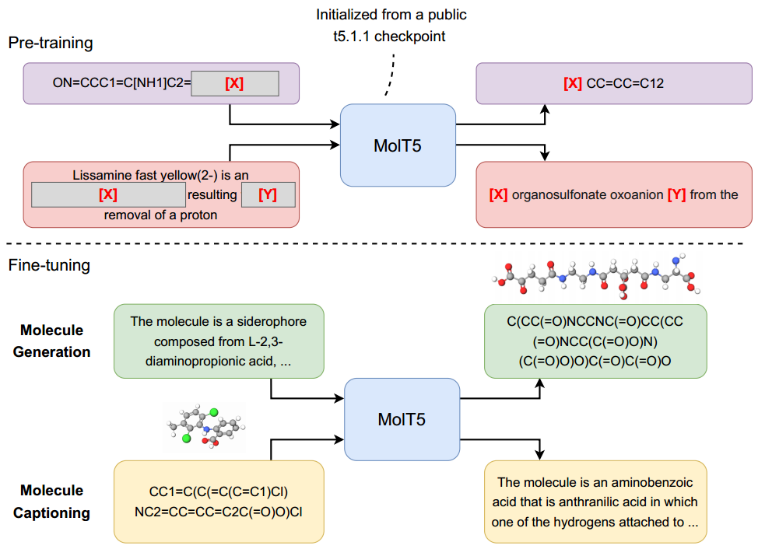
MolT5 – Multimodal Text-Molecule Representation Model
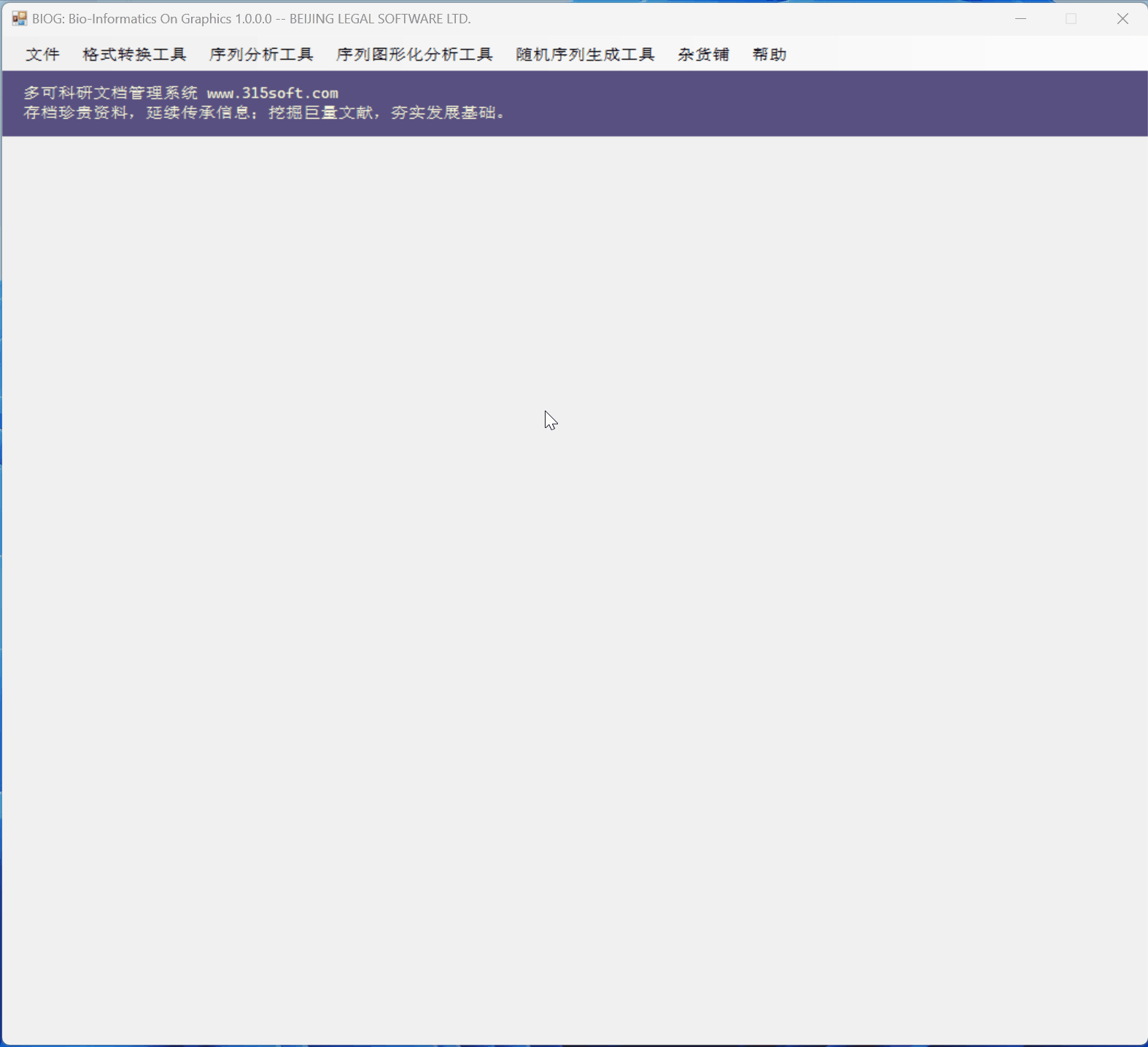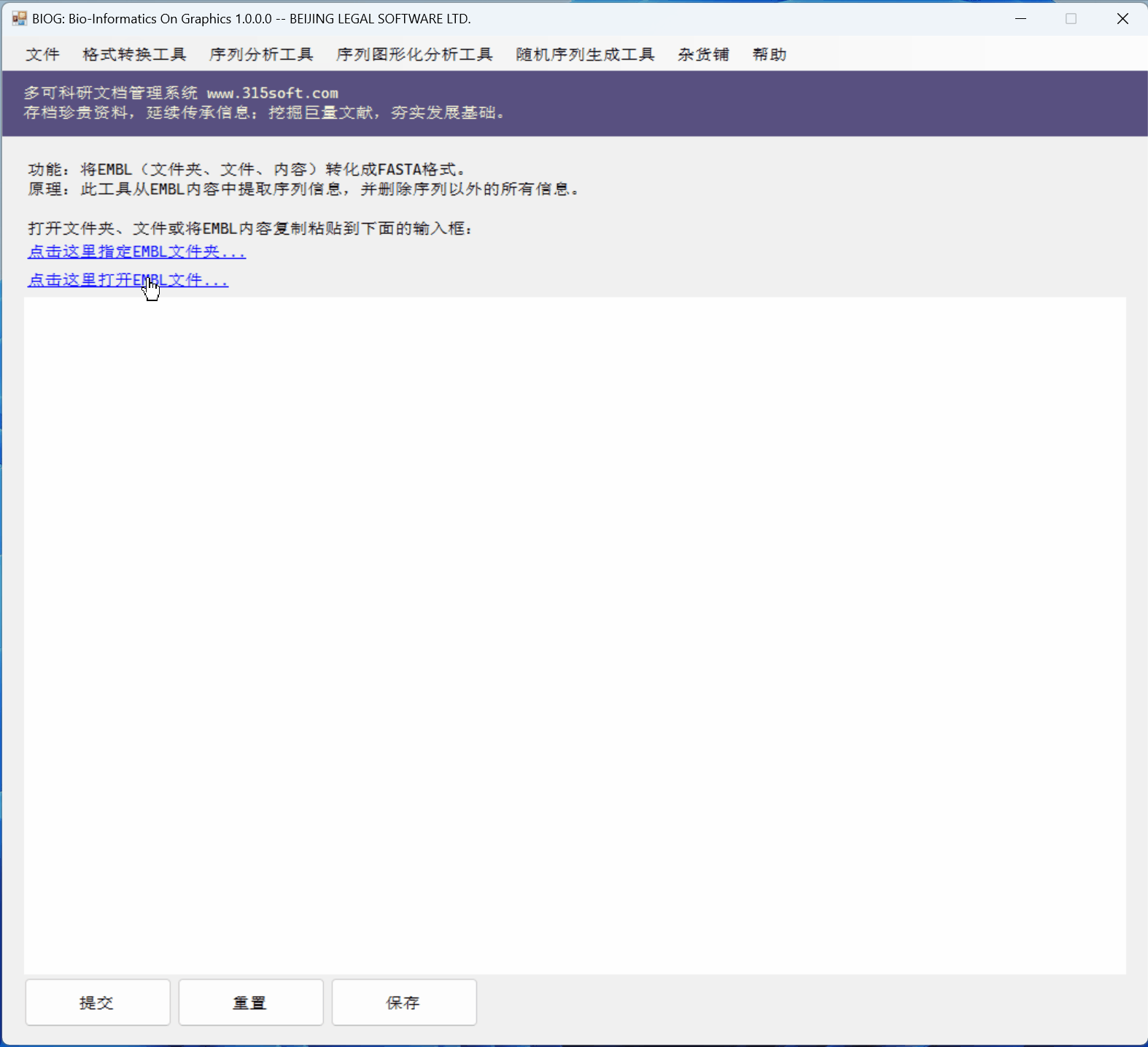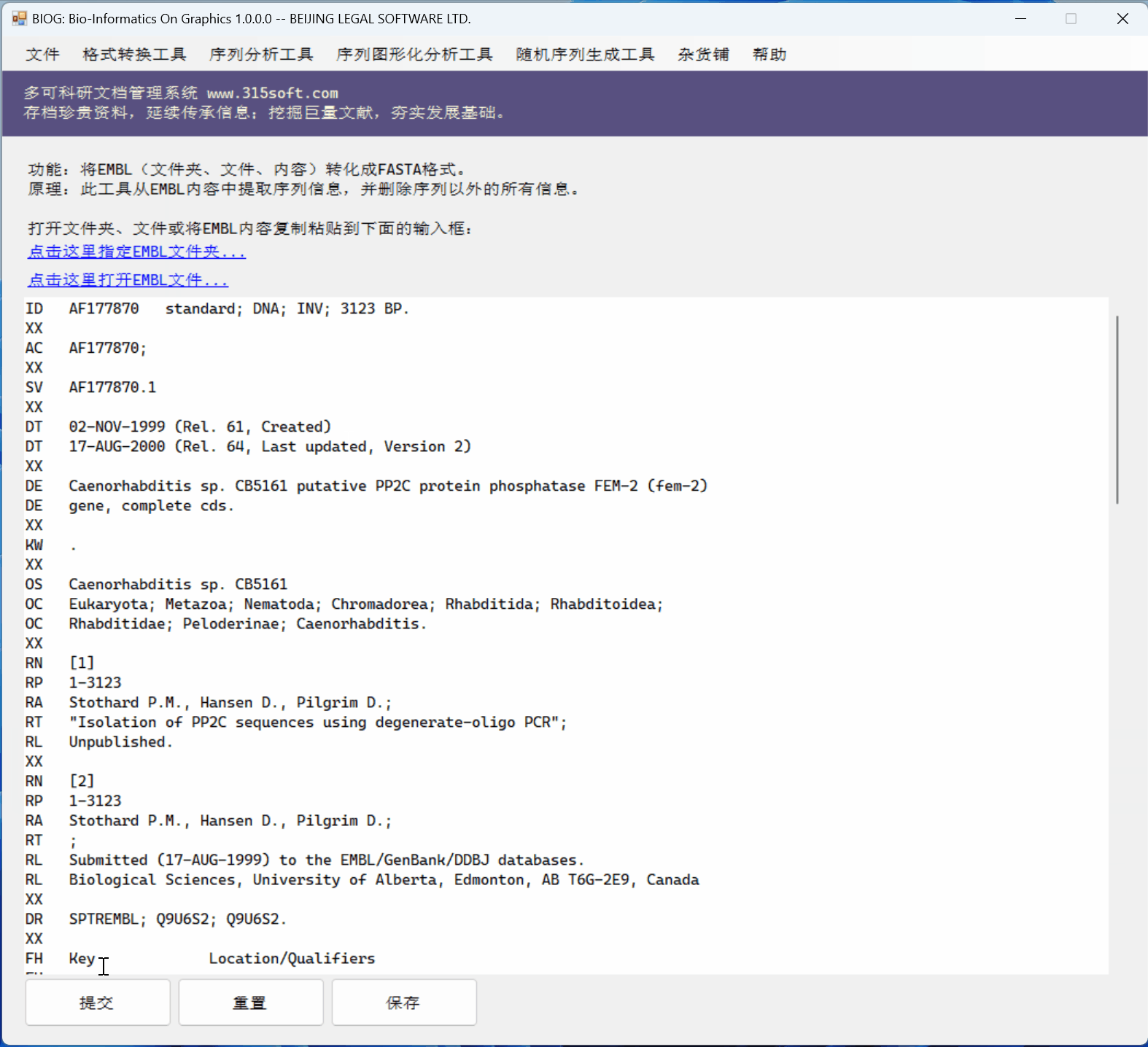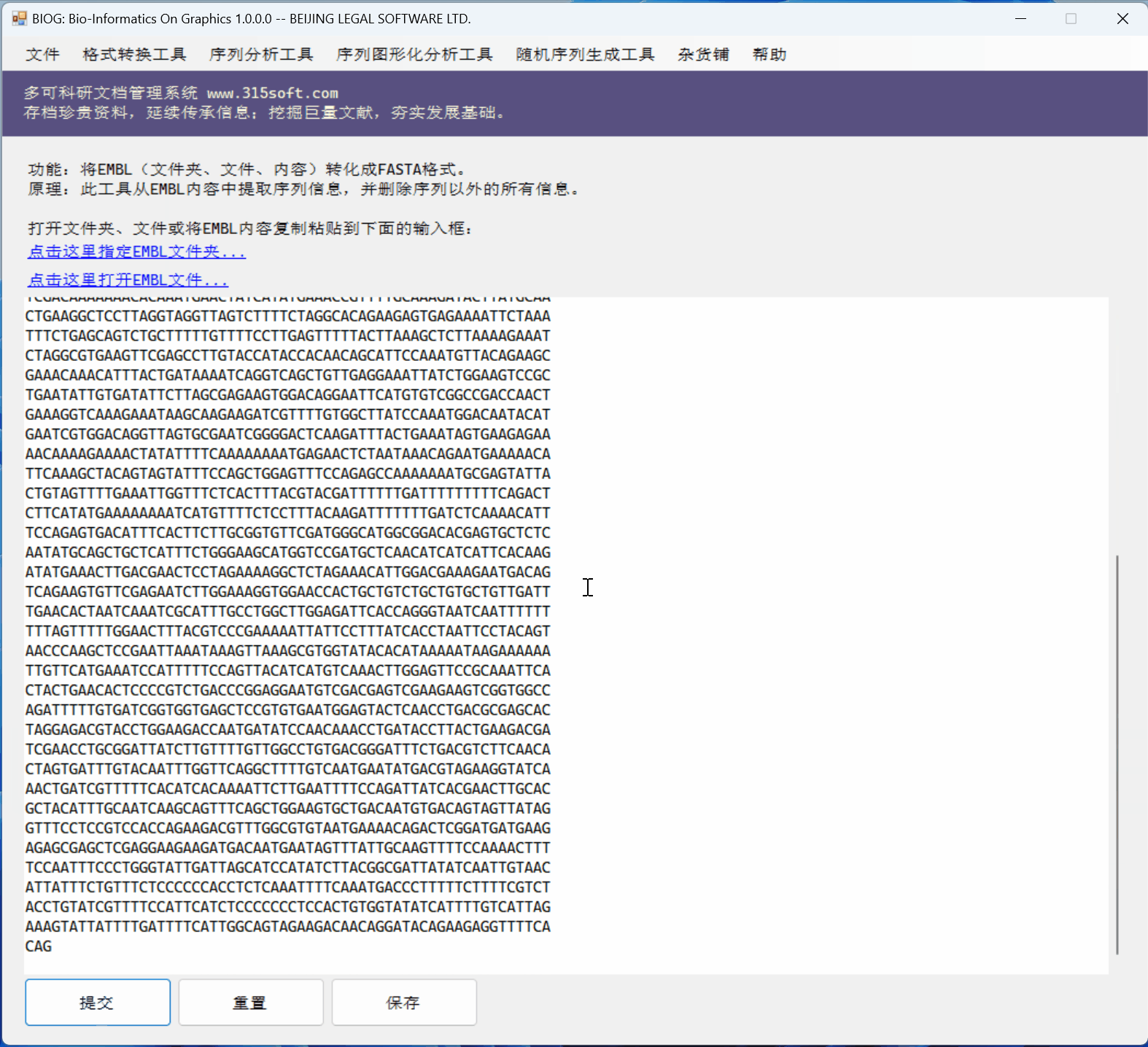
首先使用T5.1.12的public checkpoint初始化transformer encoder-decoder,T5.1.12是T5的改进版本。使用“replace corrupted spans”对模型进行预训练。
对于每个序列,随机选择序列中的一些单词进行破坏。损坏token的每个连续范围都被一个sentinel token (替换(如图3中的[X]和[Y]所示)。接下来预测dropped-out spans
预训练阶段基本上是在来自两种不同语言的两个单语语料库上训练单一语言模型,并且两个语料库之间没有明确的对齐。这种方法类似于一些多语言语言模型,如mBERT、mBART的预训练。
预训练后,对预训练模型进行微调。在分子生成中,输入是描述,输出是目标分子的SMILES。在分子标题中,输入是某些分子的SMILES字符串,输出是描述输入分子的caption。
Evaluation Metrics
1、Text2Mol Metric
【Text2mol: Cross-modal molecule retrieval with natural language queries.】:检索模型,根据其文本描述对与分子相似度进行排序。排序函数使用两个embeddings之间的余弦相似度,可以用于评估真实分子/描述与生成的描述/分子(分别)之间的相似性。
2、Evaluating Molecule Captioning
传统上,caption任务是通过自然语言生成指标来评估的,如BLEU、ROUGE和METEOR。与COCO 等每幅图像有几个标题的标题任务不同,在这项任务中,只有一个参考captioning(但是一个分子可以有多个SMILES string?)。这使得这些指标不那么有效,特别是因为有许多不重叠的方法来描述一个分子。然而,为了比较,仍然报告这些分数(例如,汇总句子级METEOR分数)。
3、Evaluating Text-Based de Novo Molecule Generation
希望生成的分子与输入文本匹配,而不是普遍地多样化(novelty 、scaffold similarity等),考虑度量生成的分子到基本真实分子或基本真实描述的距离的度量。
采用了三个指纹指标:MACCS FTS、RDK FTS和Morgan FTS,还报告SMILES-strings匹配、Levenshtein距离和SMILES BLEU分数。
对于使用SMILES-string的模型,生成的分子可能在语法上无效。因此,也关注validity,计算能被RDKIT处理的分子百分比(不能处理就invalid)。
Experiments and Results
Pretraining Data
MolT5的预训练阶段需要两个单语语料库:一个由自然语言文本组成,另一个由分子表征组成。使用“Colossal Clean crawl Corpus”(C4) 作为文本模态的预训练数据集。对于分子,直接利用Chemformer中使用的1亿个SMILES字符串(从ZINC-15中搜集)
Finetuning and Evaluation Data
ChEBI-20(33,010 molecule-description pairs)80/10/10% train/validation/test,为了迫使模型关注描述的语义,将分子的名称替换为“该分子是……”(例如,“该分子是从……中分离出来的有机二硫化物”)。
Results




Limitations
由于这项工作的重点是大型语言模型的新应用程序,因此许多相同的限制也适用于此。也就是说,该模型是在从互联网收集的大型数据集上训练的,因此它可能包含意想不到的偏差。限制是使用SMILES字符串——最近的工作提出了一种具有有效性保证的字符串表示。在实践中,发现这对于预训练的T5检查点(从计算的角度来看这很重要)的效果很差。还注意到,ChEBI-20中的一些化合物可能会在默认的SELFIES实现中导致有效性问题。
Usage
# https://huggingface.co/laituan245
from transformers import T5Tokenizer, T5ForConditionalGeneration
tokenizer = T5Tokenizer.from_pretrained("laituan245/molt5-large-caption2smiles", model_max_length=512)
model = T5ForConditionalGeneration.from_pretrained('laituan245/molt5-large-caption2smiles')
input_text = 'The molecule is a monomethoxybenzene that is 2-methoxyphenol substituted by a hydroxymethyl group at position 4. It has a role as a plant metabolite. It is a member of guaiacols and a member of benzyl alcohols.'
input_ids = tokenizer(input_text, return_tensors="pt").input_ids
outputs = model.generate(input_ids, num_beams=5, max_length=512)
print(tokenizer.decode(outputs[0], skip_special_tokens=True))