>生信老白写的基础代码.fasta
MAYBENOANYUSAGE
1 EMBL
1.1 EMBL组织
欧洲分子生物学实验室EMBL(European Molecular Biology Laboratory)1974年由欧洲14个国家加上亚洲的以色列共同发起建立,现在由欧洲30个成员国政府支持组成,目的在于促进欧洲国家之间的合作来发展分子生物学的基础研究和改进仪器设备、教育工作等。EMBL分为7个部分:结构、分化、物理仪器、生化仪器、生物仪器、计算机和应用数学。它的宗旨是:从事结构分子生物学及分子医学方面的基础研究;为科学家、学生及访问学者提供高层次的培训;为成员国的科学家提供必需的科研服务;在生命科学领域开发新型的科研仪器及研究方法;积极参与生物技术的转化及应用。
1.2 EMBL 文件
DNA和通过多种DNA序列的程序使用的蛋白质序列文件格式;包含序列数据,与关于序列,如名称,类型和描述信息一起;可以存储多个序列。
EMBL格式将序列及其注释存储在一起。注释部分的开头用一行以单词“ID”开头。序列的开始部分用一个以单词“SQ”开头的行来标记。“//”(终止符)行也不包含数据或注释,并指定条目的结尾。
EMBL文件可能以.EMBL或.txt扩展名结尾。
格式规范
State: Experimental as of 0.5.1-dev.
前面的节 FH (Feature Header)
之前的所有章节 FH (Feature Header) 将被读入 metadata . 节的头及其内容作为键值对存储在中 metadata . 对于 RN (Reference Number) 节,其值以列表形式存储,因为在一个EMBL记录中通常有多个引用节。
FT 部分
见 Genbank FEATURES section
SQ 部分
中的序列 SQ 对于从ENA下载的EMBL文件,节始终是小写的。对于RNA分子, t (胸腺嘧啶),而不是 u (尿嘧啶)用于序列中。所有EMBL编写器在编写EMBL文件时都遵循这些约定。
EMBL文件示例:
>>>
embl_str = '''
ID X56734; SV 1; linear; mRNA; STD; PLN; 1859 BP.
XX
AC X56734; S46826;
XX
DT 12-SEP-1991 (Rel. 29, Created)
DT 25-NOV-2005 (Rel. 85, Last updated, Version 11)
XX
DE Trifolium repens mRNA for non-cyanogenic beta-glucosidase
XX
KW beta-glucosidase.
XX
OS Trifolium repens (white clover)
OC Eukaryota; Viridiplantae; Streptophyta; Embryophyta; Tracheophyta;
OC Spermatophyta; Magnoliophyta; eudicotyledons; Gunneridae;
OC Pentapetalae; rosids; fabids; Fabales; Fabaceae; Papilionoideae;
OC Trifolieae; Trifolium.
XX
RN [5]
RP 1-1859
RX DOI; 10.1007/BF00039495.
RX PUBMED; 1907511.
RA Oxtoby E., Dunn M.A., Pancoro A., Hughes M.A.;
RT "Nucleotide and derived amino acid sequence of the cyanogenic
RT beta-glucosidase (linamarase) from white clover
RT (Trifolium repens L.)";
RL Plant Mol. Biol. 17(2):209-219(1991).
XX
RN [6]
RP 1-1859
RA Hughes M.A.;
RT ;
RL Submitted (19-NOV-1990) to the INSDC.
RL Hughes M.A., University of Newcastle Upon Tyne, Medical School,
RL Newcastle
RL Upon Tyne, NE2 4HH, UK
XX
DR MD5; 1e51ca3a5450c43524b9185c236cc5cc.
XX
FH Key Location/Qualifiers
FH
FT source 1..1859
FT /organism="Trifolium repens"
FT /mol_type="mRNA"
FT /clone_lib="lambda gt10"
FT /clone="TRE361"
FT /tissue_type="leaves"
FT /db_xref="taxon:3899"
FT mRNA 1..1859
FT /experiment="experimental evidence, no additional
FT details recorded"
FT CDS 14..1495
FT /product="beta-glucosidase"
FT /EC_number="3.2.1.21"
FT /note="non-cyanogenic"
FT /db_xref="GOA:P26204"
FT /db_xref="InterPro:IPR001360"
FT /db_xref="InterPro:IPR013781"
FT /db_xref="InterPro:IPR017853"
FT /db_xref="InterPro:IPR033132"
FT /db_xref="UniProtKB/Swiss-Prot:P26204"
FT /protein_id="CAA40058.1"
FT /translation="MDFIVAIFALFVISSFTITSTNAVEASTLLDIGNLSRS
FT SFPRGFIFGAGSSAYQFEGAVNEGGRGPSIWDTFTHKYPEKIRDGSNADITV
FT DQYHRYKEDVGIMKDQNMDSYRFSISWPRILPKGKLSGGINHEGIKYYNNLI
FT NELLANGIQPFVTLFHWDLPQVLEDEYGGFLNSGVINDFRDYTDLCFKEFGD
FT RVRYWSTLNEPWVFSNSGYALGTNAPGRCSASNVAKPGDSGTGPYIVTHNQI
FT LAHAEAVHVYKTKYQAYQKGKIGITLVSNWLMPLDDNSIPDIKAAERSLDFQ
FT FGLFMEQLTTGDYSKSMRRIVKNRLPKFSKFESSLVNGSFDFIGINYYSSSY
FT ISNAPSHGNAKPSYSTNPMTNISFEKHGIPLGPRAASIWIYVYPYMFIQEDF
FT EIFCYILKINITILQFSITENGMNEFNDATLPVEEALLNTYRIDYYYRHLYY
FT IRSAIRAGSNVKGFYAWSFLDCNEWFAGFTVRFGLNFVD"
XX
SQ Sequence 1859 BP; 609 A; 314 C; 355 G; 581 T; 0 other;
aaacaaacca aatatggatt ttattgtagc catatttgct ctgtttgtta ttagctcatt
cacaattact tccacaaatg cagttgaagc ttctactctt cttgacatag gtaacctgag
tcggagcagt tttcctcgtg gcttcatctt tggtgctgga tcttcagcat accaatttga
aggtgcagta aacgaaggcg gtagaggacc aagtatttgg gataccttca cccataaata
tccagaaaaa ataagggatg gaagcaatgc agacatcacg gttgaccaat atcaccgcta
caaggaagat gttgggatta tgaaggatca aaatatggat tcgtatagat tctcaatctc
ttggccaaga atactcccaa agggaaagtt gagcggaggc ataaatcacg aaggaatcaa
atattacaac aaccttatca acgaactatt ggctaacggt atacaaccat ttgtaactct
ttttcattgg gatcttcccc aagtcttaga agatgagtat ggtggtttct taaactccgg
tgtaataaat gattttcgag actatacgga tctttgcttc aaggaatttg gagatagagt
gaggtattgg agtactctaa atgagccatg ggtgtttagc aattctggat atgcactagg
aacaaatgca ccaggtcgat gttcggcctc caacgtggcc aagcctggtg attctggaac
aggaccttat atagttacac acaatcaaat tcttgctcat gcagaagctg tacatgtgta
taagactaaa taccaggcat atcaaaaggg aaagataggc ataacgttgg tatctaactg
gttaatgcca cttgatgata atagcatacc agatataaag gctgccgaga gatcacttga
cttccaattt ggattgttta tggaacaatt aacaacagga gattattcta agagcatgcg
gcgtatagtt aaaaaccgat tacctaagtt ctcaaaattc gaatcaagcc tagtgaatgg
ttcatttgat tttattggta taaactatta ctcttctagt tatattagca atgccccttc
acatggcaat gccaaaccca gttactcaac aaatcctatg accaatattt catttgaaaa
acatgggata cccttaggtc caagggctgc ttcaatttgg atatatgttt atccatatat
gtttatccaa gaggacttcg agatcttttg ttacatatta aaaataaata taacaatcct
gcaattttca atcactgaaa atggtatgaa tgaattcaac gatgcaacac ttccagtaga
agaagctctt ttgaatactt acagaattga ttactattac cgtcacttat actacattcg
ttctgcaatc agggctggct caaatgtgaa gggtttttac gcatggtcat ttttggactg
taatgaatgg tttgcaggct ttactgttcg ttttggatta aactttgtag attagaaaga
tggattaaaa aggtacccta agctttctgc ccaatggtac aagaactttc tcaaaagaaa
ctagctagta ttattaaaag aactttgtag tagattacag tacatcgttt gaagttgagt
tggtgcacct aattaaataa aagaggttac tcttaacata tttttaggcc attcgttgtg
aagttgttag gctgttattt ctattatact atgttgtagt aataagtgca ttgttgtacc
agaagctatg atcataacta taggttgatc cttcatgtat cagtttgatg ttgagaatac
tttgaattaa aagtcttttt ttattttttt aaaaaaaaaa aaaaaaaaaa aaaaaaaaa
//提取
DNA 对象:
>>>
import io
from skbio import DNA, RNA, Sequence
embl = io.StringIO(embl_str)
dna_seq = DNA.read(embl)
dna_seq
DNA
----------------------------------------------------------------------
Metadata:
'ACCESSION': 'X56734; S46826;'
'CROSS_REFERENCE': <class 'list'>
'DATE': <class 'list'>
'DBSOURCE': 'MD5; 1e51ca3a5450c43524b9185c236cc5cc.'
'DEFINITION': 'Trifolium repens mRNA for non-cyanogenic beta-
glucosidase'
'KEYWORDS': 'beta-glucosidase.'
'LOCUS': <class 'dict'>
'REFERENCE': <class 'list'>
'SOURCE': <class 'dict'>
'VERSION': 'X56734.1'
Interval metadata:
3 interval features
Stats:
length: 1859
has gaps: False
has degenerates: False
has definites: True
GC-content: 35.99%
----------------------------------------------------------------------
0 AAACAAACCA AATATGGATT TTATTGTAGC CATATTTGCT CTGTTTGTTA TTAGCTCATT
60 CACAATTACT TCCACAAATG CAGTTGAAGC TTCTACTCTT CTTGACATAG GTAACCTGAG
1740 AGAAGCTATG ATCATAACTA TAGGTTGATC CTTCATGTAT CAGTTTGATG TTGAGAATAC
1800 TTTGAATTAA AAGTCTTTTT TTATTTTTTT AAAAAAAAAA AAAAAAAAAA AAAAAAAAA
既然这是一个mRNA分子,我们可能想把它解读为 RNA . 就像EMBL文件通常有 t 而不是 u 在序列中,我们可以把它理解为 RNA 通过转换 t 到 u :
>>>
embl = io.StringIO(embl_str)
rna_seq = RNA.read(embl)
rna_seq
RNA
----------------------------------------------------------------------
Metadata:
'ACCESSION': 'X56734; S46826;'
'CROSS_REFERENCE': <class 'list'>
'DATE': <class 'list'>
'DBSOURCE': 'MD5; 1e51ca3a5450c43524b9185c236cc5cc.'
'DEFINITION': 'Trifolium repens mRNA for non-cyanogenic beta-
glucosidase'
'KEYWORDS': 'beta-glucosidase.'
'LOCUS': <class 'dict'>
'REFERENCE': <class 'list'>
'SOURCE': <class 'dict'>
'VERSION': 'X56734.1'
Interval metadata:
3 interval features
Stats:
length: 1859
has gaps: False
has degenerates: False
has definites: True
GC-content: 35.99%
----------------------------------------------------------------------
0 AAACAAACCA AAUAUGGAUU UUAUUGUAGC CAUAUUUGCU CUGUUUGUUA UUAGCUCAUU
60 CACAAUUACU UCCACAAAUG CAGUUGAAGC UUCUACUCUU CUUGACAUAG GUAACCUGAG
1740 AGAAGCUAUG AUCAUAACUA UAGGUUGAUC CUUCAUGUAU CAGUUUGAUG UUGAGAAUAC
1800 UUUGAAUUAA AAGUCUUUUU UUAUUUUUUU AAAAAAAAAA AAAAAAAAAA AAAAAAAAA
2 FASTA
2.1 FASTA格式
fasta格式是一种基于文本用于表示核酸序列或多肽序列的格式。其中核酸或氨基酸均以单个字母来表示,且允许在序列前添加序列名及注释。该格式已成为生物信息学领域的一项标准。
fasta格式,又称Pearson格式,主要发明人是威廉·皮尔森(William Raymond Pearson)和戴维德.李普曼(David J. Lipman),威廉·雷蒙德·皮尔森是美国弗吉尼亚大学的生物化学与分子遗传学教授,戴维德.李普曼在1989年至2017年期间担任NCBI主任,他也是BLAST算法的发明人之一。1985年3月,双方在科学期刊Science上合作发表了相关成果。
fasta格式文件的第一行是由大于号“>”(较常用)或分号“;”打头的任意文字说明,用于序列标记。从第二行开始为序列本身,只允许使用既定的核苷酸或氨基酸编码符号(参见支持代码类型)。通常核苷酸符号大小写均可,而氨基酸常用大写字母。使用时应注意有些程序对大小写有明确要求。一般每行60~80个字母。
核苷酸序列
fasta格式首先以大于号“>”开头,接着是序列的标识符“gi|187608668|ref|NM_001043364.2|”,然后是序列的描述信息。换行后是序列信息,序列中允许空格,换行,空行,直到下一个大于号,表示该序列的结束。
所有来源于NCBI的序列都有一个gi号“gi|gi_identifier”,gi号类似与数据库中的流水号,由数字组成,一条核酸或者蛋白质改变了,将赋予一个新的gi号(这时序列的接收号可能不变),不能重复。
gi号后面是序列的标识符,下表是来源于不同数据库的标识符的说明。标识符由序列来源标识、序列标识(如接收号、名称等)等几部分组成,他们之间用“|”隔开,如果某项缺失,可以留空但是“|”不能省略。如上例中标识符为“ref|NM_001043364.2|”,表示序列来源于NCBI的参考序列库,接收号为“NM_001043364.2”。
文件中和每一行通常60到80个字符。
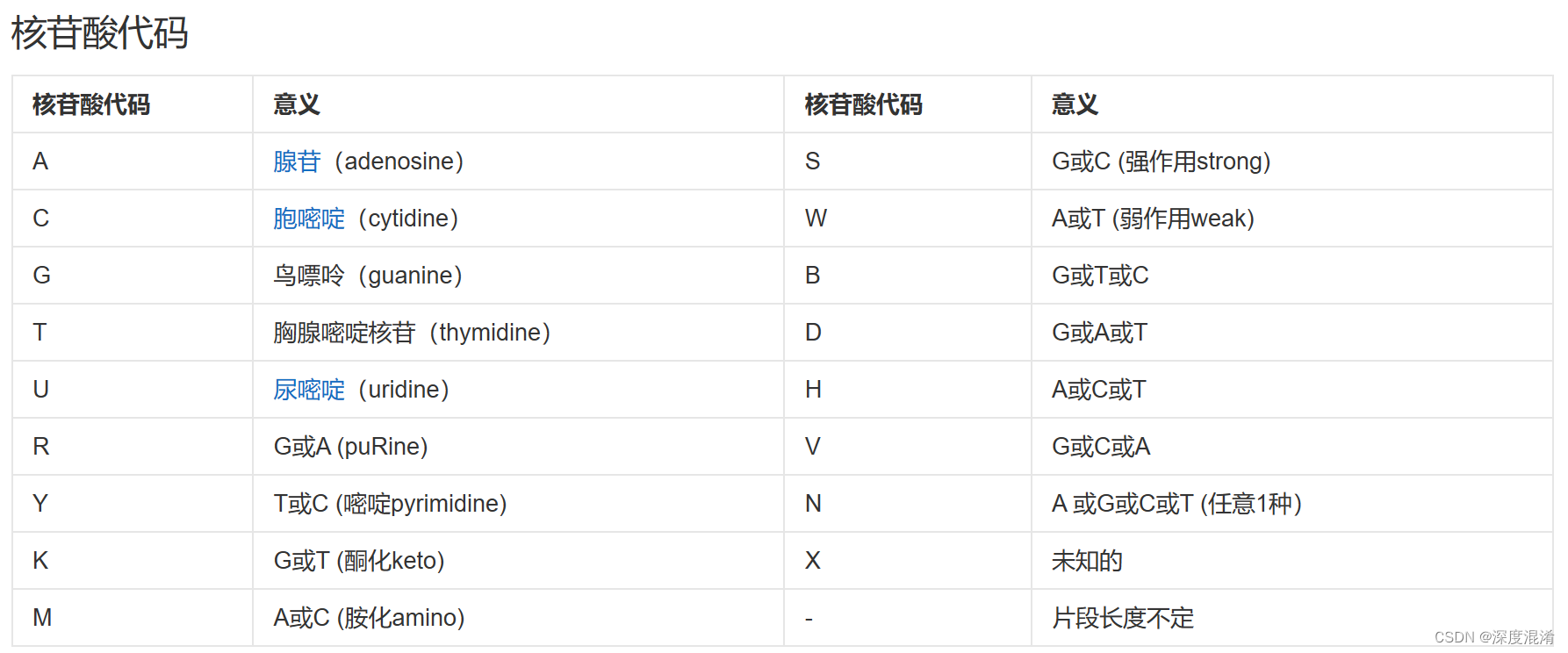
对于核酸序列,除了为大家所熟知的A、C、G、T、U外,R代表G或A(嘌呤);Y代表T或C(嘧啶);K代表G或T(带酮基);M代表A或C(带氨基);S代表G 或C(强);W代表A或T(弱);B代表G、T或C;D代表G、A或T;H代表A、C或T;V代表G、C或A;N代表A、G、C、T中任意一种。
氨基酸序列
>MCHU - Calmodulin - Human, rabbit, bovine, rat, and chicken ADQLTEEQIAEFKEAFSLFDKDGDGTITTKELGTVMRSLGQNPTEAELQDMINEVDADGNGTID FPEFLTMMARKMKDTDSEEEIREAFRVFDKDGNGYISAAELRHVMTNLGEKLTDEEVDEMIREA DIDGDGQVNYEEFVQMMTAK*
2.2 核苷酸代码
2.3 氨基酸代码

2.4 FASTA 实例
>NC_000006.12:c31170682-31164337 Homo sapiens chromosome 6, GRCh38.p14 Primary Assembly
GAGTAGTCCCTTCGCAAGCCCTCATTTCACCAGGCCCCCGGCTTGGGGCGCCTTCCTTCCCCATGGCGGG
ACACCTGGCTTCGGATTTCGCCTTCTCGCCCCCTCCAGGTGGTGGAGGTGATGGGCCAGGGGGGCCGGAG
CCGGGCTGGGTTGATCCTCGGACCTGGCTAAGCTTCCAAGGCCCTCCTGGAGGGCCAGGAATCGGGCCGG
GGGTTGGGCCAGGCTCTGAGGTGTGGGGGATTCCCCCATGCCCCCCGCCGTATGAGTTCTGTGGGGGGAT
GGCGTACTGTGGGCCCCAGGTTGGAGTGGGGCTAGTGCCCCAAGGCGGCTTGGAGACCTCTCAGCCTGAG
GGCGAAGCAGGAGTCGGGGTGGAGAGCAACTCCGATGGGGCCTCCCCGGAGCCCTGCACCGTCACCCCTG
GTGCCGTGAAGCTGGAGAAGGAGAAGCTGGAGCAAAACCCGGAGGAGGCAAGTGAGCTTCGACGGGGTTG
GGGTGTGGGGAGGTGGTCATGACAGGGCAGCCTGATGGGGAAGTGGTCACCTGCAGCTGCCCAGACCTGG
CACCCAGGAGAGGAGCAGGCAGGGTCAGCTGCCCTGGCCAGGGAGGGGTGTGTATCAACTGCTGGCAGCC
CTGGCAGGCAGGGGCCAGGTGGGAAGTGGAAGCTGGATTTCGAAGAGACAACTGCCGGTGAGGGCAGAGC
AGCCTGGGAGAGTCGGAAGCTGGCCCAGGCTGGCCTTTGCTCTGGCCCAGCCCTTGTCAGGGTCTCTCAC
ATCTCCTAGGCCTGCCCAGGGTCTGGTCACTCATTACTGGCCCAGCACCAGACCCAGCTTGGGGTTGGTT
TGAGCCCCTTTTCCCACCCTTAGTCCTGCTTGAAAATTTGACCCTTATCAGACCCAAGATTTTGGCCTTA
GGGTTAAGCATAGCCTGAGGGTAAAAACAGTGCTCATTCCAGGATTATTGTTCCTGAAAGTCTAGGGTGT
GACTCGTTTCTGATAGGATCTCCTGTTTGGGCTGTGTGTGTGCGCGTTGTGAGCTGGGTTTACCTCCAGT
CAAGTATAGGGCTTGTCTTCCCCGGATCTCTGCCTCAGGCCAATGACTGGCCACTGTGTTAAGGTGCACA
CCCTGGCACCCCTTGTAGAAAGCTGGATTTTGATTGACTTCAGCCTCAGTTCCAAAGTTGTAAACAAGAA
AAATGGTGAGAGATTTCTCCAGGCCATTTGCAAATATAGAGCTGCTGCGGGATTGAAGGCATCCAGCCCT
GCTGAGGACTATTAAAGATGTATCTTCCAGTCCTTCAAGGCGACAAGTGTAAGCAATTAGAGATTAAATA
CTAAGCCTTGAGACCTCACAGAAAGGTGTGACTGGTTTCTGGAGTGACCGAGAAGCCCCAACCTCTTCGC
AGGAGGTCACTGCTGAGCCTTGAATGATAATGGCTGGCAATTGTGGTCCACTTCCTAAGTGCCTGGCTGT
GTGCTCCGTTTATACATCATTATCTCATTAACCAGCACAAAATCTCCTAGGGGGAGGTATTATTATCCTA
TTTAACGGGTTTTAACTGCTAAATGATGAAGCGAGGATTTGGACCAGTGTTTATTCCAAAACCCCAAAAC
AGAATTTGGAAAATCCAAGATAGCAGAGGGCATTTATCAGTTTGAGTTATTGGCTGAGCAGAAGTTGGGG
ATGAAAACAGCCTATTTGAAATTGATATGATCAAGCACCATTGAAACACTTCCTTGAGGCTTCAGAACTA
CAAAAAGGCCTTGTTTTTTTCTCACTAGCTGTGCACCTCTGTCTGCCGGCAGCCTCATATGGCATGCCCC
AGGGCTCAGTCCTTCAACCTCTGCTCTATCTACCCTTCCTTCCTCTCACCCACCCTCAAGGCTTAAATGC
CATTTAGACACCAGATGACTACCGCGTTTTCTGTCTCTTGTGATGGCTCCCTGAACTGCTCCACCCTGAT
CACCCAGTTGCTCAAGGCCAAACCCAGTCATCCTCAGTTTCTTTCATGTCCTACATCCTATCCTTAAGAA
ACATCCTGAATCAATCACAACCTAACCCTGGCCTCAGCCACCATCATCTCTGCTGGGATTACCGCAGTAG
CTTCTCAAATTATACTGCTTCCTCCCTACTGTCTGTGGCCAACACGTCAACTAGAGTCAGTGTTTTAAAA
GGTGTGGCCAGGCACTTTGGGAGGCCGAGGCAGGCGAATCACCTGAGGTCGGGAGTTCGAGTCCAGCCTG
ACCAACATGGCGAAACCCCATCTCTACTAAAAATACAAAATTAGCTGGGCGTGGTGACGCATGCCTGTAA
TCTCAGCTACTCAGGAAGCTGAGGCAGGAGAATCGCTTGAACCTGGGAGATGGAGGTTGCGGTGAGCCGA
GATCGCGCCAGTGCACTCCAGCCTAGGCAACAAAAGCGAAACTCTCAAAAAAAAAAAAAAAAAAGGTGAG
GCTAGGTGCGGTGGTTCACACCTGTAATCCCAGCACTTTGGGAGGCCAAGGTGGACAGATCACTTGAGGT
CTCCTGACCAGCCTGGCCAACATGGTGAAACCCCATATCTACTAAAAATACAAAAATTAGCCGGGCATGG
TGGTGGGTGCCTGTAGTCCCAGCTACTCAGGCGGCTGAGGCAGAATAGCTTGAACCCAGAAGGCGGAGAT
TGCAGTGAGCCAAGATCACGCCACAGCACTCCAGCCTGGGCGATAGAACGAGATTCCGTCTTGGTGGGGA
GAAAAAGGGTGAGAGATCATTTCGCTTGGACTAAAACAAAGTCACTATGTCTGCAACAGGATCTACCTAG
CCACCAGACCAGCTTTGGGCTCTGGAAGGCCCACTTCAGGGCCTTGCCACATTAGACTCTTGTCCTTTGC
TCAAACAATCACCTTCTCTGTCTTTAAAAGTGTCACCCTCCTCCATAATCTCCTTCCCTCCTTTACCCTA
CTCCTATAGACTGCTTTATTTTTTTTTTAATTTTTGAGATGGAGTCTCACTCTGTCCCTCAGGCTGGAGT
GCAGTGGTGCGATCTTGGCTCACTGCAAACTCCACCTCCTAGGTTCAAGCAATTCTCCTGCCTCAGCCTC
CTGAGTAGCTGGGATTATAGGGGAGCGCCATGATGCCCAGCTAATTTTTGTATTTTTAGTAGAGACAGAG
TCTCACTATGTTGACCAGGCTAGTCTTGAACTCCTGACCTCAAGTGATCTACCCACCTTGGCCTCCCAAA
GTGAAGGGATTACAGGCATGACCACTGCGCCCAGACTGCTTTACTTTTTTCCATAATATATATATATATT
TTAAATAGAGGCAGCAGGGGTGGGAGAAGGGGCGGCACGGGTCTCACTATGTTACCCAGGCTGCTTTCTA
ACTCTTGGGCTCAAGCAGTCTGCCCACCTTGGCCTCCCAAAGTGCTAGGATTTACAGACATGAGCCACTG
TGCCTGGCCATTTTTTATTTTATTTACTTTTTTATTTTTCAGAGCAGGAGTGGAAGTTTATTATTAAAAA
GTTATAGGGCAGGGAAAAAAGGAAAGTGCACTTGGAAGAGATCCAAGTGGGCAACTTGAAGAACAAGTGC
CAAATAGCACTTCTGTCATGCTGGATGTCAGGGCTCTTTGTCCACTTTGTATAGCCGCTGGCTTATAGAA
GGTGCTCGATAAATCTCTTGAATTTAAAAATCAATTAGGATGCCTCTATAGTGAAAAAGATACAGTAAAG
ATGAGGGATAATCAATTTAAAAAATGAGTAAGTACACACAAAGCACTTTATCCATTCTTATGACACCTGT
TACTTTTTTGCTGTGTTTGTGTGTATGCATGCCATGTTATAGTTTGTGGGACCCTCAAAGCAAGCTGGGG
AGAGTATATACTGAATTTAGCTTCTGAGACATGATGCTCTTCCTTTTTAATTAACCCAGAACTTAGCAGC
TTATCTATTTCTCTAATCTCAAAACATCCTTAAACTGGGGGTGATACTTGAGTGAGAGAATTTTGCAGGT
ATTAAATGAACTATCTTCTTTTTTTTTTTTCTTTGAGACAGAGTCTTGCTCTGTCACCCAGGCTGGAGTG
CAGTGGCGTGATCTCAGCTCACTGCAACCTCCGCCTCCCGGGTTCAAGTGATTCTCCTGCCTCAGCCTCC
TGAGTAGCTGGGATTACAGGTGCGTGCCACCGTGCCCAGCTAATTTTTGTGTTTTTAGTAGAGACGGGGT
TTCACCATGTTGGCCATGCTGGTCTTGAACTCCTGACCTCGTGATCTGCCCACCTCGGCCTCCCAAAGTG
CTGGAATTATAGGCGTGAGCCACCGCGCCCAGCAAAGAACTTCTAACCTTCATAACCTGACAGGTGTTCT
CGAGGCCAGGGTCTCTCTTTCTGTCCTTTCACGATGCTCTGCATCCCTTGGATGTGCCAGTTTCTGGGGG
AAGAGTAGTCCTTTGTTACATGCATGAGTCAGTGAACAGGGAATGGGTGAATGACATTTGTGGGTAGGTT
ATTTCTAGAAGTTAGGTGGGCAGCTTGGAAGGCAGAGGCACTTCTACAGACTATTCCTTGGGGCCACACG
TAGGTTCTTGAATCCCGAATGGAAAGGGGAGATTGATAACTGGTGTGTTTATGTTCTTACAAGTCTTCTG
CCTTTTAAAATCCAGTCCCAGGACATCAAAGCTCTGCAGAAAGAACTCGAGCAATTTGCCAAGCTCCTGA
AGCAGAAGAGGATCACCCTGGGATATACACAGGCCGATGTGGGGCTCACCCTGGGGGTTCTATTTGGTGG
GTTCCCCTCTGCAGATTCTGACCGCATCTCCCCTCTAAGGAGTATCCCTGAACCTAGTGGGGAGGGGCAG
GGGCAGACTACCCTCACCCATGAAGAGGAGTAGGGAGAGGGAGAAGATGCTTTGAGCTCCCTCTGGGAAG
AGGTGGTAAGCTTGGATCTCAGGGTCACAAGGGCCCTGCGTGCTCCCTCACTTTGCTTCTCTTTTGACTG
GCCTCCCCCAGGGAAGGTATTCAGCCAAACGACCATCTGCCGCTTTGAGGCTCTGCAGCTTAGCTTCAAG
AACATGTGTAAGCTGCGGCCCTTGCTGCAGAAGTGGGTGGAGGAAGCTGACAACAATGAAAATCTTCAGG
AGGTAAGGGTGGGAGGGGGATACCCGGGGACCTTCCCTTTCTTGGCCTAATTTCCATTGCTTCCATCACT
GGCTCGTAGCTCTCCGTCTTTGGTGCAGTGGTTCTCAGTGGGATGGAGTGAAATTCCTCAGTTCTGCTGG
GATAAGGTCCAGAGCCAACCCTTCCAGGATCCTGCCTTTTCACACCACCACCTGGCTCTGCTGACACATC
TAGTCACAGACCCCTGTGATGCTGTTACTCAGCAAGTCCAAAGCTTGCCCTTGTCACCCCCTTCCCACCT
GCACAGATATGCAAAGCAGAAACCCTCGTGCAGGCCCGAAAGAGAAAGCGAACCAGTATCGAGAACCGAG
TGAGAGGCAACCTGGAGAATTTGTTCCTGCAGTGCCCGAAACCCACACTGCAGCAGATCAGCCACATCGC
CCAGCAGCTTGGGCTCGAGAAGGATGTGAGTGCCATGTCTCTCTGCGGGCTCCATCTCTTTCCCCTGTCA
CCACCTCGCTTTCCCTAGCTCTGGCTCCTCCAACTGCTCTAGGGCTGTTGGCTTTGGACAGAATGTCCAA
GCAGTCAGGCCTGTCTCAGCTCATTCTCTAATGTCCTCCTCTAACTGCTCTAGGGCTGTTGGCTTTGGAT
AGAATGTCCAAGCAGAGTCAGGCCCGTCTCAGCTCATTGTCTAATGTCATTCTCCTTTCTGTCATTCACT
TGCAGGTGGTCCGAGTGTGGTTCTGTAACCGGCGCCAGAAGGGCAAGCGATCAAGCAGCGACTATGCACA
ACGAGAGGATTTTGAGGCTGCTGGGTCTCCTTTCTCAGGGGGACCAGTGTCCTTTCCTCTGGCCCCAGGG
CCCCATTTTGGTACCCCAGGCTATGGGAGCCCTCACTTCACTGCACTGTACTCCTCGGTCCCTTTCCCTG
AGGGGGAAGCCTTTCCCCCTGTCTCCGTCACCACTCTGGGCTCTCCCATGCATTCAAACTGAGGTGCCTG
CCCTTCTAGGAATGGGGGACAGGGGGAGGGGAGGAGCTAGGGAAAGAAAACCTGGAGTTTGTGCCAGGGT
TTTTGGGATTAAGTTCTTCATTCACTAAGGAAGGAATTGGGAACACAAAGGGTGGGGGCAGGGGAGTTTG
GGGCAACTGGTTGGAGGGAAGGTGAAGTTCAATGATGCTCTTGATTTTAATCCCACATCATGTATCACTT
TTTTCTTAAATAAAGAAGCCTGGGACACAGTAGATAGACACACTTA3 EMBL 转 FASTA
生信学员常常用到的功能。
3.1 基本需求
(1)可以一次性翻译大量(比如1,000,000个)的文件;
(2)可以翻译很大(比如含有1,000,000条序列)的文件;
(3)可以看到翻译中的进度;
(4)速度要尽量快一点;
(5)可靠性要高一点;
3.2 核心代码
using System;
using System.IO;
using System.Text;
using System.Collections;
using System.Collections.Generic;
using System.Runtime.Serialization;
namespace Legal.BIOG
{
public static class EMBL
{
/// <summary>
/// EMBL格式转为FASTA格式
/// </summary>
/// <param name="embl"></param>
/// <returns></returns>
public static string EMBL_To_FASTA(string embl)
{
string[] xlines = embl.Split(new char[] { '\r', '\n' }, StringSplitOptions.RemoveEmptyEntries);
StringBuilder sb = new StringBuilder();
for (int i = 0; i < xlines.Length; i++)
{
if (xlines[i].StartsWith("DE ") && sb.Length == 0)
{
sb.AppendLine(">" + xlines[i].Substring(2).Trim());
}
else if (xlines[i].StartsWith("SQ "))
{
int j = i + 1;
while (j < xlines.Length)
{
if (xlines[j].StartsWith("//")) break;
string[] xa = xlines[j].Split(new char[] { ' ' }, StringSplitOptions.RemoveEmptyEntries);
xa[xa.Length - 1] = "";
sb.AppendLine(String.Join("", xa).ToUpper());
j++;
}
break;
}
}
return sb.ToString();
}
}
}
3.3 运行效果
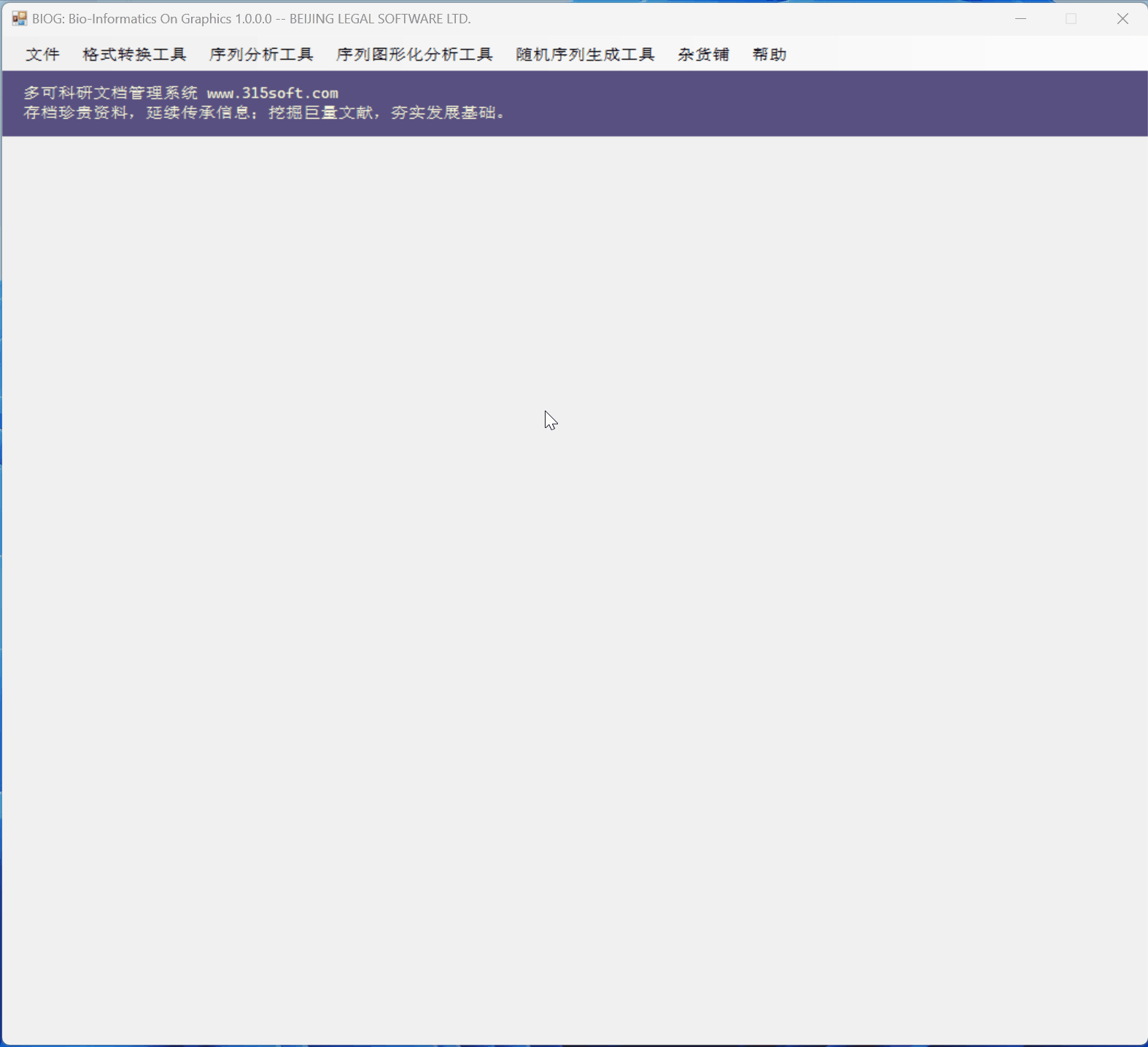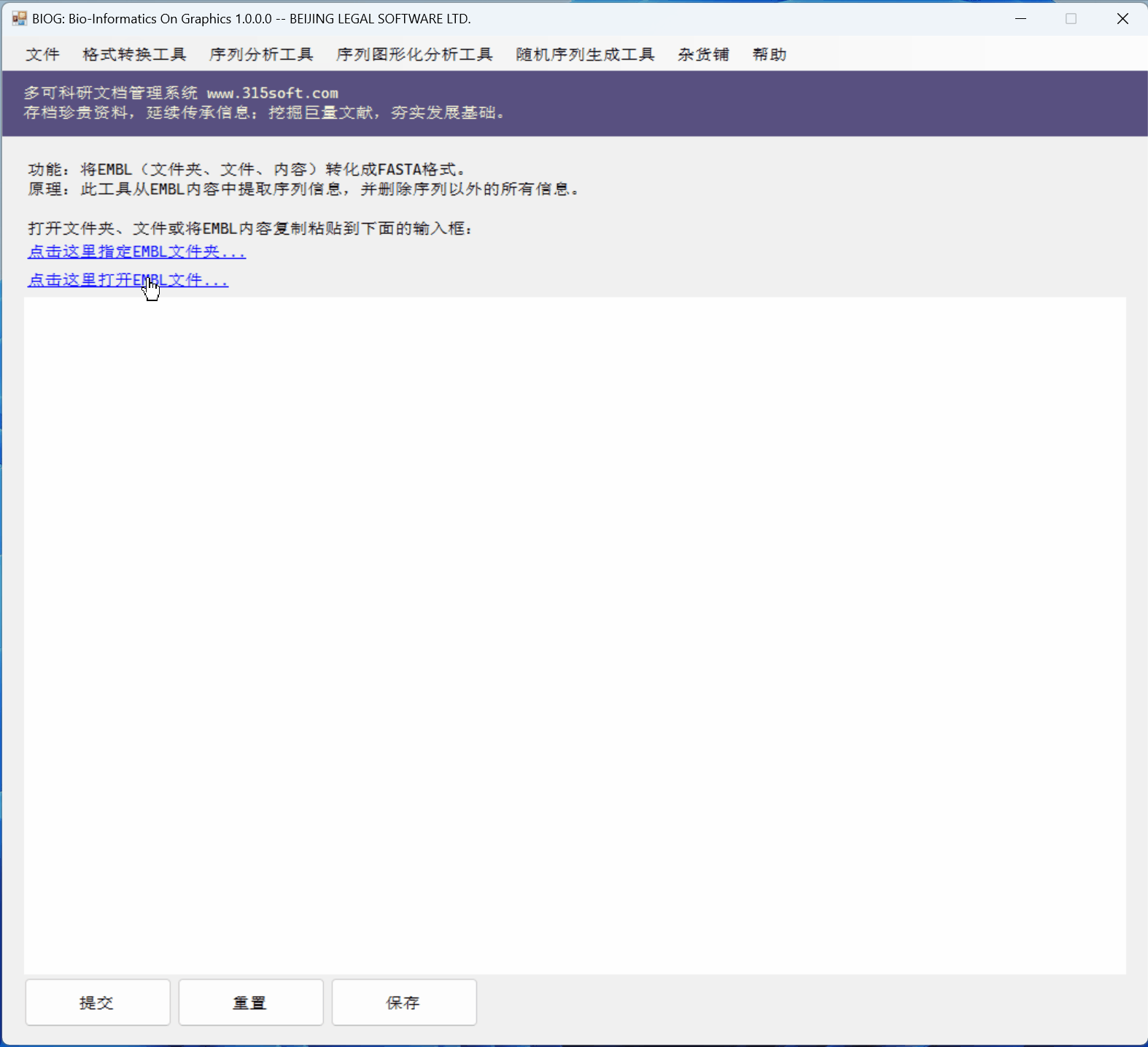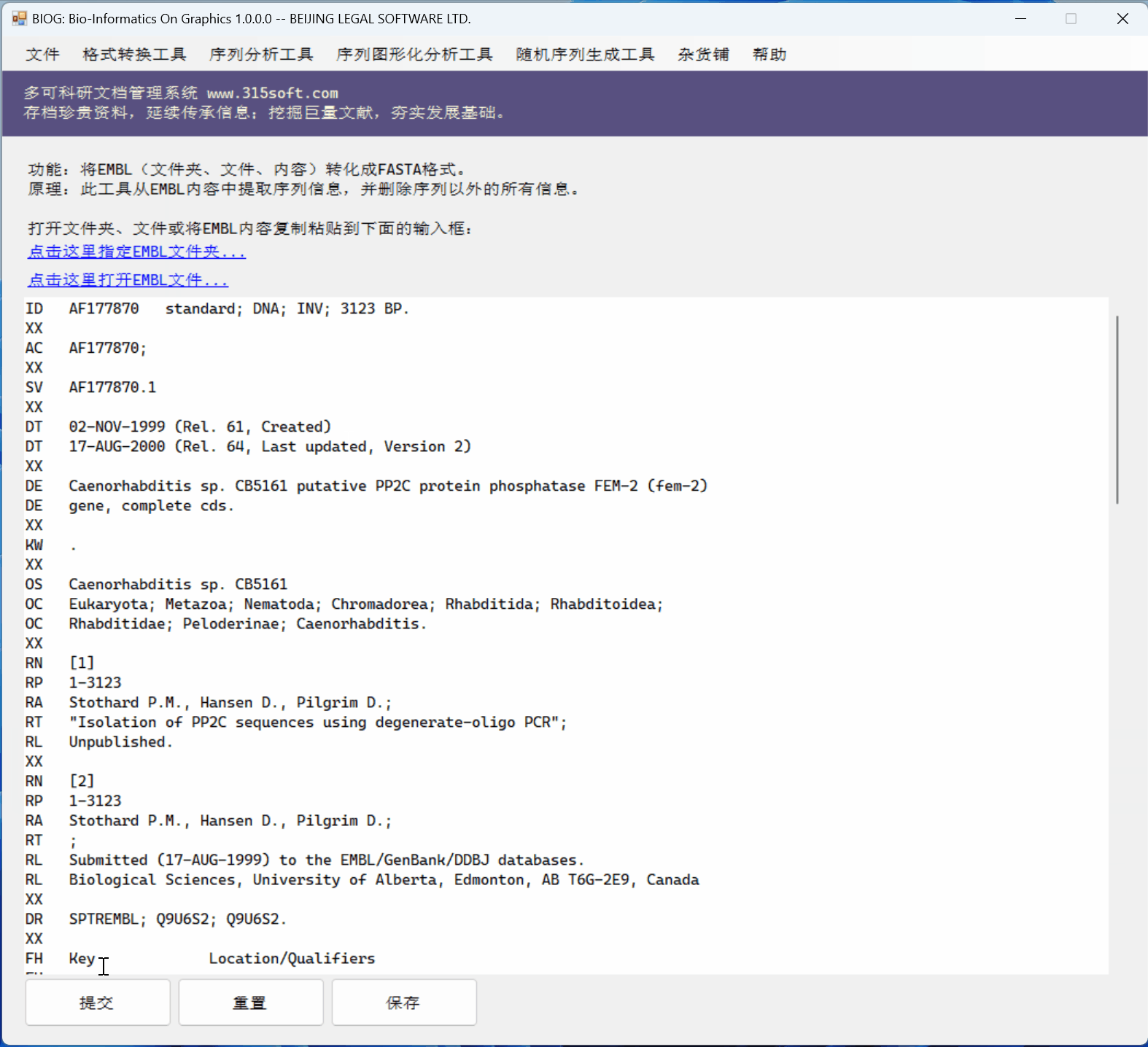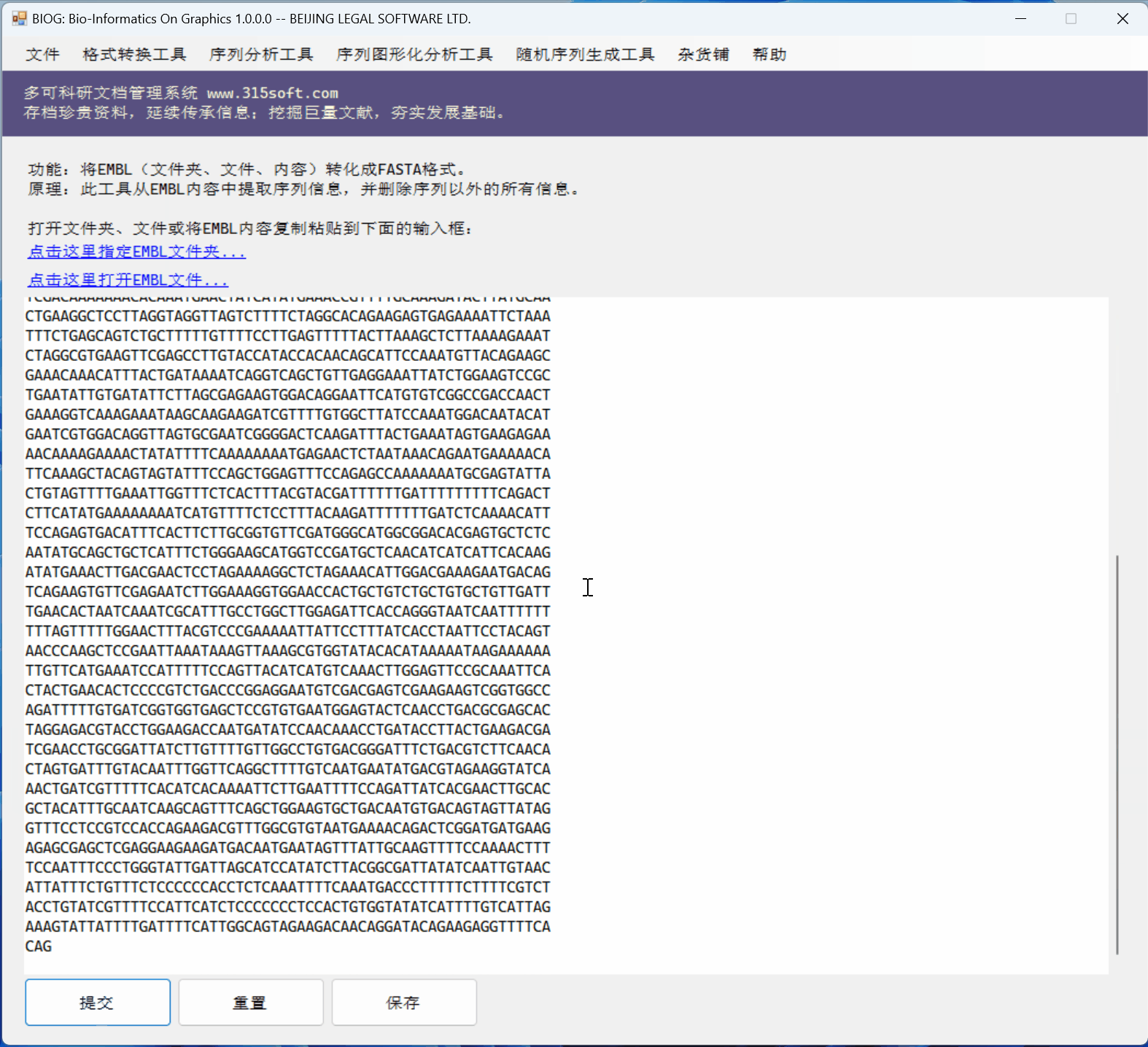
桌面软件 BIOG 的几个优点:
(1)桌面软件能一次性处理很多很多的文件,比如一个文件夹及其子目录下的所有文件;百万个也可以的;
(2)桌面软件可以处理很大的文件;比如超过2GB的文件,在线版本是无法处理的!
(3)桌面软件的速度当然比较快!












![[OtterCTF 2018]之Misc篇(NSSCTF)刷题记录⑦](https://img-blog.csdnimg.cn/54900bb54c1b45808fa02b4ca5a16e54.png)