利用HMM纠正测序错误(Viterbi算法的python实现)
问题背景
对两个纯系个体M和Z的二倍体后代进行约~0.05x的低覆盖度测序,以期获得后代个体的基因型,即后代中哪些片段分别来源于M和Z。已知:
-
后代中基因型为MM、MZ(杂合)和ZZ的比例是0.4:0.2:0.4;(初始概率)
-
直接通过测序结果判断单个位点的基因型的错误率为3%;(输出概率)
-
测序获得的M和Z之间的多态性位点相互之间距离恰好一致,重组率均为0.01。(转移概率)
有一个后代的一段染色体,测序获得的109个位点的基因型依次为:
1111111011111101111111000000001000000001000001000000000000001011001010101010111111101111111111101111111011111
其中1表示MM,0表示ZZ。由于低覆盖度测序,杂合位点只能测到其中一个等位基因,因此表现为MM或ZZ。
请构建隐马科夫模型,并推断这段序列各位点真正的基因型。
参考结果:
1111111111111111111111000000000000000000000000000000000000002222222222222222111111111111111111111111111111111
其中1表示MM,0表示ZZ,2表示MZ(杂合)。
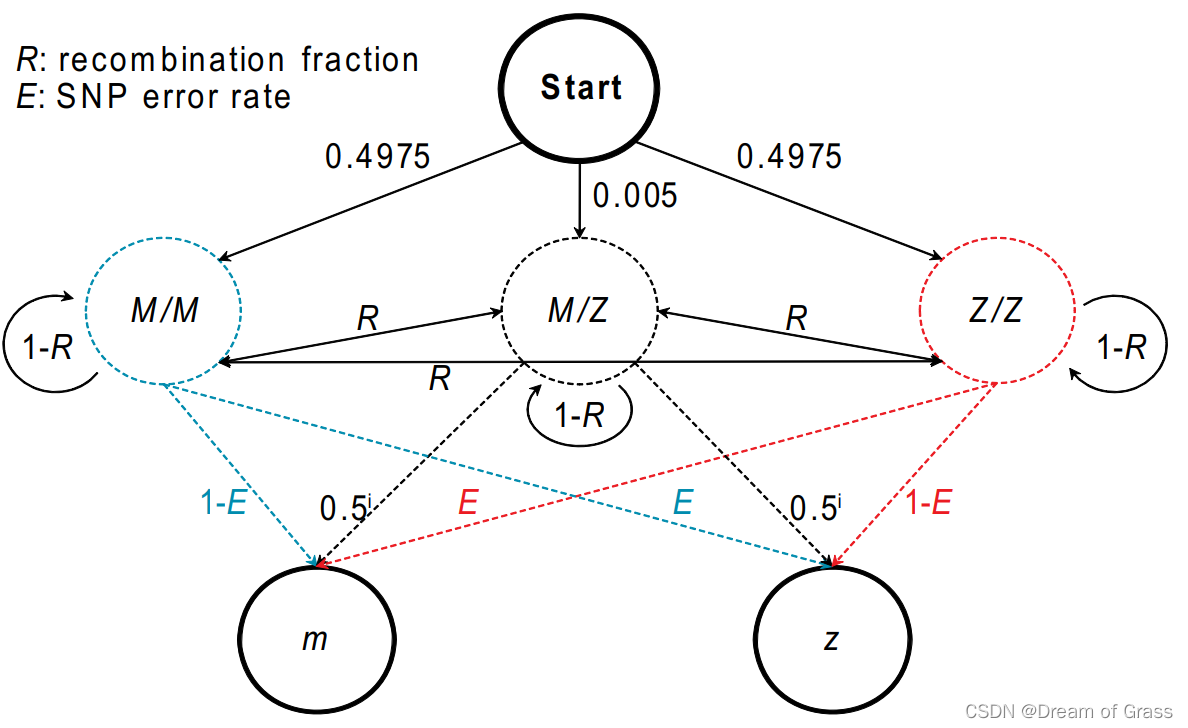
五大参数说明
状态集
| MM | MZ | ZZ |
|---|---|---|
| 1 | 2 | 0 |
观测集
| m | z |
|---|---|
| 1 | 0 |
初始分布
| MM | MZ | ZZ |
|---|---|---|
| 0.4 | 0.2 | 0.4 |
状态转移概率矩阵
重组率表示重组型配子占总配子数的比例,MM→MZ和MM→ZZ加起来应为重组率。
二者在重组类型中的比例则是通过初始分布得到的,即MM:MZ:ZZ=2:1:2。
故相较于论文中转移概率矩阵,调整后如下:
| MM | MZ | ZZ | |
|---|---|---|---|
| MM | 1 − R 1-R 1−R | R 3 \frac{R}{3} 3R | 2 R 3 \frac{2R}{3} 32R |
| MZ | R 2 \frac{R}{2} 2R | 1 − R 1-R 1−R | R 2 \frac{R}{2} 2R |
| ZZ | 2 R 3 \frac{2R}{3} 32R | R 3 \frac{R}{3} 3R | 1 − R 1-R 1−R |
https://doi.org/10.1073/pnas.1005931107
输出概率矩阵
| z | m | |
|---|---|---|
| MM | E E E | 1 − E 1-E 1−E |
| MZ | 1 2 \frac{1}{2} 21 | 1 2 \frac{1}{2} 21 |
| ZZ | 1 − E 1-E 1−E | E E E |
代码实现
参数设置
import numpy as np
# 转移概率矩阵
A = [[0.99, 0.02 / 3, 0.01 / 3],
[0.01 / 2, 0.99, 0.01 / 2],
[0.02 / 3, 0.01 / 3, 0.99]]
# 行列为 MM MZ ZZ
# 输出概率矩阵
B = [[0.03, 0.97],
[0.5, 0.5],
[0.97, 0.03]]
# 行为 MM MZ ZZ, 列为 z m
# 初始状态分布
initp = [0.4, 0.2, 0.4]
# 对应着 MM MZ ZZ
# 状态数和状态名
S = 3
S_name = ['1', '2', '0']
# 观察序列
Y = '1111111011111101111111000000001000000001000001000000000000001011001010101010111111101111111111101111111011111'
viterbi实现
# viterbi实现
# 向前计算
Probability = []
Position = []
Position.append([0, 0, 0])
first = []
for i in range(len(initp)):
first.append(initp[i] * B[i][int(Y[i])])
Probability.append(first)
for i in range(1, len(Y)):
ProbabilityTemp = [] # 用来存放每一层观察值对应的三种状态概率值
GetPosition = []
for j in range(S):
ProbabilityTemp.append((np.max(np.array(Probability[i - 1]) * np.array(A[j]))) * B[j][int(Y[i])])
GetPosition.append(np.argmax(np.array(Probability[i - 1]) * np.array(A[j])))
Probability.append(ProbabilityTemp)
Position.append(GetPosition)
traceback_idx = np.argmax(np.array(Probability[-1]))
path = [S_name[traceback_idx]]
for i in range(len(Y) - 2, -1, -1):
path.append(S_name[traceback_idx])
traceback_idx = Position[i][traceback_idx]
print('程序输出如下:')
print((''.join(path))[::-1])
print('参考答案如下:')
print('1111111111111111111111000000000000000000000000000000000000002222222222222222111111111111111111111111111111111')
程序输出如下:
1111111111111111111111000000000000000000000000000000000000002222222222222222111111111111111111111111111111111
参考答案如下:
1111111111111111111111000000000000000000000000000000000000002222222222222222111111111111111111111111111111111










![[激光原理与应用-60]:激光器 - 光学 - 光的四大理论框架与其层次:几何光学、波动光学、电磁光学、电子光学](https://img-blog.csdnimg.cn/255216ad005943f9a16ba679b7bb17d8.png)