
–https://arxiv.org/abs/2405.06708
代码开源:https://github.com/PharMolix/OpenBioMed
LangCell: Language-Cell Pre-training for Cell Identity Understanding
留意更多内容,欢迎关注微信公众号:组学之心
研究团队和研究单位
聂再清–清华大学

研究简介
当前单细胞表示模型面临的挑战
目前,用于表示单细胞数据的模型已经取得了显著进展。然而,这些模型在理解细胞身份这一关键生物学问题上仍存在一些挑战:
-
1.对人类专家知识的利用不足: 现有的模型主要通过自监督学习来捕捉基因之间的共表达关系,但未能充分利用人类专家对细胞类型的深入理解。这限制了模型在理解细胞身份方面的能力,从而影响其在下游任务中的表现。
-
2.缺乏细胞与文本/标签的配对数据: 细胞身份信息通常由人类专家通过自然语言描述。然而,现有的模型难以直接将这些文本信息与单细胞数据关联起来,这阻碍了模型对细胞身份的深入理解。
-
3.数据标注成本高昂: 在实际应用中,获取大量高质量的标注数据成本很高,特别是在研究新的疾病或细胞亚型时。
LangCell模型
为了解决上述问题,研究人员提出了LangCell模型。该模型通过将scRNA-seq数据与多角度的文本注释对齐,能够使得模型对细胞身份的有更深入的理解。
-
1.整合多模态数据: LangCell将scRNA-seq数据的特征空间与文本信息进行结合,使得模型能够同时学习基因表达模式和文本描述中的语义信息。
-
2.提升模型泛化能力: 通过将模型暴露于大量多样化的文本数据,LangCell能够从已知的细胞类型扩展到新的细胞类型,提高模型的泛化能力。
-
3.增强预测精度: 模型通过语义一致性引导,能够更准确地预测细胞类型,并提高其在多种生物医学场景中的适用性。
1.LangCell模型
1.1 数据处理
scRNA-seq数据
模型使用Raw count矩阵 A ∈ N m × n A ∈ N^{m×n} A∈Nm×n. m是细胞,n是基因。
质控中过滤了重复、表达基因少于 200 个、meta数据缺失过多或用于下游任务的数据。
-
1.去除测序深度的影响:分别对每个细胞的基因表达进行归一化,得到A′。
-
2.消除整体表达水平带来的差异:找到每列 (基因)𝐴′的非零中位数 Font metrics not found for font: . 作为每个基因的中位表达值,并使用 𝛽对 𝐴′的每列进行归一化,得到 𝐴′′。

例如,一些管家基因可能有更高的绝对表达量,但这并不意味着该基因在该细胞中有特别显著的高表达。
基于𝐴′′,根据每个细胞中基因的相对表达进行排序,获得该细胞的序列表示(基因表达值排序和的对应顺序位置,用于Geneformer模型构建embedding,将基因表达矩阵转换为类似于自然语言的序列数据)。
文本注释知识库
研究从CELLxGENE中获取了原始的scRNA-seq数据和相应的meta数据,构建了一个细胞-文本数据集–scLibrary,包含大约 2750 万对 scRNA-seq 数据和相关文本描述的综合数据集。
研究选择了八个可能包含重要见解的细胞身份关键方面,包括细胞类型、发育阶段和疾病信息,并从开放生物学和生物医学本体论库(OBO Foundry)中尽可能获得详细的描述。
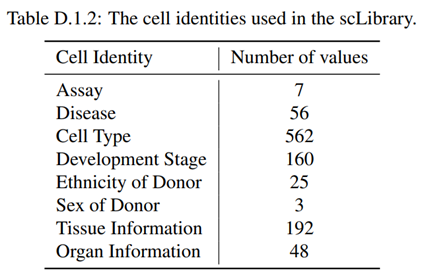
1.2 模型架构

模型主要分为上半部分(Cell encoder)和下半部分(Text encoder)。
Cell encoder
使用预训练的 Geneformer来初始化细胞encoder,它将顺序的细胞输入编码为embedding序列。并在每个序列的开头添加了一个 𝐶𝐿𝑆 token。每个细胞把自己高表达的基因排好序,每个细胞允许输入最大长度为2048个基因(有一个全部基因的字典)。
Text encoder
该encoder使用 PubMedBERT预训练权重进行初始化,有两种encoder模式:单模态 g 1 g_1 g1和多模态 g 2 g_2 g2。
对于单模态文本encoder,它相当于 BERT。
对于多模态encoder,研究在每个自注意力模块之后添加了一个可切换的跨注意力模块,用于计算联合嵌入和通过线性层计算细胞-文本匹配概率。

定义细胞encoder为 f f f,它用于从单细胞数据 c c c 中获取嵌入 z c z_c zc。
定义文本encoder的单模态模式为 g 1 g_1 g1,它负责从文本数据 t t t 中生成嵌入 z t z_t zt。
定义文本encoder的多模态模式为 g 2 g_2 g2,它用于计算细胞数据和文本数据之间的匹配概率 p c , t p_{c,t} pc,t,这三种编码方式的公式如下:
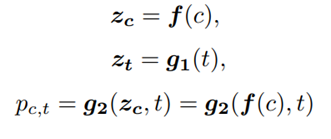
1.3 预训练过程
模型旨在将 scRNA-seq 数据和文本映射到一个共享的潜在空间,并利用自然语言中包含的非结构化知识作为远程监督,优化细胞表示学习。
所以在预训练过程中联合优化了四个目标损失函数,包括掩码基因建模(MGM)、模态内和模态间对比学习(C-C, C-T),以及细胞-文本匹配(CTM)。
掩码基因建模 (MGM):
随机masked细胞输入序列中的部分基因,并使用模型在这些位置的输出嵌入来预测原始输入的重建。使用交叉熵损失函数作为该多分类任务的损失函数,公式如下:
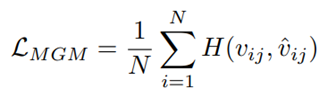
其中,𝑁 是掩码基因的数量, v i j v_{ij} vij 和 v ^ i j \hat{v}_{ij} v^ij 分别表示第 𝑖 个掩码位置的真实标签和预测概率,𝐻 是交叉熵损失函数。
细胞-细胞模态内对比学习 (C-C):
引入细胞-细胞对比学习来缓解 BERT 方法引起的表示退化问题。
在 scRNA-seq 数据中,每个基因表达水平都有独特的含义,而数据顺序和输入扰动等人工数据增强方法可能会破坏基因表达的语义。
研究认为特征级的扰动更适合用于 scRNA-seq 数据中的数据增强,因此把标准 dropout 的两个实例应用于相同的单细胞来构建正样本,而同一批次中的其他单细胞作为负样本,这在自然语言研究中已被证明是有效的。
为了在有限内存下扩大批次大小,研究采用了动量编码器方法。用InfoNCE 损失函数,公式如下:

其中,𝑇 是批次大小,sim 是余弦相似度函数,𝜏 是温度参数, 𝑧 𝑐 ( 𝑖 ) 𝑧_𝑐^{(𝑖)} zc(i)和 𝑧 𝑐 ( 𝑖 ) + 𝑧_𝑐^{(𝑖)+} zc(i)+分别表示第 𝑖 个细胞及其正样本的嵌入。
细胞-文本模态间对比学习 (C-T):
研究通过细胞-文本对比学习将细胞和文本投射到相同的嵌入空间中。文本encoder采用单模态编码模式,并使用动量encoder来扩大批次大小。其损失函数如下:
公式中的符号具有与前面相似的含义。 𝑧 t ( 𝑖 ) 𝑧_t^{(𝑖)} zt(i)表示文本的嵌入。

细胞-文本匹配 (CTM):
在计算此损失时,文本encoder采用多模态编码模式,经过每个自注意力层后进行细胞嵌入与文本的交叉注意力计算,最终的输出用于二分类,以预测细胞是否与文本匹配。
该任务旨在以更高的分辨率探索细胞与文本之间的匹配关系,选择尽可能与正样本细胞-文本对相似的细胞和文本来形成负样本。其损失函数为二元交叉熵:

其中,𝑦 表示细胞是否与文本匹配的标签。
预训练第一阶段
用 Geneformer 初始化细胞encoder参数,并仅使用 𝐿 𝑀 𝐺 𝑀 𝐿_{𝑀𝐺𝑀} LMGM和 𝐿 𝐶 − 𝐶 𝐿_{𝐶−𝐶} LC−C 损失函数进行单模态训练,这样可以获得更好的单细胞表示学习模型。训练三轮。
预训练第二阶段
用 PubMedBERT 初始化文本encoder参数,并使用所有四个损失函数进行多模态训练。研究优化四个损失的加权和,以同时探索 scRNA-seq 数据的内在模式及其与文本的关联性。训练三轮。其公式为:

其中,𝜃 表示模型参数,𝛾𝑖 是作为超参数的权重。
训练参数如下:

2.下游应用
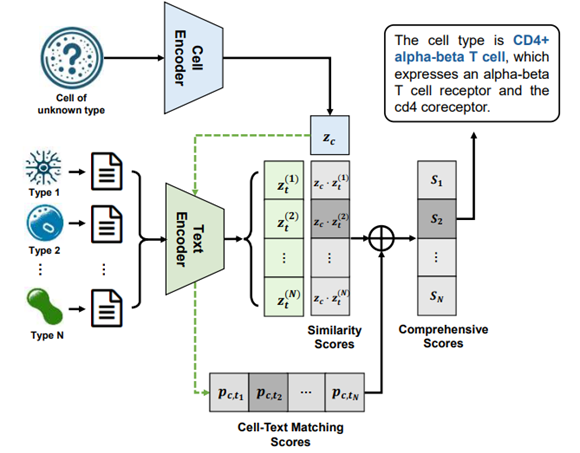
对于给定的单个细胞 𝑐 和 𝑁个候选文本描述 [ 𝑡 ( 𝑖 ) ] 𝑁 𝑖 = 1 [𝑡^{(𝑖)}]^{𝑖=1}_𝑁 [t(i)]Ni=1,研究通过比较它们在共享嵌入空间中的余弦距离获得 𝑙 𝑜 𝑔 𝑖 𝑡 𝑠 1 𝑙𝑜𝑔𝑖𝑡𝑠_1 logits1,并通过比较细胞-文本匹配模块给出的分数获得 𝑙 𝑜 𝑔 𝑖 𝑡 𝑠 2 𝑙𝑜𝑔𝑖𝑡𝑠_2 logits2 。
两者根据权重 𝛼 进行综合考虑用于分类。在实际应用中,由于 𝑙 𝑜 𝑔 𝑖 𝑡 𝑠 2 𝑙𝑜𝑔𝑖𝑡𝑠_2 logits2 计算较慢,研究可以仅对 𝑙 𝑜 𝑔 𝑖 𝑡 𝑠 1 𝑙𝑜𝑔𝑖𝑡𝑠_1 logits1分数较高的候选者计算 𝑙 𝑜 𝑔 𝑖 𝑡 𝑠 2 𝑙𝑜𝑔𝑖𝑡𝑠_2 logits2。符号 ⊕ 表示 Softmax 操作后的加权和。
2.1 零样本(zero-shot)细胞身份理解
2.1.1 新细胞类型鉴定
该任务要求模型在没有微调数据的情况下执行细胞类型注释,称为“新型细胞类型识别”。
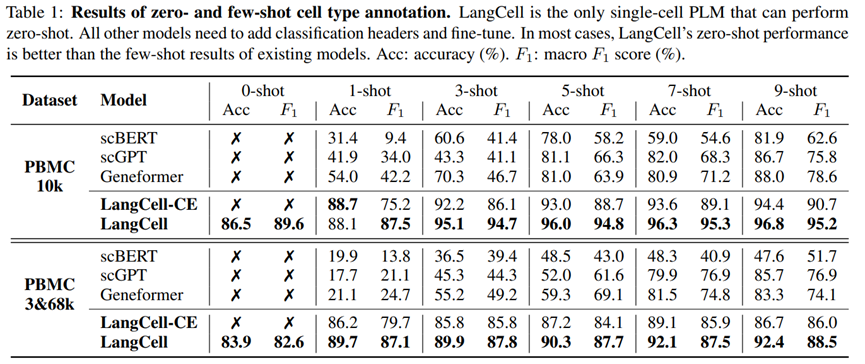
在零样本(zero-shot)学习之外,还参与了少样本(few-shot)学习,以针对基线进行基准测试,并评估 LangCell 的数据效率。研究仔细选择了具有实际意义的少样本设置,并在生物信息学中具有重要意义。本研究关注两个在实际应用中常见的场景:
-
- 零样本和少样本细胞类型注释:适用于具有较少备选细胞类型的场景,通常出现在对来自特定组织的小规模单细胞数据进行注释或将某一特定细胞类型分成多个亚型时。少样本方法配置为在微调期间使用每个类别的 n 个训练样本 (1≤n≤9)。该配置考虑了实际情况,即在现实设置中备选类型较少,使得为每个类别提供少量手动标注数据变得可行。在少样本任务中,所有基线模型都添加了一个线性层作为分类头。
实验结果表明,LangCell 在零样本和少样本设置的这两类任务中表现优异:

-
- 细胞-文本检索:适用于具有许多备选细胞类型的场景,通常见于复杂环境中的单细胞注释或完全未知的单细胞注释。鉴于细胞类型的丰富性,这些细胞类型可能包含边界模糊的亚型。少样本方法结构化为在有限选择的细胞类型上进行微调,然后在一系列未见的细胞类型上进行测试。
该方法与现有唯一具有跨类型迁移能力的模型 BioTranslator 进行比较。
研究测试了具有 161 种细胞类型的具有挑战性的综合人类细胞数据集 Tabula Sapiens,其中 66 种完全是 scLibrary 中未包括的新类型。使用常见的多模态检索任务评估指标 recall@k。模型效果如下:

2.1.2 NSCLC亚型分类
研究在非小细胞肺癌 (NSCLC) 2个癌症亚型肺鳞状细胞癌 (LUSC) 和肺腺癌 (LUAD)的一共2658 个恶性细胞上测试了 LangCell模型效果,来评估其在识别与疾病相关的细胞身份方面的有效性。
所有细胞类型标签均为“恶性”,使用这两种疾病的描述来构建文本。LangCell 将单细胞和疾病文本很好地对齐在潜在空间中:

其零样本分类比 Geneformer(使用 10-shot 学习微调)提高了大约 20%,无论是在准确性还是宏观 F1 分数上。这项实验展示了 LangCell 在理解与疾病相关的细胞身份以及在分析具有高突变负担的单细胞(如恶性细胞)方面的强大能力。

2.1.3 单细胞批次整合
研究在 PBMC10K 和大脑外嗅皮质 (Perirhinal Cortex) 上评估了 LangCell 在细胞批次整合中的性能,并将其与领域内的经典模型 scVI 以及多个单细胞 PLM 进行了比较。
这些指标分别评估了模型的生物整合能力、批次效应消除能力,以及两者的综合评价。实验结果表明,LangCell 的所有三个指标上都超过了现有的最优模型。这证明了 LangCell 对转录组数据的深刻洞察、准确保留重要生物学信息的能力,以及在纠正无关批次效应方面的有效性。

2.2 LangCell-CE 的细胞表示学习
LangCell-CE 是cell encoder+分类头
2.2.1 细胞类型注释 (微调)
研究评估了 LangCell-CE 在经典的细胞类型注释任务上的表示能力。结果表明,模型在所有三个数据集上都实现了 SOTA 性能。说明LangCell 成功地将非结构化知识注入了细胞编码器,增强了对 scRNA-seq 数据的理解。此外,在 LiverCross 上的实验证明了 LangCell 在跨数据集任务中的有效性

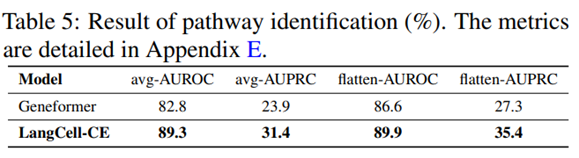
2.2.2 通路识别
目前我们还无法确定 LangCell-CE 的出色表现是因为它在学习细胞表示能力上的全面提升,还是由于它在这一特定任务上的深刻见解。
为了验证这一点,研究探索了一个预训练中未涉及的新细胞身份:细胞通路,并围绕它设计了一个具有挑战性的表示学习任务。
对于每个单细胞,模型需要从 41 个重要通路中识别多个通路,这实质上构成了一个具有 41 个独立标签的多标签二元分类问题。
考虑到由于只有少量细胞表达每个通路而导致的数据不平衡,研究在微调过程中使用了 focal loss。
表E所示,LangCell 在 AUROC 和 AUPRC 这两个指标上显著超越了现有的最先进模型 Geneformer,在这一具有挑战性的任务中表现出色。
3.模型使用
模型代码开源在这:https://github.com/PharMolix/LangCell
3.1 环境配置
推荐用python 3.9.18,需要首先安装好Geneformer。Geneformer可以从Hugging Face中安装:
https://huggingface.co/ctheodoris/Geneformer
对于LangCell:
git clone git@github.com:PharMolix/LangCell.git
pip install -r requirements.txt
3.2 预训练权重下载
模型的 checkpoint 分为 text_bert、cell_bert、text_proj、cell_proj、ctm_head 五个模块,可以根据下游任务需求选择加载必要的模块。
其中,cell_bert 为标准的 Huggingface BertModel;text_bert 为 utils.py 中提供的多功能编码器;cell_proj 和 text_proj 为线性层,分别将 cells 和 text 中 [CLS] 位置对应的模型输出映射到统一的特征空间;ctm_head 为线性层,将 text_bert 的输出映射到进行 Cell-Text Matching 时的匹配分数。
具体加载方法请参考 LangCell-annotation-zeroshot/zero-shot.ipynb 中的使用方法。
下载:https://drive.google.com/drive/folders/1cuhVG9v0YoAnjW-t_WMpQQguajumCBTp

其中obo.json是常见细胞身份的文字描述。
3.3 使用模型
数据预处理
与data_preprocess/preprocess.py中的示例类似,可以使用scanpy读取任意单细胞数据并将其处理成模型接受的格式。处理方法类似于Geneformer。
细节:https://huggingface.co/ctheodoris/Geneformer/blob/main/examples/tokenizing_scRNAseq_data.ipynb
LangCell 零样本细胞类型注释
作者为这个任务准备了一个演示数据集https://drive.google.com/drive/folders/1cuhVG9v0YoAnjW-t_WMpQQguajumCBTp?usp=sharing;
下载数据集并运行 LangCell-annotation-zeroshot/zero-shot.ipynb 即可。
LangCell 小样本细胞类型注释
通过对极少量数据进行小样本训练,LangCell 的性能可以得到进一步提升。
cd LangCell-annotation-fewshot/
python fewshot.py --data_path [data_path] --model_path [model_path] --nshot [nshot] --device [device]
LangCell-CE 细胞类型注释
实验证明,仅使用 LangCell 的 Cell Encoder(LangCell-CE)进行微调也可以在下游任务上取得优异的表现。使用以下命令使用 LangCell-CE 运行微调和少样本实验:
cd LangCell-CE-annotation/
python finetune.py --data_path [data_path] --model_path [model_path] --device [device]
python fewshot.py --data_path [data_path] --model_path [model_path] --nshot [nshot] --device [device]










![HTB:Funnel[WriteUP]](https://i-blog.csdnimg.cn/direct/5fccefa72f6e40648705eb54d581e560.png)







