目录
- 写在前面
- 一、使用string分析数据
- 二、使用Cytoscape构建网络
- 1. 导入TSV文件
- 2. Analyze Network
- 3. Generate Style
- 4. CytoNCA计算Betweenness
- 三、美化网络
- 1. 根据Betweenness调整网络
- 2. 选择你需要的蛋白,做个双环网络图
- 3. 调整字体大小颜色、气泡大小等
- 4. 保存图片及图例
写在前面
在做蛋白网络互作图时,使用string出的图不太好看,想要一个好看一些的图。在看了一些攻略之后,自己也能绘制出还不错的图,奈何步骤太多,怕自己遗忘,所以就将过程记录下来。
一、使用string分析数据
string官网: https://cn.string-db.org/
首先打开string,找到Multiple proteins,将目标基因输入,然后选择物种进行分析。

之后,string会自动把gene name转为protein ID,然后点击CONTINUE。

这时会出现一个简易的PPI网络图,可以点击More或者Less来控制节点数量,也可以在Settings中设置具体的筛选标准。
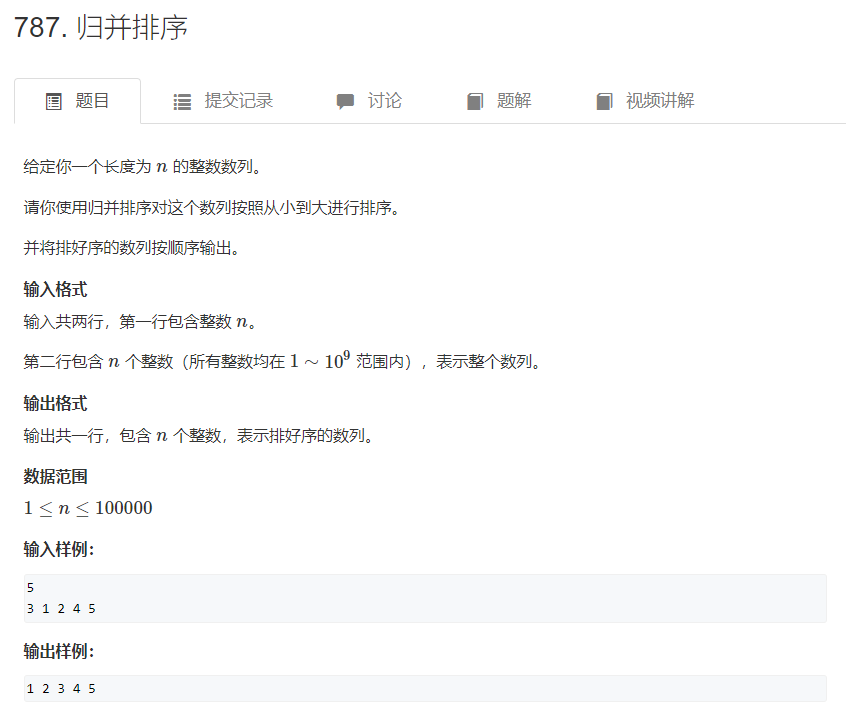
第四个TSV文件可以导入Cytoscape,我们把这个文件下载下来,然后再继续操作。

二、使用Cytoscape构建网络
Cytoscape Version: 3.7.2
Java Version: 1.8.0_162
注意软件版本,后续版本有些按钮被删除了。
1. 导入TSV文件

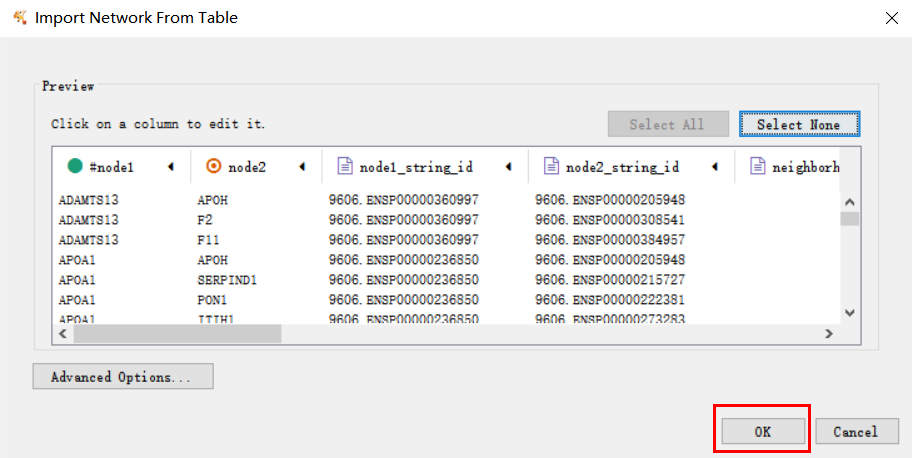
2. Analyze Network

这里我们的蛋白连接是没有指向性的,所以选择第二个。
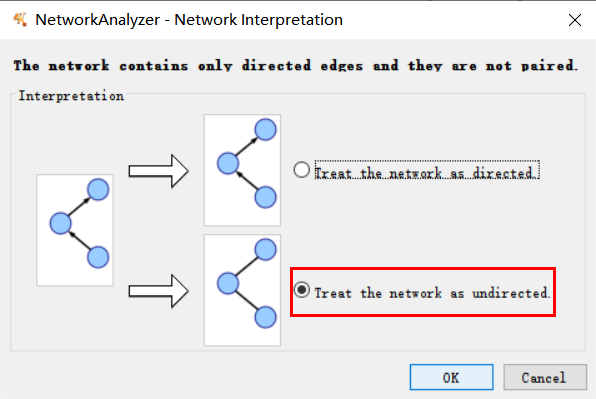
3. Generate Style

degree是指一个蛋白与其余蛋白连接的数量,combined_score是指连接度(TSV文件中包含的指标)。

4. CytoNCA计算Betweenness
经过某个节点的最短路径的数目来刻画节点重要性的指标就称为介数中心性(Betweenness centrality)

三、美化网络
1. 根据Betweenness调整网络

删除单个游离节点,快捷键(CTRL+6)选择与选中节点有连接的节点。
调整好的网络如下:

2. 选择你需要的蛋白,做个双环网络图
CTRL+鼠标:选择蛋白

先将两个圈画出来

将外圈扩大

将两个圈套在一起

3. 调整字体大小颜色、气泡大小等

其实这里有个不严谨的地方,我调整了Size中的Current Mapping中的Node Size。这里的节点大小应该按照degree自动生成的,但是有的节点实在是太小了,为了美观,我就手动把调整了节点大小,但是大概趋势是符合的。
最后看一下美化前和美化后的对比吧!


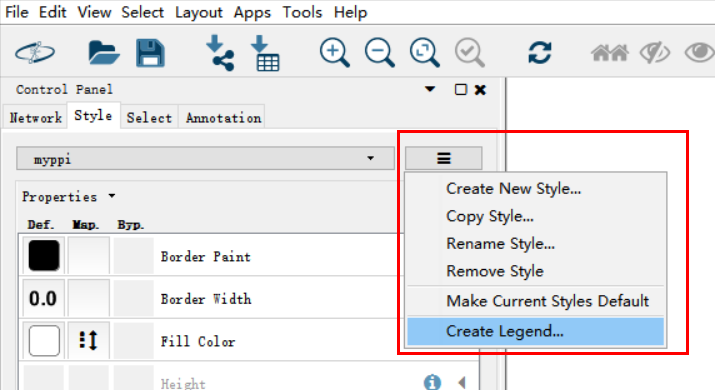
4. 保存图片及图例
保存为PDF,方便后续处理


保存图例

参考:
https://www.bilibili.com/video/BV1b7411m7Jg/?spm_id_from=333.337.search-card.all.click&vd_source=9ce796351a7e29b016ec545e9117bc55
https://www.bilibili.com/video/BV1n34y1b7by/?spm_id_from=333.788.recommend_more_video.0&vd_source=9ce796351a7e29b016ec545e9117bc55









![LeetCode[剑指offer 40]最小的k个数](https://img-blog.csdnimg.cn/img_convert/fc4126d762ee9f82d66a93b9b722ad6a.png)