1.题目
DNA序列 由一系列核苷酸组成,缩写为 'A', 'C', 'G' 和 'T'.。
例如,"ACGAATTCCG" 是一个 DNA序列 。
在研究 DNA 时,识别 DNA 中的重复序列非常有用。
给定一个表示 DNA序列 的字符串 s ,返回所有在 DNA 分子中出现不止一次的 长度为 10 的序列(子字符串)。你可以按 任意顺序 返回答案。
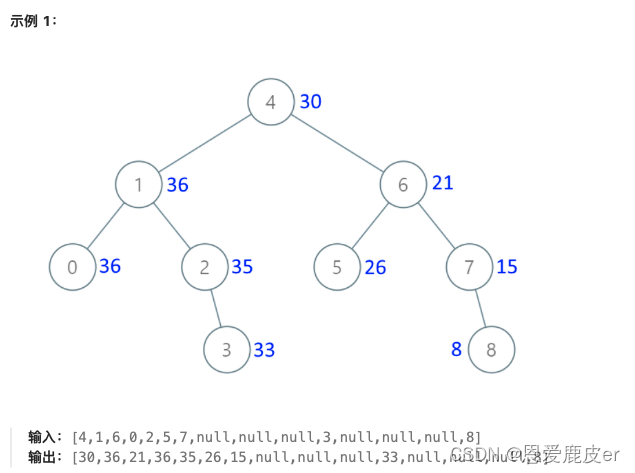
示例 1:
输入:s = "AAAAACCCCCAAAAACCCCCCAAAAAGGGTTT"
输出:["AAAAACCCCC","CCCCCAAAAA"]
示例 2:
输入:s = "AAAAAAAAAAAAA"
输出:["AAAAAAAAAA"]
提示:
0 <= s.length <= 10^5
s[i]=='A'、'C'、'G' or 'T'
来源:力扣(LeetCode)
2.思路及代码
返回所有在 DNA 分子中出现不止一次的 长度为 10 的序列,即统计不同子字符串的出现的次数,并将出现次数大于1的储存起来。至于如何统计就要用到STL中的map或unordered_map,简单来讲,两者储存的元素都是键(第一个元素)值(第二个元素)对,比如:map<int, int>就类似于vector<int>,而map<string, int>就可以通过string来查询int。至于两者的区别,map的内部是一个红黑树,会根据键的大小自动排序,unordered_map的内部则是哈希表,查找效率更高但更占内存。(具体可看大佬)显然这题用unordered_map更好,但map也不会超时。代码如下:
class Solution {
public:
vector<string> findRepeatedDnaSequences(string s) {
//当s的长度小于等于10直接返回空数组
if (s.size() <= 10) return {};
vector<string>arr;
unordered_map<string, int>mp;
string::iterator it = s.begin();
//遍历字符串,统计所有长度为10的字符串
for (; it <= s.end() - 10; ++it) {
string tmp(it, it + 10);
++mp[tmp];
}
unordered_map<string, int>::iterator it_map = mp.begin();
//将出现次数超过1的字符串加入数组
for (; it_map != mp.end(); ++it_map) {
if (it_map->second > 1) {
arr.push_back(it_map->first);
}
}
return arr;
}
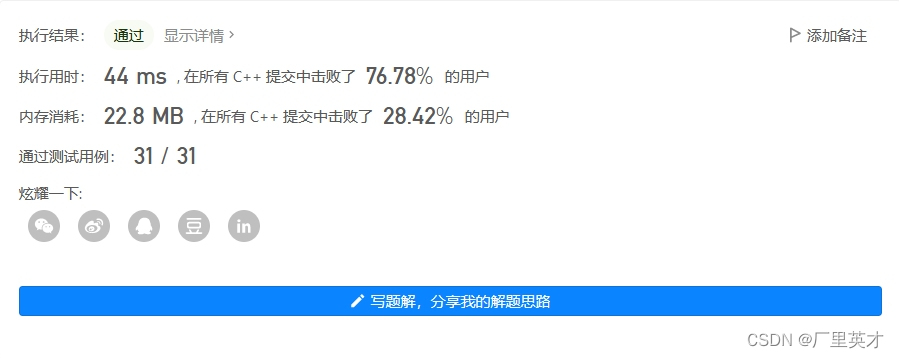
};map的运行结果:

unordered_map的运行结果:

![[SpringDataMongodb开发游戏服务器实战]](https://img-blog.csdnimg.cn/direct/16f74fa4de8044cea54c4cceff13e856.png)