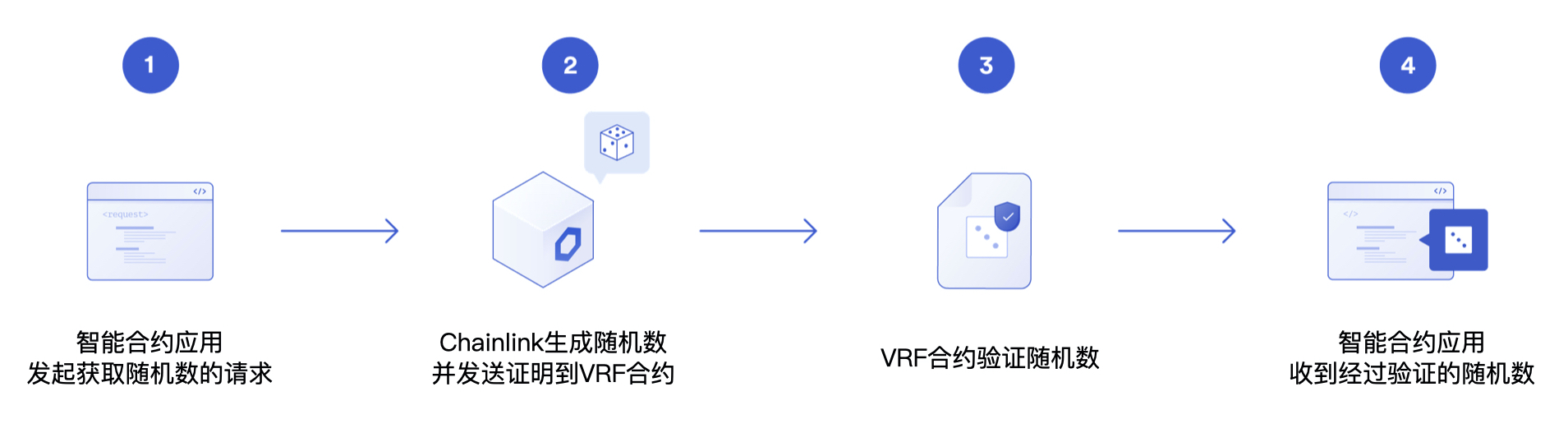
1.MedPy简介
MedPy 是一个图像处理库和针对医学(如高维)图像处理的脚本集合,此处主要讨论利用该库计算常见的医学图像分割任务评价指标,如Dice、Jaccard、Hausdorff Distance、Sensitivity、Specificity、Positive predictive value等。
论文表格的表头一般使用评价指标的英文全称首字母大写简写,如PPV代表阳性预测值,故此处也给出。
-
Dice相似系数(Dice Similarity Coefficient,DSC)或Dice系数(Dice Coefficient,DC)
最常用的一项评价指标,用于有效衡量分割算法预测标签与真实标注标签的空间重叠程度,其对应值越大表示分割精度越高。
D C = 2 ∣ A ∩ B ∣ ∣ A ∣ + ∣ B ∣ DC=\frac{2|A\cap B|}{|A|+|B|} DC=∣A∣+∣B∣2∣A∩B∣ -
Jaccard相似系数(Jaccard Similarity Coefficient,JSC)或Jaccard系数(Jaccard Coefficient,JC)
与 Dice 相似系数指标相似,也是一种衡量两幅图像相似程度的指标,其由实际分割结果与真实标签的交集同二者并集的比值得出,反映了分割方法的准确程度,其值越高,分割结果越准确。
-
豪斯多夫距离(Hausdorff distance,HD)
表示预测分割区域边界与真实肿瘤区域边界之间的最大距离,其值越小代表脑肿瘤边界分割误差越小、质量越好。
d H ( X , Y ) = max { d X Y , d Y X } = max { max x ∈ X mind y ∈ Y ( x , y ) , max y ∈ Y min x ∈ X d ( x , y ) } d_H(X, Y)=\max \left\{d_{X Y}, d_{Y X}\right\}=\max \left\{\max _{x \in X} \operatorname{mind}_{y \in Y}(x, y), \max _{y \in Y} \min _{x \in X} d(x, y)\right\} dH(X,Y)=max{dXY,dYX}=max{x∈Xmaxmindy∈Y(x,y),y∈Ymaxx∈Xmind(x,y)} -
95% 豪斯多夫距离(95% Hausdorff distance,HD95)
为了排除一些离群点造成的不合理距离,保持整体数值稳定性,一般选择从小到大排名前 95%的距离作为实际豪斯多夫距离,称之为 95% 豪斯多夫距离。
-
灵敏度(Sensitivity)
也称真阳性率(True Positive Rate)和召回率(Recall),表示预测为正的肿瘤标签占真实肿瘤标签的比例,其值越大,漏检率越低。
-
特异度(Specificity)
也称真阴性率,表示预测为正的背景标签占所有真实背景标签的比例,其值越高则误诊率越低。
-
阳性预测值(Positive Predictive Value,PPV)
也称精确度,指在所有样本的预测结果中,真阳性占所有阳性样本的比例,其值越大越好。
2.MedPy安装
medpy安装较为简单,此处以Ubuntu 18.04.5 LTS系统的root用户为例,其他情况详见参考文献安装即可。
为了启用graph-cut包,我们需要安装下列软件包
sudo apt-get install libboost-python-dev build-essential
然后使用阿里云镜像源高速安装medpy
sudo pip install medpy -i http://mirrors.aliyun.com/pypi/simple/ --trusted-host mirrors.aliyun.com
3.MedPy常用函数
3.1 medpy.io.load(image)
加载图像并返回包含图像像素内容以及头部对象的 ndarray
import os
from medpy.io import load,save
# 文件夹路径和文件名
dirname="/dataset/RSNA_ASNR_MICCAI_BraTS2021_TrainingData_16July2021/BraTS2021_00000/"
basename="BraTS2021_00000_seg.nii.gz"
full_path=os.path.join(dirname,basename)
# 加载脑肿瘤标记图像返回图像数据数组和头部信息
image_data, image_header = load(full_path)
print("图像数据类型为{},图像数据形状为{}".format(type(image_data),image_data.shape))

# 获取一个切片数组
slice_0=image_data[:,:,77]
print("切片数组的形状为{},切片数组的数据类型为{}".format(slice_0.shape,slice_0.dtype))

# 转置显示灰度图
plt.imshow(slice_0.T, cmap="gray")

# 将纵坐标设置为对数尺度,等宽区间数量设置为为8个绘制切片数组灰度直方图
plt.hist(image_data.flatten(), bins=8, log=True)

3.2 medpy.metric.binary.dc(result, reference)
计算二值图像之间的Dice系数(也称为索伦森指数)
首先编辑第三方库源代码在最后追加一行输出语句,保存退出后重启kernel。该输出语句指示此时调用的numpy定义,若调用dc函数前有输出,则表示调用依赖numpy的函数,否则调用依赖jax.numpy的兼容函数实现加速运算
vim /opt/conda/lib/python3.7/site-packages/medpy/metric/binary.py
然后定义统一的一个数组大小参数,最后使用魔术命令%%timeit评估CPU和GPU计算两种方法的运行效率
# 设置数组形状为10000*10000的元组
tup=(10000,10000)
-
调用依赖numpy的函数
%%timeit from medpy.metric.binary import dc import numpy as np # 设置伪随机数种子 seed=np.random.seed(6) # 定义预测结果和真实标记数组 predict=np.random.randint(0,4,size=tup) ground_truth=np.random.randint(0,4,size=tup) # 计算Dice相似系数 dice1=dc(predict,ground_truth) print("Dice相似系数为{}".format(dice1))

-
调用依赖jax.numpy的兼容函数
%%timeit from medpy.metric.binary import dc import jax.numpy as numpy # 重载numpy定义使得dc函数底层可以加速计算 from jax import random # 设置伪随机数种子 rng1=random.PRNGKey(1) rng2=random.PRNGKey(2) # 定义预测结果和真实标记数组 predict=random.randint(key=rng1,shape=tup,minval=0,maxval=4) ground_truth=random.randint(key=rng2,shape=tup,minval=0,maxval=4) # 计算Dice相似系数 dice2=dc(predict,ground_truth) print("Dice相似系数为{}".format(dice2))

3.3 medpy.metric.binary.jc(result, reference)
计算两个图像中二值对象间的Jaccard系数,下面通过两种方式计算
- 直接调用第三方库函数计算jaccard系数
%%timeit
from medpy.metric.binary import jc
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=tup,minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=tup,minval=0,maxval=4)
# 直接计算Jaccard相似系数
jaccard=jc(predict,ground_truth)
print("Jaccard相似系数为{}".format(jaccard))

- 通过Dice系数和Jaccard系数的如下关系推导计算
KaTeX parse error: Undefined control sequence: \nonumber at position 29: …c{Dice}{2-Dice}\̲n̲o̲n̲u̲m̲b̲e̲r̲ ̲
%%timeit
from medpy.metric.binary import dc
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=tup,minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=tup,minval=0,maxval=4)
# 通过Dice相似系数推导Jaccard相似系数
dice=dc(predict,ground_truth)
jaccard=dice/(2-dice)
print("通过Dice相似系数推导Jaccard相似系数为{}".format(jaccard))

- 对比两种计算结果、源代码实现、运行效率可知,两种方式结果一样但通过Dice系数推导Jaccard系数运行效率更高,其原因是将一个统计非零值的 O ( n ) O(n) O(n)时间复杂度的函数替换成了 O ( 1 ) O(1) O(1)时间复杂度的简单推导。
3.4 medpy.metric.binary.hd(result,reference,voxelspacing=None,connectivity=1,)
计算两个图像中二值对象之间的(对称)豪斯多夫距离(HD)。它被定义为对象之间的最大表面距离。
%%timeit
from medpy.metric.binary import hd,hd95
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算豪斯多夫距离
hausdorff_distance=hd(predict,ground_truth)
print("豪斯多夫距离为{}".format(hausdorff_distance))

3.5 medpy.metric.binary.hd95(result,reference,voxelspacing=None,connectivity=1,)
计算两个图像中二值对象之间的(对称)豪斯多夫距离 (HD) 的第 95 个百分位数。与豪斯多夫距离相比,该指标对于小异常值稍微稳定一些,通常用于生物医学分割挑战。
%%timeit
from medpy.metric.binary import hd,hd95
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算95%豪斯多夫距离
hausdorff_distance95=hd95(predict,ground_truth)
print("95%豪斯多夫距离为{}".format(hausdorff_distance95))

3.6 medpy.metric.binary.recall(result, reference)
返回召回率
%%timeit
from medpy.metric.binary import recall
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算召回率/灵敏度/真阳性率
sensitivity=recall(predict,ground_truth)
print("召回率/灵敏度/真阳性率为{}".format(sensitivity))

3.7 medpy.metric.binary.sensitivity(result, reference)
返回灵敏度,内部实现直接调用recall
%%timeit
from medpy.metric.binary import sensitivity
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算召回率/灵敏度/真阳性率
recall=sensitivity(predict,ground_truth)
print("召回率/灵敏度/真阳性率为{}".format(recall))

3.8medpy.metric.binary.true_positive_rate(result, reference)
返回真阳性率,内部实现直接调用recall
%%timeit
from medpy.metric.binary import true_positive_rate
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算召回率/灵敏度/真阳性率
sensitivity=true_positive_rate(predict,ground_truth)
print("召回率/灵敏度/真阳性率为{}".format(sensitivity))

3.9medpy.metric.binary.specificity(result, reference)
返回特异度
%%timeit
from medpy.metric.binary import specificity
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算特异度/真阴性率
tnr=specificity(predict,ground_truth)
print("特异度/真阴性率为{}".format(tnr))

3.10medpy.metric.binary.true_negative_rate(result, reference)
返回真阴性率
%%timeit
from medpy.metric.binary import true_negative_rate
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算特异度/真阴性率
specifity=true_negative_rate(predict,ground_truth)
print("特异度/真阴性率为{}".format(specifity))

3.11medpy.metric.binary.precision(result, reference)
返回精确度
%%timeit
from medpy.metric.binary import precision
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算精确度/阳性预测值
Precision=precision(predict,ground_truth)
print("精确度/阳性预测值为{}".format(Precision))

3.12 medpy.metric.binary.positive_predictive_value(result, reference)
返回阳性预测值
%%timeit
from medpy.metric.binary import positive_predictive_value
import jax.numpy as numpy
from jax import random
# 设置伪随机数种子
rng1=random.PRNGKey(1)
rng2=random.PRNGKey(2)
# 定义预测结果和真实标记数组
predict=random.randint(key=rng1,shape=(50,50),minval=0,maxval=4)
ground_truth=random.randint(key=rng2,shape=(50,50),minval=0,maxval=4)
# 计算精确度/阳性预测值
ppv=positive_predictive_value(predict,ground_truth)
print("精确度/阳性预测值为{}".format(ppv))

3.13 medpy.io.save(arr, filename, hdr=False, force=True, use_compression=False)
使用“hdr”中编码的信息将图像“arr”保存为文件名。目标图像格式由“文件名”后缀确定。如果“force”参数设置为 true,以静默方式覆盖已存在的映像。否则将引发错误。
import os
from medpy.io import load,save
# 文件夹路径和文件名
dirname="/dataset/RSNA_ASNR_MICCAI_BraTS2021_TrainingData_16July2021/BraTS2021_00000/"
basename="BraTS2021_00000_seg.nii.gz"
full_path=os.path.join(dirname,basename)
# 加载脑肿瘤标记图像返回图像数据数组和头部信息
image_data, image_header = load(full_path)
# 将图像数据保存为BraTS2021_00000_seg.nii
save(image_data,"BraTS2021_00000_seg.nii")

4.参考文献
-
MedPy — MedPy 0.4.0 documentation
-
Precision_and_recall
-
Sensitivity_and_specificity
-
Confusion_matrix#Table_of_confusion




![[附源码]计算机毕业设计汽配管理系统Springboot程序](https://img-blog.csdnimg.cn/22ad3cd0ec2e49a08e165edb871b83f1.png)