原文标题:Sc2Mol:a scaffold-based two-step molecule generator with variational autoencoder and transformer
论文地址:Sc2Mol: a scaffold-based two-step molecule generator with variational autoencoder and transformer | Bioinformatics | Oxford Academic
代码链接:https://github.com/aspuru-guzik-group/chemical_vae.
一、思想
通常分子生成的表征为SMILES string或者Graph。包括两个任务:分布学习(从给定数据集中学习分布,并随机抽样生成与具有与训练数据集相似的新分子)+分子优化(根据给定的生成分数,修改输入分子)。
分子生成应当同时支持随机从头生成+先导分子优化。(这个想法和分子生成应当性质驱动的生成模型应当擅长性质识别的想法都很棒,可以融合)
性质驱动的生成模型应当擅长性质识别:Nat. Mach. Intell 2023 | RT:首个统一分子性质预测(回归) 与条件生成的模型_羊飘的博客-CSDN博客
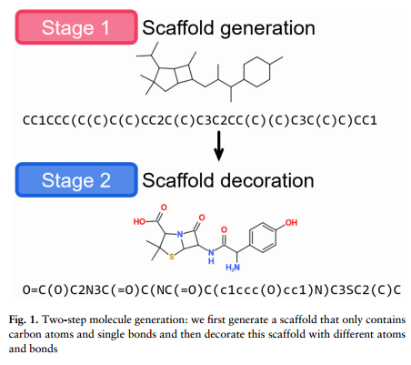
首先通过变分自编码器(VAE)生成仅包含碳原子和单键的骨架 ( VAE提供了支架分布以寻找新的骨架)。然后,通过transformer改变原子和键类型来丰富生成的骨架,从而产生具有所需属性的分子(见图1)。我们的模型不需要额外的专业知识,例如语法规则和预定义的子结构,并从随机变量或给定骨架生成分子。
二、Materials and methods
1、Problem formulation
使用SMILES,使用原分子x = [x1,x2,...,xl],生成新分子y=[y1,y2,...,yl']
2、Scaffold generation
假设潜在先验分布是标准正态分布。单个字符用one-hot向量表示,输入SMILES字符串可以用矩阵表示。对embedding E:
![]()
通过对字符串和残差连接堆叠m个gated卷积神经网络(GatedConv)来实现VAE:

然后使用maxpool层,接两个全连接层得到mean,var,拟合分布:

得到latent vector z, 解码器重构SMILES:
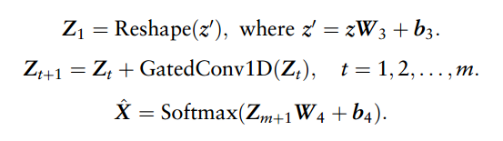
对通用C scaffold建模,然后从空间抽取随机变量构建骨架,支持scaffold hopping
3、Scaffold decoration
通过transformer装饰骨架。骨架只是一个概率矩阵,且argmax是不可微的,因此,在训练过程中采用teacher forcing(ground truth scaffold)避免不可微,对于transformer的输入如下:
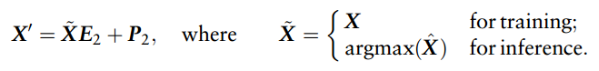
4、End-to-end style training of the VAE and transformer
VAE:

Transformer:
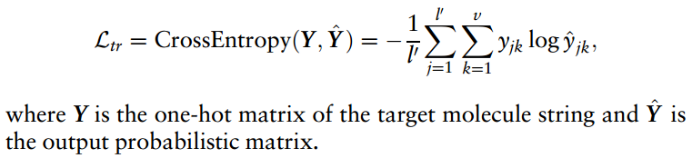
总损失:
![]()
训练

5、Two types of molecule generation:
允许两种类型的生成。
从正态分布中随机抽取一个变量,没有任何专业知识,使用VAE和transformer解码器获取分子
输入骨架,基于骨架获取适当的原子和键的分子
在模型中添加一个有效性检查组件(用于根据SMILES语法对生成的分子进行筛选,如果无效就抛弃继续生成)。
三、Experiments
1、Dataset
MOSES数据集(分子量150-350Da,无带电原子,无大于8个原子的环,原子仅仅限于H,C,N,O,F,S,Cl,Br)。分子都是药物样(通过药物化学过滤器+PAINS过滤器)
160W训练 + 17.6W测试+ 17.6W新骨架集
2、Experiment setting
维数d为256,潜在维数dz为64; VAE和transformer都有一个三层编码器和一个三层解码器。VAE中所有门控卷积层的核大小为3,步长为1。transformer采用heads=4,feedforward=1024。将batch size为64,采用Adam优化器,drop-out rate为0.1。
RDKit 进行数据预处理,并在NVIDIA GeForce GTX 1080 Ti GPU,Tensorflow
3、Baselines

4、Evaluation measures
任务1:从标准正态分布中随机抽取了3W个样本,这些样本是VAE decoder和transformer的输入。
Metrics:Validity、Uniqueness、Novelty,Recovery
检查了分子量的分布,计算了辛醇-水分配系数(logP) 和药物相似性(QED)的定量估计
任务2和任务3:从测试/新支架组中随机选择3W个分子并提取骨架,然后将其作为整个模型的输入。
Metrics:Recovery(所需有效生成的分子(与测试集中相应的参考分子相同)与完全有效生成的分子的比率)、Similarity(生成分子的指纹[半径为2的1024位扩展连接指纹(ECFP4) ]与测试集中相应参考分子的平均谷本相似度)、Scaffold similarity (SS)(生成的分子骨架指纹与测试集中对应参考分子骨架指纹的平均谷本相似度。中等SS是有利的,因为高SS意味着没有新的支架,低SS意味着在不考虑输入支架的情况下任意生成)
5、Results
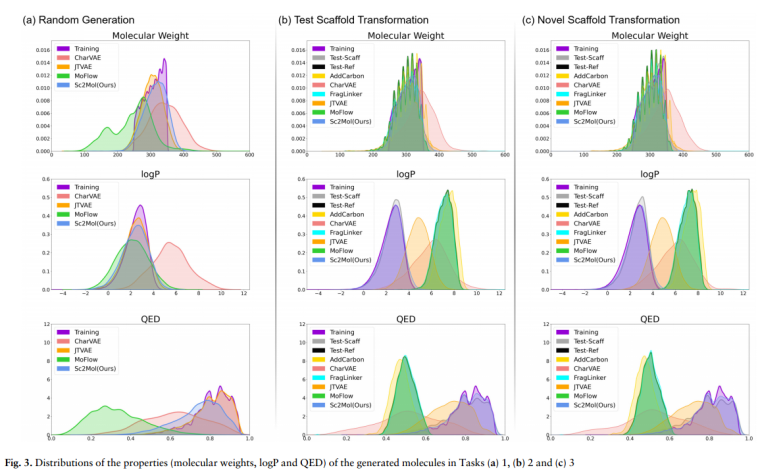
Task 1: Random generation

由于受益于有效性检查组件,模型也实现了100%的有效性。
图3a JTVAE和模型显示出更接近训练集的分布。对于QED(可以受到更多物理特征的影响,例如分子的极性表面积),由于JTVAE预定义了子图词汇表,因此JTVAE的分布与训练集最相似。
在没有任何先验知识的情况下,模型使分布稍微偏离训练集,而CharVAE使其偏离得更大。MoFlow生成的大多数分子QED明显较低,其分布与训练集距离较远
Task 2: Test scaffold transformation

获得了最好的有效性,唯一性和相似性,甚至恢复了一些(4.93%)所需的参考分子,尽管其新颖性有所下降。我们的模型的SS也下降到一个中等值(72.94%),表明模型总体上表现最好
图3b显示了Task 2生成的分子的分子量、logP和QED的分布。除CharVAE外,各组分分子量分布基本相似。对于logP和QED, CharVAE和JTVAE的分布与其他分布没有明显的重叠。
Task 3: Novel scaffold transformation
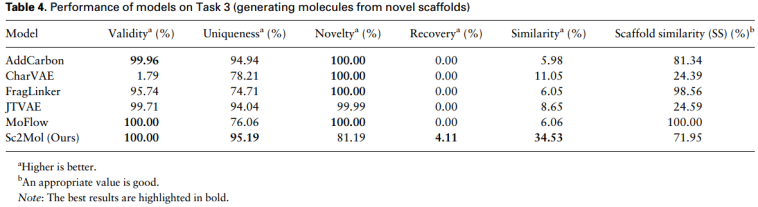
图3c显示了所有模型生成的分子分布。结果与Task 2完全一致。
Ablation study
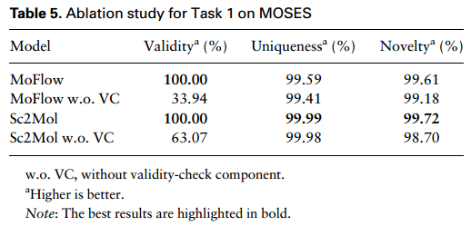
由于SMILES语法检查的成本很低,并且即使没有有效性检查,模型仍然可以达到可接受的有效性,因此向我们的模型引入有效性检查不会增加相当大的计算量

Examples of the generated molecules

Case study 1: Auglurant

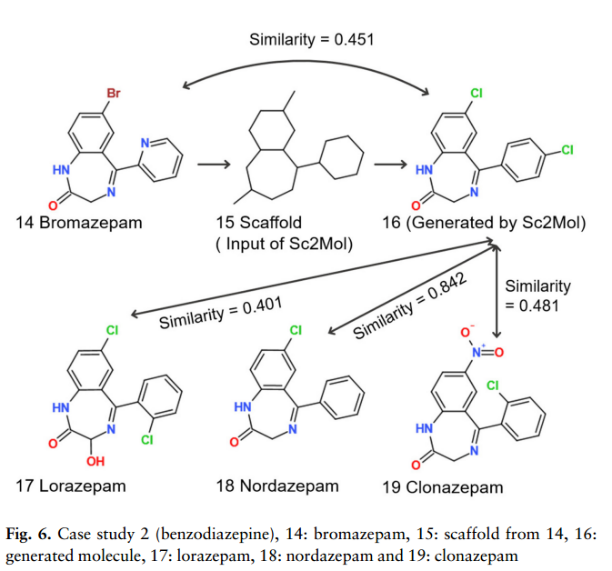
Case study 2: Benzodiazepines