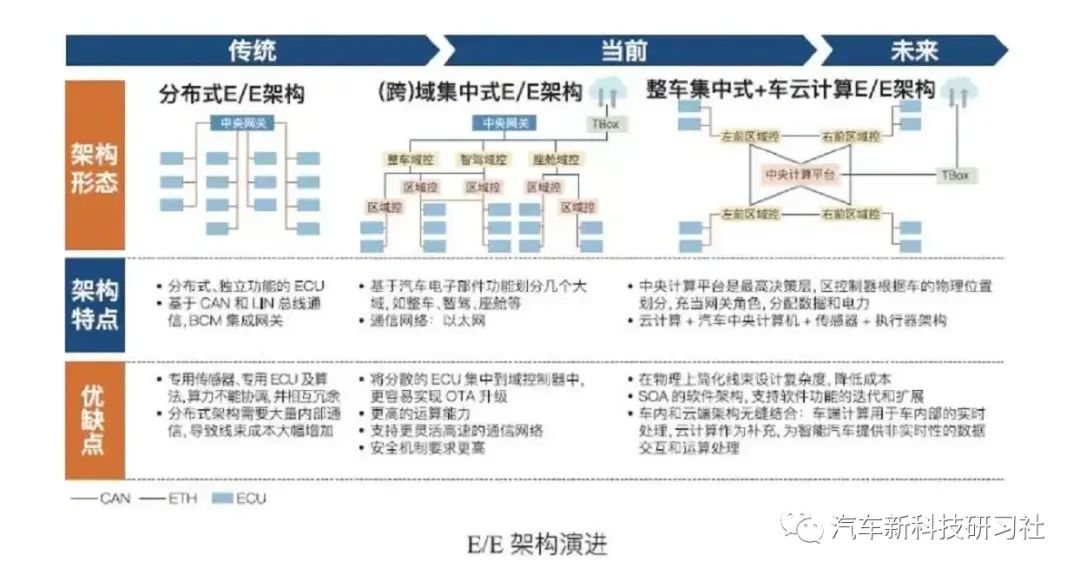
KaplanMeierFitter是lifelines库中用于计算生存分析的一个类。使用KaplanMeierFitter类,我们可以对我们的数据进行不同组之间的生存分析,比如根据年龄、性别、治疗手段等分类变量进行分组分析。
该类包含许多方法和属性,其中最常用的是fit()方法和plot()方法。
fit(): 该方法根据输入的时间和截尾数据来估计生存函数,返回一个KaplanMeierFitter对象。plot(): 根据估计的生存函数绘制Kaplan Meier曲线,并可以通过传递参数来自定义曲线的颜色、标签等属性。
简单案例
例如,以下代码块展示了如何使用KaplanMeierFitter类对数据进行分组生存分析并绘制生存曲线:
from lifelines import KaplanMeierFitter
kmf = KaplanMeierFitter()
# 分组后的时间和截尾数据
T1 = df.loc[df['Stage_group'] == 1, 'Time']
E1 = df.loc[df['Stage_group'] == 1, 'Censor']
T2 = df.loc[df['Stage_group'] == 2, 'Time']
E2 = df.loc[df['Stage_group'] == 2, 'Censor']
# 对两组数据分别进行生存分析
kmf.fit(T1, event_observed=E1, label='Stage III')
kmf.fit(T2, event_observed=E2, label='Stage IV')
# 绘制两条生存曲线
kmf.plot()
在上面的代码中,我们首先创建一个KaplanMeierFitter对象,并使用fit()方法对两组数据分别进行生存分析。然后,我们通过plot()方法绘制了两条生存曲线。
其中,生存曲线下方的阴影部分是95%置信区间,可以帮助我们评估估计值的精度。
KaplanMeierFitter类中的fit()用法
KaplanMeierFitter类中的fit()方法可以根据输入的时间和截尾数据来估计生存函数。该方法有以下几个参数:
durations: 表示各个事件的时间数列,可以是一个numpy数组或一个pandas的Series。event_observed: 表示事件是否被观察到,也就是截尾信息的数列,可以是一个numpy数组或一个pandas的Series。该数列中1表示事件被观察到,0表示事件未被观察到。timeline: 表示需要计算生存函数的时间点,可以是一个numpy数组或一个pandas的Series。如果不指定,则默认使用所有时间点。entry: 表示入组时间,也就是开始观测的时间点,可以是一个numpy数组或一个pandas的Series。如果不指定,则默认所有样本的入组时间均为0。
下面是一个具体使用fit()方法的示例:
from lifelines import KaplanMeierFitter
import pandas as pd
# 读取数据
data = pd.read_csv('survival_data.csv')
# 创建KaplanMeierFitter对象
kmf = KaplanMeierFitter()
# 对数据进行生存分析
kmf.fit(durations=data['Time'], event_observed=data['Event'])
在上面的代码中,我们首先使用pandas库读取了名为’survival_data.csv’的数据集,然后创建了一个KaplanMeierFitter对象。接着,我们调用fit()方法对数据进行生存分析,其中传入了时间和截尾数据。
在fit()方法中,我们可以使用label参数来为当前分析添加标签(label),方便与其他分析结果区分。例如:
kmf.fit(durations=T1, event_observed=E1, label='Group 1') # 添加标签'Group 1'
此外,KaplanMeierFitter类还提供了许多方法和属性,详细使用方法可以参见官方文档:
常见的属性及其使用方法示例:
在执行完fit()方法后,我们可以通过许多属性来查看估计的生存函数。例如:
survival_function_: 包含估计的生存函数。cumulative_hazard_: 包含估计的累积危险函数。confidence_interval_: 包含估计的生存函数的置信区间。median_survival_time_: 表示中位生存时间。
具体展开讲
-
survival_function_: 返回一个pandas DataFrame,表示估计的生存函数,其中index是时间,columns是生存率,例如:print(kmf.survival_function_)输出结果如下:
timeline KM_estimate 0.0 1.000000 7.0 0.985714 8.0 0.971429 9.0 0.957143 10.0 0.942857 11.0 0.928571 14.0 0.914286 16.0 0.900000 18.0 0.885714 ... ...
在上面的示例中,survival_function_属性返回了一个DataFrame,其index是时间点,columns是生存率,即在该时间点之前尚存活的患者占全部患者的比例。我们可以使用Pandas的数据处理方法对其进行进一步的操作和可视化。
-
cumulative_hazard_:返回一个pandas DataFrame,表示累积危险函数的估计值,例如:print(kmf.cumulative_hazard_)输出结果如下:
KM_estimate timeline 0.0 0.000000 7.0 0.015152 8.0 0.030303 9.0 0.045455 10.0 0.060606 11.0 0.075758 14.0 0.090909 16.0 0.106061 18.0 0.121212 ... ...在上面的示例中,
cumulative_hazard_属性返回了一个DataFrame,其index是时间点,columns是累积危险函数的估计值。使用该属性可以进一步计算和可视化危险函数。 -
confidence_interval_: 返回一个pandas DataFrame,表示生存函数的置信区间,例如:print(kmf.confidence_interval_)输出结果如下:
KM_estimate_lower_0.95 KM_estimate_upper_0.95 timeline 0.0 1.000000 1.000000 7.0 0.940789 0.998414 8.0 0.882671 0.997766 9.0 0.828292 0.996916 10.0 0.772771 0.995863 11.0 0.724301 0.994594 14.0 0.677130 0.992959 16.0 0.632644 0.990914 18.0 0.586813 0.988317 ... ... ...在上面的示例中,
confidence_interval_属性返回了一个DataFrame,其index是时间点,columns是置信区间的上限和下限。可以使用该属性对生存函数进行统计分析。 -
median_survival_time_: 返回一个float值,表示中位生存时间,例如:print(kmf.median_survival_time_)输出结果如下:
369.0在上面的示例中,
median_survival_time_属性返回了一个float值,表示中位生存时间。可以使用该属性进行中位数分析。
KaplanMeierFitter类中的plot()用法
在生存分析中,KaplanMeierFitter类的plot()方法是可视化生存分析结果的重要工具。该方法可以绘制 Kaplan-Meier 生存曲线和其他一些统计信息,例如置信区间、中位生存时间等。下面详细解释kmf.plot()的用法和参数:
-
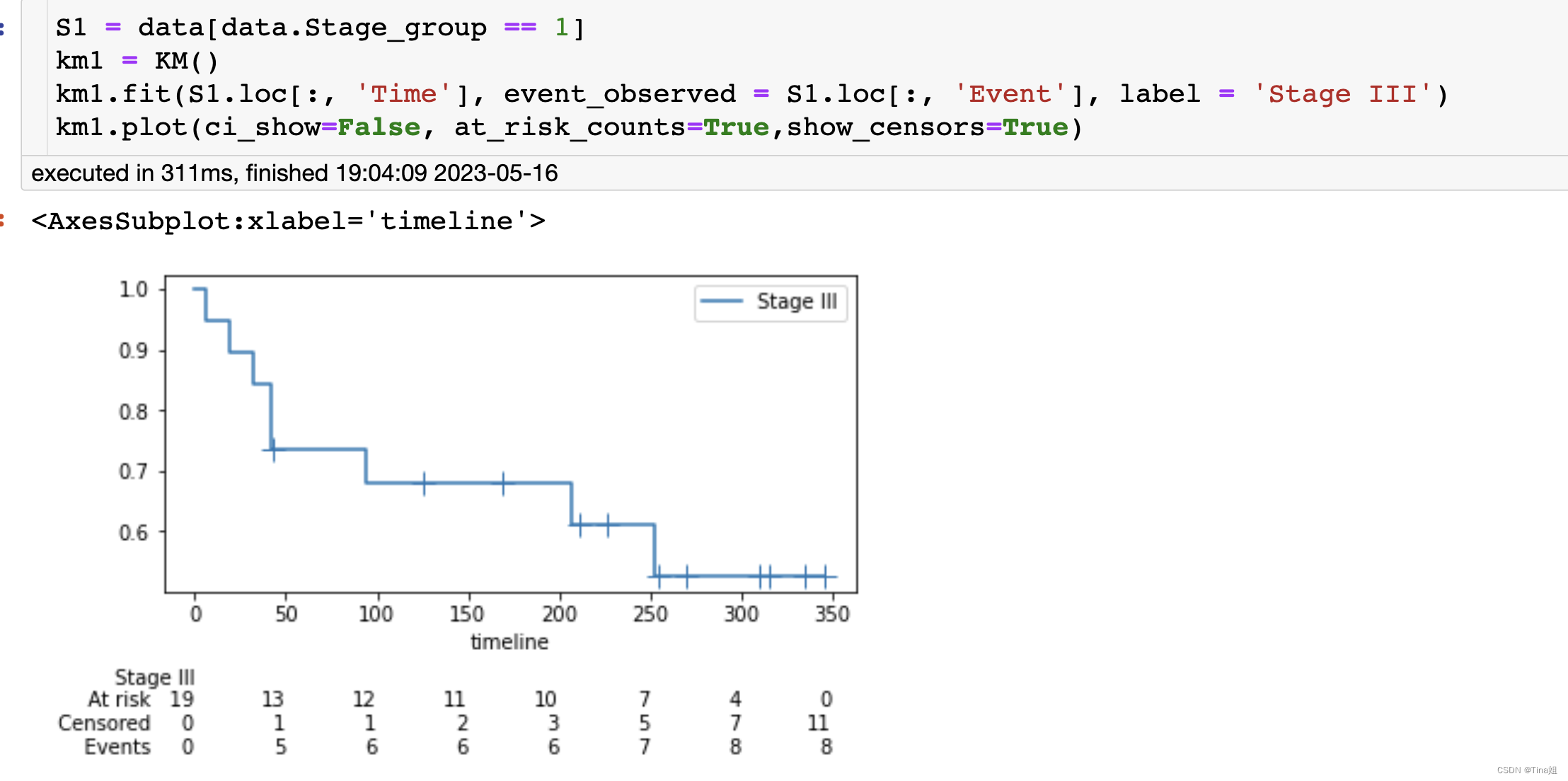
at_risk_counts: bool类型参数,默认值为 False,表示是否在图表中显示每个时间点处的“风险人数”,即仍然有可能发生事件的患者数量。若指定该参数为 True,则在图表顶部会出现黑色竖线,代表当前时刻有多少患者仍在观察中。 -
show_censors: bool类型参数,默认值为 False,表示是否在图表中显示被审查数据,即发生在患者失去随访前的事件。若指定该参数为 True,则在事件曲线上会出现垂直的标记。 -
cumulative_events: bool类型参数,默认值为 False,表示是否在图表中显示累积事件数。若指定该参数为 True,则除了 Kaplan-Meier 生存曲线外,还会绘制事件曲线,显示事件数随时间的增加情况。 -
legend: bool类型参数,默认值为 True,表示是否在图表中显示图例。若指定该参数为 False,则将不会显示图例。如果我们手动创建绘图,该参数可能会很有用。 -
ylabel: str类型参数,默认值为“生存概率”,表示y轴上的标签。可以更改为其他自定义标签,例如“累积生存率”。 -
ax: matplotlib.axes.Axes类型对象,默认值为 None,表示在哪个Matplotlib Axes对象上绘制。如果没有指定该参数,则将在新的图表中创建Axes对象并进行绘制。
此外,kmf.plot()还返回了一个matplotlib.axes.Axes类型对象,它可以进一步被修改或可视化。
下面是一个示例:
from lifelines import KaplanMeierFitter
import pandas as pd
import matplotlib.pyplot as plt
# 读取数据
data = pd.read_csv('data.csv')
# 处理数据
T = data['T'] # 时间列
E = data['E'] # 事件列
# 创建 Kaplan-Meier Fitter 对象
kmf = KaplanMeierFitter()
# 计算生存分析
kmf.fit(T, event_observed=E)
# 绘制 Kaplan-Meier 生存曲线
fig, ax = plt.subplots()
kmf.plot(ax=ax)
# 设置标签和标题
plt.xlabel('Time (days)')
plt.ylabel('Cumulative Survival Probability')
plt.title('Kaplan-Meier Plot')
# 显示图形
plt.show()
输出类似下面这个
注意:库的版本不同,参数有细微差别
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连