编译 | 于洲
今天我们介绍来自德国弗赖堡大学计算机科学系的Jörg K.H. Franke, Frederic Runge以及Frank Hutter发表在NeurIPS 2022会议上的工作,该文章介绍了一种新颖的基于概率的神经网络架构ProbTransformer,它是Transformer生态系统的一种层级增强,在数据分布恢复上有很强的学习能力。文章还介绍了GECO训练过程及其敏感性分析,以及在线自适应技术kappa annealing在ELBO变分训练中的潜在优势。文章还在实证分析中验证了ProbTransformer的有效性。同时还对该架构与领域内其他类似工作进行了对比。

背景介绍
我们的世界是模糊的,这反映在本文用来训练算法的数据上。当本文试图为自然过程建模时尤其如此,其中收集的数据受到噪声测量和测量技术差异的影响。有时,这个过程本身是不明确的,比如在RNA折叠的情况下,相同的核苷酸序列可以折叠成不同的结构。这表明一个预测模型应该有相似的概率特征来匹配它所建模的数据。因此,本文提出了一种分层潜在分布来增强最成功的深度学习模型——Probabilistic Transformer,以适应模糊性和数据分布。本文展示了该方法的好处:(1)在一个合成任务上获得了学习隐藏数据分布的能力,(2)在RNA折叠方面的最先进的结果显示了在高度模糊的数据上的优势,(3)通过隐式学习底层分布和优于现有工作,展示了其在基于性质的分子设计上的生成能力。
本文的创新与贡献:
本文提出了ProbTransformer,这是对Transformer生态系统的一种新的分层概率体系结构增强。
本文使用GECO的训练程序,分析其超参数κ的敏感性,并引入在线适应技术kappa退火,这可能有利于用ELBO进行变分训练。
全面的实证分析,验证ProbTransformer的能力学习和恢复数据分布在一个新的合成顺序分布任务,评估其在实践中处理数据模糊性的能力,通过实现最先进的RNA折叠性能,并通过在分子设计中优于现有工作来证明其生成特性。
方法介绍
ProbTransformer采用的是概率自动编码器和最大边际似然的ELBO优化方法。首先,使用Transformer模型对输入的序列进行编码,得到表示输入序列的向量。接着,使用Probabilistic Transformer模型对该向量进行处理,生成一个概率分布,表示每个目标标记在给定输入时的可能性。在推断时,本文可以从预测模型中采样得到不同的预测结果。但是,本文也可以使用各自(高斯)分布的均值,而不是从中采样。本文称之为均值推断,与样本推断相对应。
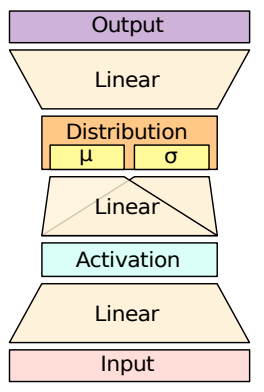
图1:概率前馈层
在训练过程中,研究者使用了GECO方法优化ELBO,并引入了kappa退火技术,为变分训练提供了潜在的好处。模型通过将生成条件与输入SMILES的分子属性相结合来隐式地学习训练数据的属性,使模型具有更好的生成和分类能力。在推断时,本文可以生成具有多个所需性能值的新分子。
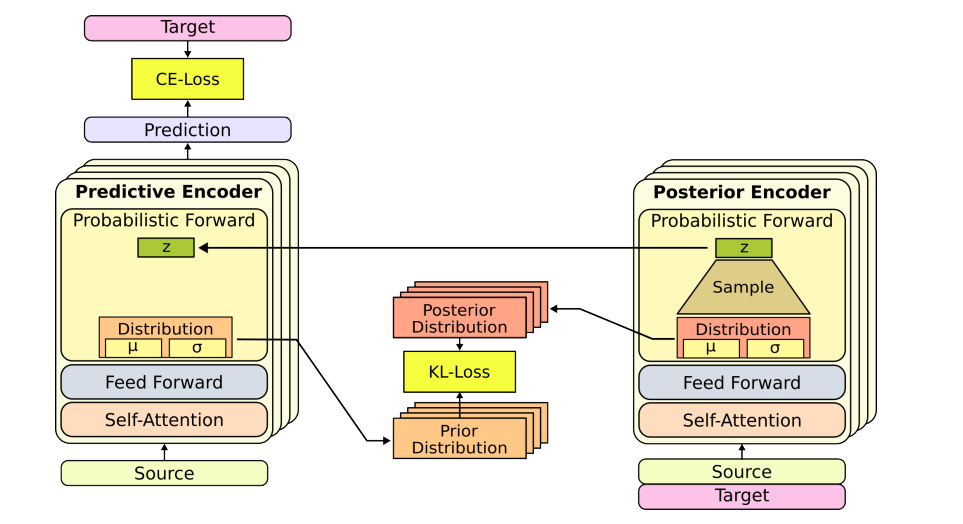
图2:ProbTransformer的训练设置:预测编码器和后验编码器联合训练
实验介绍
数据集:本文使用多个数据集,从公开出版物中收集数据样本,创建预定义验证集和测试集VL0、TS0。派生出结构相同的测试集TSsameStruc(149个样本)和模棱两可的测试集TSsameSeq(20个样本)。删除训练池中与测试和验证集相似度大于80%的样本,保留其余样本以捕捉不确定性。最终数据包括52007个训练样本、1299个验证样本和1304个测试样本。此外,收集来自多个数据库的数据样本,包括BpRNA、RNA Stralign、ArchiveII和RNA-Strand等。从TrainSetA和TrainSetB中删除重复和未包含成对碱基样本。限制序列长度不超过500个核苷酸。使用CD-HIT-EST-2D删除训练数据中与验证和测试样本相似度超过80%的样本,使用BpRNA对文件格式进行注释。
实验过程与结果:本文实验分别在合成的序列分布任务、RNA折叠和分子设计领域进行了实验。首先,在合成的序列分布任务中,通过使用ProbTransformer模型,在处理数据的模糊性方面表现出了最先进的性能。其次,在RNA折叠方面,实验结果表明ProbTransformer模型具有生成能力,表现优于现有的工作。最后,在分子设计领域,实验结果表明ProbTransformer模型可以生成多个期望的属性值的新颖分子,并在药物传递方面提供了更好的性能。该实验过程中,本文采用了类似的模型结构和训练方式,并通过使用ProbTransformer模型,将概率层集成到模型中,以更好地处理数据的模糊性和获得更好的生成能力。实验结果表明,ProbTransformer模型具有很强的实用性和可扩展性,可应用于多种领域。

表1:综合任务中五个随机种子的均值测度
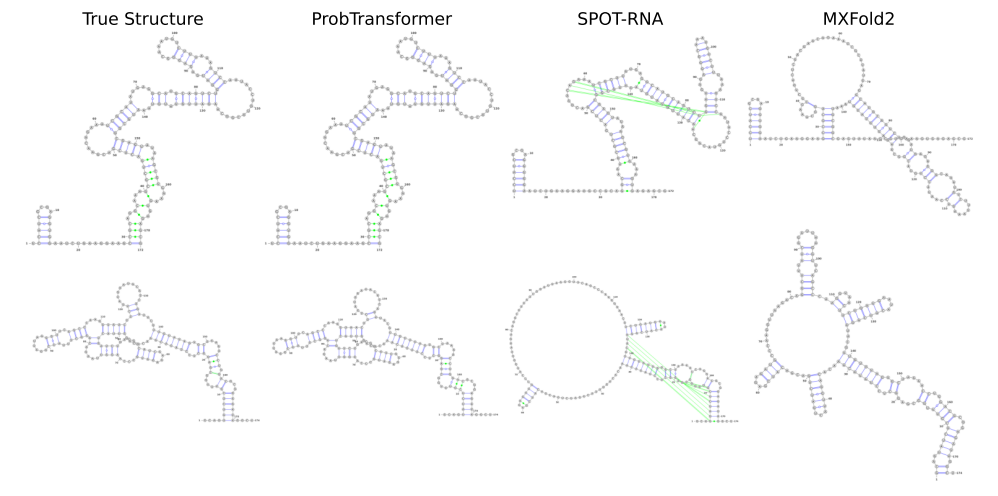
图3:基于深度学习的TS0挑战性RNA方法的预测示例
本文展示了(顶层)一个II族催化内含子(RF02001)和(底层)一个m盒核糖开关(RF00380)
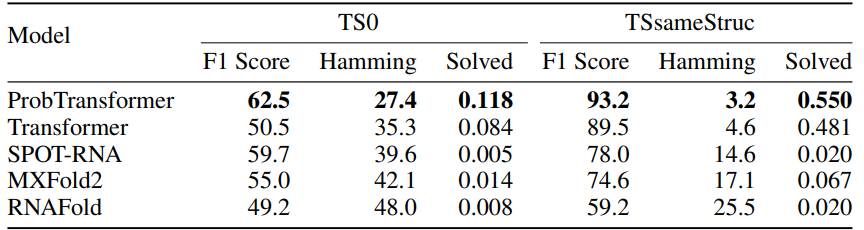
表2:TS0和TSsameStruc上不同RNA折叠方式的结构保真度。对于ProbTransformer和vanilla Transformer,本文显示了三个随机种子的平均结果
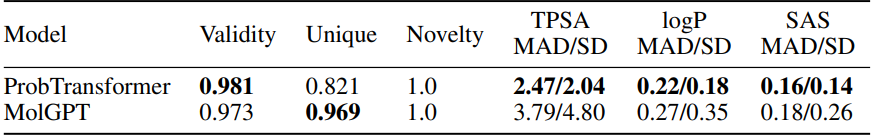
表3:GuacaMol数据集上的多属性(TPSA+logP+SAS)条件训练(在五种不同种子上的平均值)
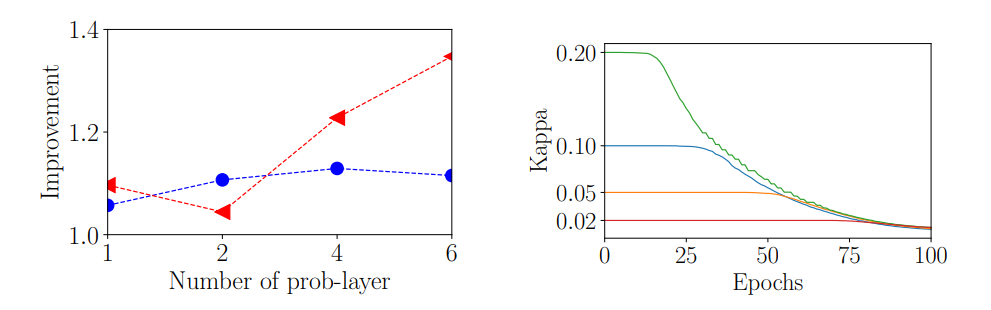
图4:(左)按问题层数量的性能改进:TS0上的圆点(蓝色)和TSsameStruc上的三角形(红色)。(右)100个训练周期中不同初始化的Kappa退火
表4:不同初始化条件下的汉明距离
结果与讨论
本文提出了一种新的概率层,在保持全局接受域的同时,利用分层潜在分布增强变压器架构。ProbTransformer在一个转发路径中对相互依赖的序列进行采样。这种采样发生在潜在空间中,ProbTransformer可以细化或解释在更深一层中采样的潜在表示。与从softmax输出分布中采样相比,该方法具有更大的灵活性。它还与Transformer模型的其他增强兼容,因为它只添加了一个新层,但保持其他所有内容不变。
ProbTransformer是第一个已知的RNA折叠模型,可以为给定的RNA序列提供多种正确的结构建议,这为RNA结构预测的新研究路径打开了大门,这些研究路径与RNA结构动力学的实验证据一致,例如,NMR研究,如磨损、凸起迁移和波动碱基对。在具有挑战性的多目标优化任务中,设计具有所需属性的分子,与最先进的vanilla Transformer架构相比,本文在仅解码的设置中演示了对分子属性的优越控制。
参考资料
Franke, Jörg, Frederic Runge, and Frank Hutter. "Probabilistic Transformer: Modelling Ambiguities and Distributions for RNA Folding and Molecule Design." Advances in Neural Information Processing Systems 35 (2022): 26856-26873.