共价对接(Covalent Docking)

- 随着人们对共价抑制剂的发现越来越重视,越来越多的软件开始支持共价抑制的虚拟筛选。
- 常用的共价对接软件有CovDock,AutoDock4,FITTED,GOLD,ICM-Pro与MOE等。
- 共价抑制剂的典型特征是配体的亲电部分(弹头)和蛋白受体上的亲核残基(主要是半胱氨酸)之间形成化学键。
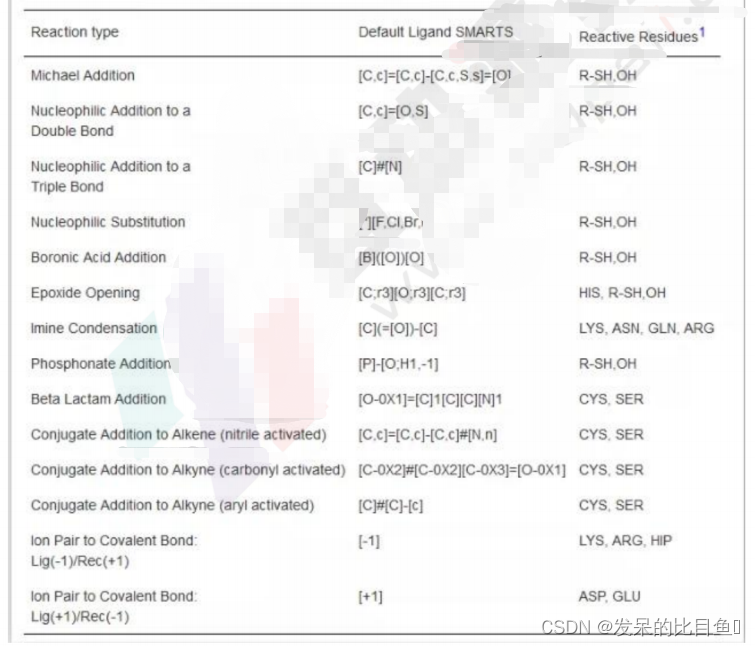
- 共价抑制剂的反应类型:迈克尔加成、亲核加成,亲核取代反应,开环反应,二硫化物的形成等。
准备
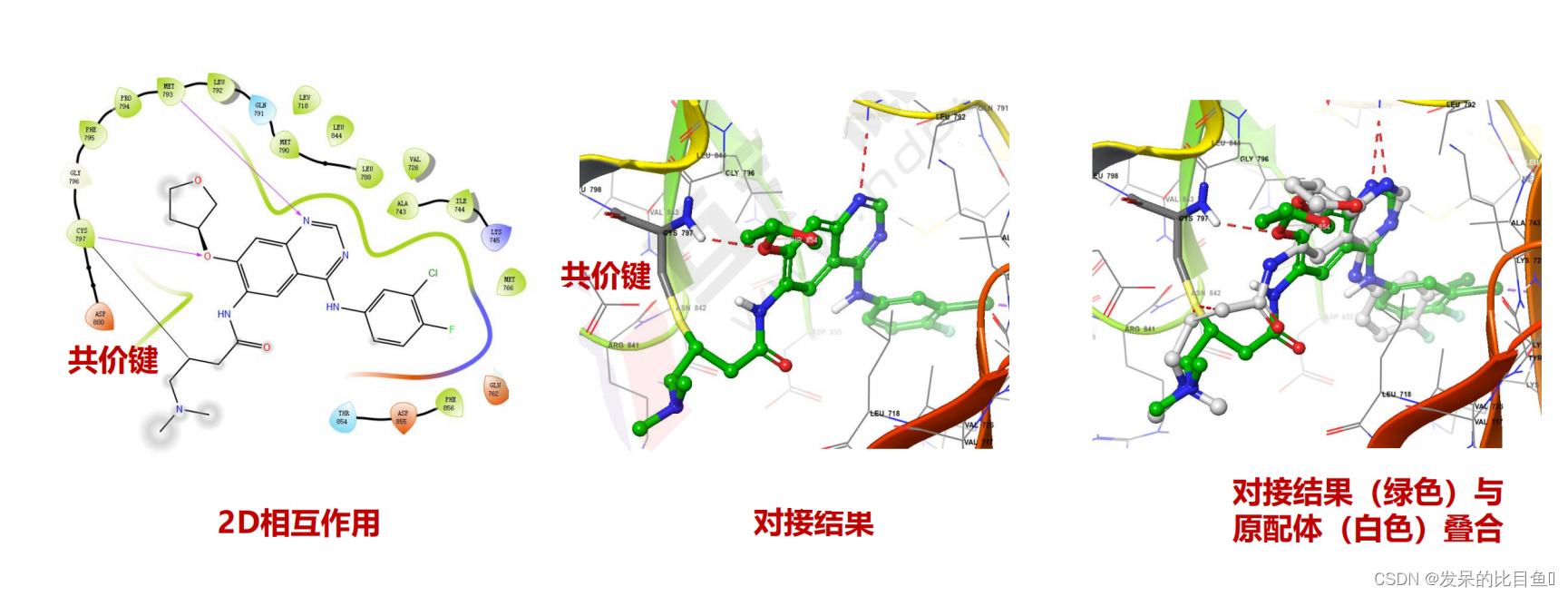
以EGFR靶点共价抑制剂Afatinib为例,展示共价对接,对应的PDB为:4G5P,可先下载备用!首先,从PDB数据库中下载复合物结构,导入至软件中,可发现Afatinib与Cys797形成共价键。
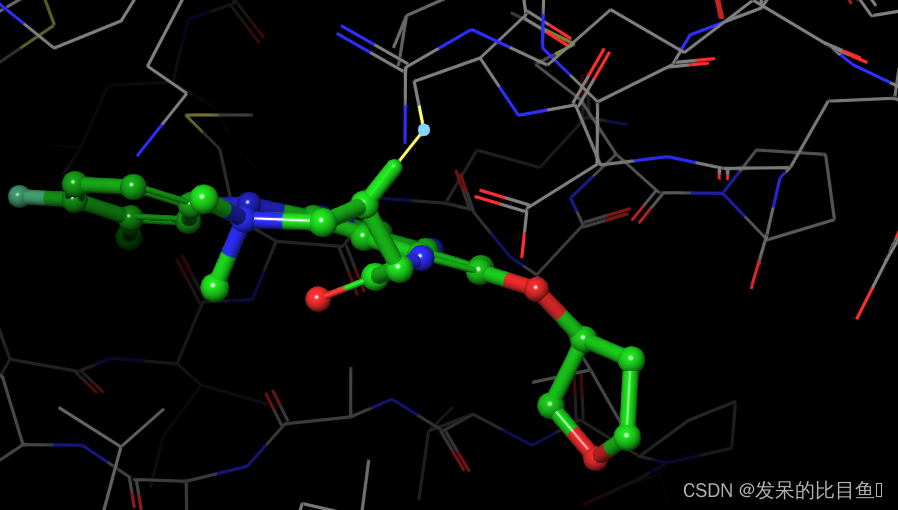
蛋白配体准备
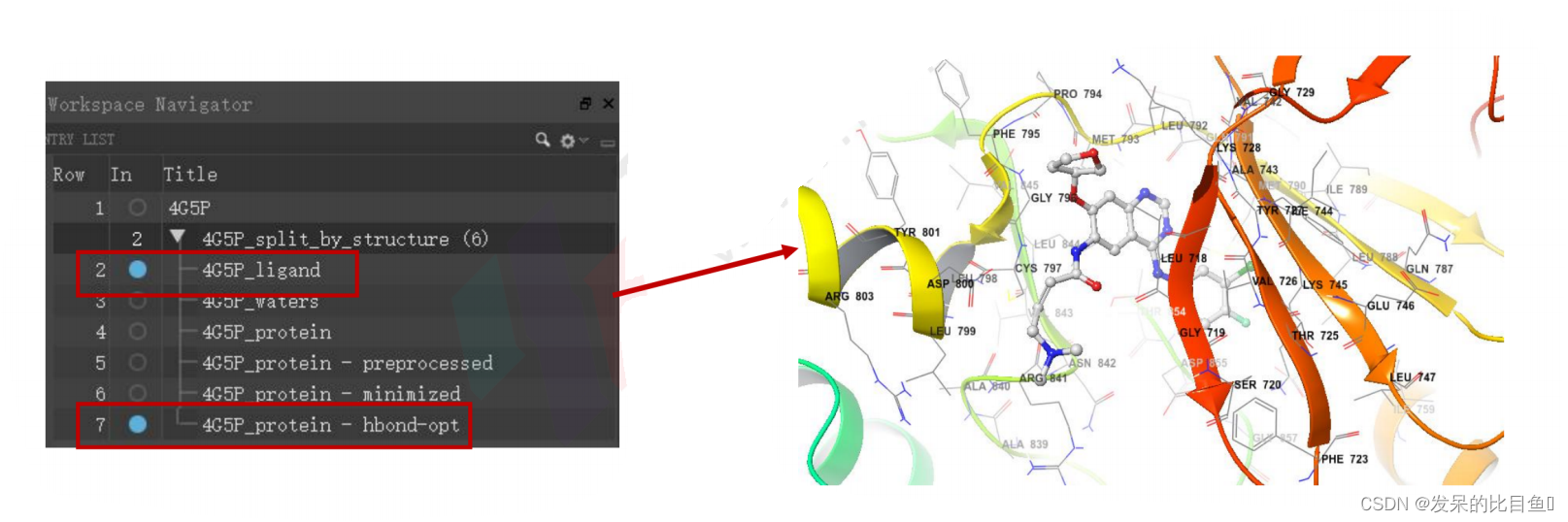
选中4G5P,右击选择Split--->Into Ligands,Water,Other

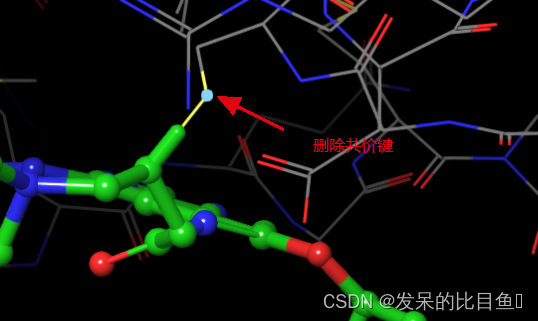
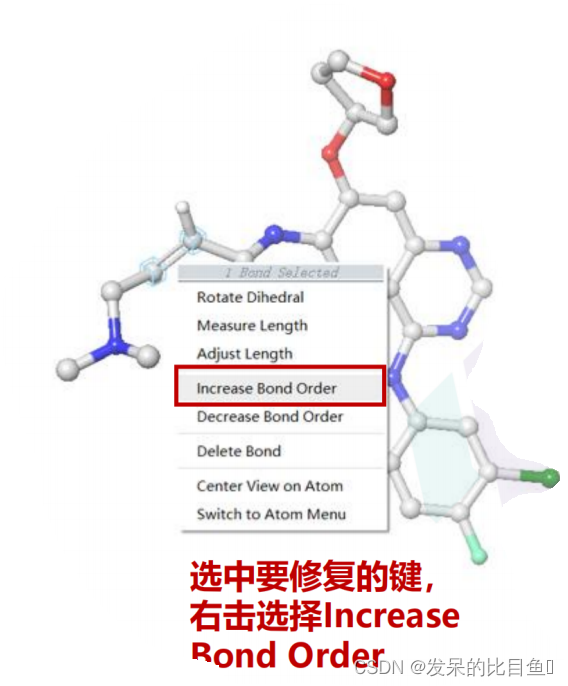
选中4G5P_ligand,进行结构修复
配体准备
用Ligpre对小配体进行优化准备,准备过程与glide对接一样,命名为ligprep_Afatinib
蛋白准备
选中4G5P_Protein,利用Protein preparation进行蛋白准备,准备过程可以参见glide蛋白准备部分。
仅保留A链
点击左侧分子目录,将4G5P_ligand和4G5P_protein-hbon-opt显示到窗口中
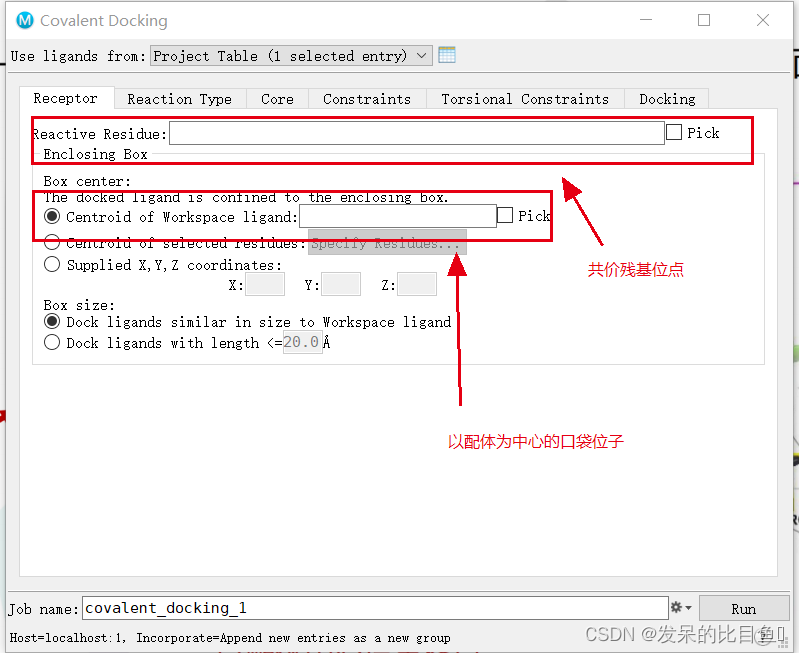
共价对接面板
点击Tasks—>输入covdock—>点击Covalent Docking,打开Covalent Docking面板
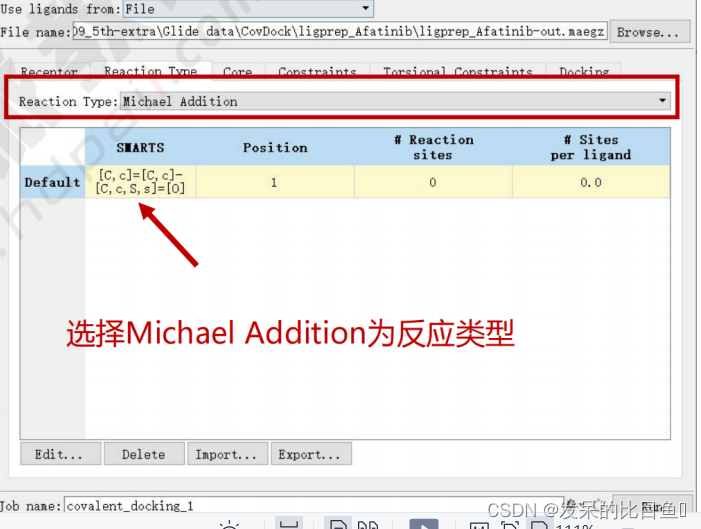
配体设置
Covalent Docking面板参数设置切换至Reaction Type选项卡;由于Afatinib是通过Michael受体丙烯酰胺与EGFR的CYS797共价结合;因此,反应类型Reaction Type的下拉菜单中选择Michael Addition即迈克尔加成反应;
小分子文件设置:Use ligands from:File,选择刚刚准备好的ligprep_Afatinib-out.maegz文件
如果是通过其他的方式反应形成共价键,可以选择其他的反应类型,具体可参考下表:
对接参数设置
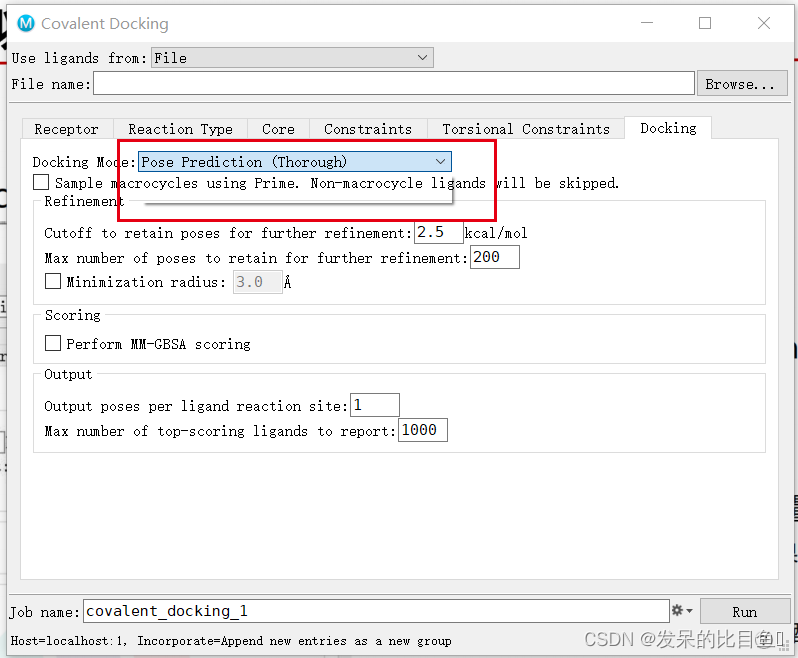
共价对接有两种模式:
- 构象预测
(Pose Prediction),执行完整的协议以进行准确的构象预测 - 虚拟筛选
(Virtual Screen),跳过费时的步骤,例如旋转取样和最小化,以筛选大量配体。- 虚拟筛选至少比构象预测快10倍。如果要从大量配体中找到可能的共价结合剂,可选择虚拟筛选
(Virtual Screen)。 - 配体数上限为10000,因此如果有更多配体,则必须分批运行。·如果要为几个配体找到合适的构象,可选择“构象预测”
(Pose Prediction)。 - 每个配体的构象预测可能需要1-2小时。
我们这里选择Pose Prediction,即在Docking Mode的选项下拉菜单中,选择Pose Prediction(Thorough)。
- 虚拟筛选至少比构象预测快10倍。如果要从大量配体中找到可能的共价结合剂,可选择虚拟筛选
对接结果
点击File--->Import Structures-->选择对接好的结果covalent docking_4G5P-Afatinib-VS-ok- out.maegz