本文摘自《Keras深度学习:入门、实战与进阶》。

1、数据理解
本节使用Pima Indians糖尿病发病情况数据集。该数据集最初来自国家糖尿病/消化/肾脏疾病研究所。数据集的目标是基于数据集中包含的某些诊断测量来诊断性的预测患者是否患有糖尿病。数据集由多个医学预测变量和一个目标变量Outcome。预测变量包括患者的怀孕次数、BMI、胰岛素水平、年龄等。数据集各变量描述如下:
Pregnancies:怀孕次数。
Glucose:葡萄糖。
BloodPressure:血压(mm Hg)。
SkinThickness:皮层厚度(mm)。
Insulin:胰岛素 2小时血清胰岛素(mu U / ml )。
BMI:体重指数(体重/身高)^2。
DiabetesPedigreeFunction:糖尿病谱系功能 。
Age:年龄(岁)。
Outcome:类标变量(0或1,糖尿病为1或非糖尿病为0)。
将数据集导入到R中,并查看数据结构。
> pima <- read.csv('../data/pima-indians-diabetes.csv',header = FALSE)
> str(pima)
'data.frame': 768 obs. of 9 variables:
$ V1: int 6 1 8 1 0 5 3 10 2 8 ...
$ V2: int 148 85 183 89 137 116 78 115 197 125 ...
$ V3: int 72 66 64 66 40 74 50 0 70 96 ...
$ V4: int 35 29 0 23 35 0 32 0 45 0 ...
$ V5: int 0 0 0 94 168 0 88 0 543 0 ...
$ V6: num 33.6 26.6 23.3 28.1 43.1 25.6 31 35.3 30.5 0 ...
$ V7: num 0.627 0.351 0.672 0.167 2.288 ...
$ V8: int 50 31 32 21 33 30 26 29 53 54 ...
$ V9: int 1 0 1 0 1 0 1 0 1 1 ...
数据集共有768行9列,由于所有变量都是数值的,因此可以直接作为神经网络的输入和输出使用。对数据集进行描述统计分析的结果所示。
> # 添加列名称
> column_names <- c('Pregnancies','Glucose','BloodPressure','SkinThickness',
+ 'Insulin','BMI','DiabetesPedigreeFunction','Age','Outcome')
> colnames(pima) <- column_names
> # 对数据进行描述统计分析
> library(skimr)
> skimmed <- skim(pima)
> skimmed
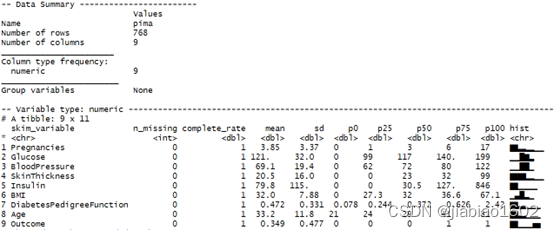
从上图可知,pima无数据缺失,前8个变量的尺度(数据范围)不一致,需要在建模前对输入变量进行标准化处理,输出变量Outcome中类别为1的占比为34.9%,属于相对平衡,在建模前不需要进行类失衡处理。
利用cor()函数计算Outcome与其他变量的相关系数,并对结果进行可视化,如下图所示。
> # 计算相关系数
> corr <- cor(pima[,-9],pima$Outcome)
> corr <- data.frame('X' = rownames(corr),
+ 'Outcome' = round(corr,3))
> # 对相关系数绘制柱状图
> library(ggplot2)
> ggplot(corr,aes(x=reorder(X,Outcome),y=Outcome,fill = I('tomato4'))) +
+ geom_bar(stat="identity") +
+ geom_text(aes(label=Outcome),vjust=1,color="white",size = 5,fontface = "bold")
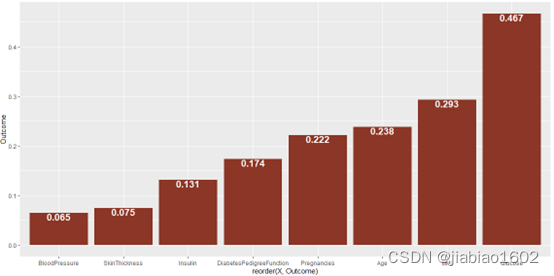
Outcome(是否患糖尿病)与Glucose(葡糖糖)的相关性最高,相关系数为0.467;其次是BMI(体重指数),相关系数为0.293。
2、印第安人糖尿病数据预处理
在对数据进行标准化前,先将数据分为两部分,80%作为训练集,剩余的20%作为测试集。
> # 数据分区
> set.seed(1234)
> library(caret)
> index <- createDataPartition(pima$Outcome,p = 0.8,list = FALSE)
> train <- pima[index,] # 训练集
> test <- pima[-index,] # 测试集
> train_x <- as.matrix(train[,-9]) # 训练集的输入变量
> train_y <- as.matrix(train[,9]) # 训练集的输出变量
> test_x <- as.matrix(test[,-9]) # 测试集的输入变量
> test_y <- as.matrix(test[,9]) # 测试集的输出变量
从描述统计分析结果可知,输入变量中的各列数据范围差异比较大。在建模前,需先对数据集进行标准化处理。此案例使用scale()函数进行Z-Score标准化,处理后的训练集输入变量各列数据符合标准正态分布,即均值为0,标准差为1。
> # 对训练集的输入变量进行标准化处理
> train_x_scale <- scale(train_x)
接着使用从训练集标准化后得到的各列均值和标准差对测试集数据进行数据处理。
> col_means_train <- attr(train_x_scale, "scaled:center")
> col_stddevs_train <- attr(train_x_scale, "scaled:scale")
> test_x_scale <- scale(test_x,
+ center = col_means_train,
+ scale = col_stddevs_train)
3、印第安人糖尿病诊断建模
在这个例子中,使用三层完全连接的网络结构,使用ReLU作为前两层的激活函数,使用sigmoid作为输出层的激活函数。第一个隐藏层有12个神经元,使用8个输入变量;第二个隐藏层由8个神经元,最后输出层有1个神经元来预测数据结果(是否患有糖尿病)。
在模型编译时,本例使用对数损失函数(二进制交叉熵)作为模型的损失函数,使用Adam作为优化器。由于这是一个分类问题,本例将采用分类准确度作为度量模型的标准。代码如下:
library(keras)
> # 构建模型函数
> build_model <- function() {
+
+ model <- keras_model_sequential() %>%
+ layer_dense(units = 12, activation = "relu",
+ input_shape = c(8)) %>%
+ layer_dense(units = 8, activation = "relu") %>%
+ layer_dense(units = 1,activation = 'sigmoid')
+
+ model %>% compile(
+ loss = "binary_crossentropy",
+ optimizer = 'adam',
+ metrics = "accuracy")
+ model
+ }
在这个示例中,epochs参数为200,batch_size参数为10,且我们设置一个回调函数,如果经过20次训练周期后验证集的损失函数没有明显改善,将自动停止训练。
> # 设置回调函数的停止条件
> early_stop <- callback_early_stopping(monitor = "val_loss",
+ patience = 20,
+ restore_best_weights = TRUE)
> # 训练模型
> mlp_model <- build_model()
> history <- mlp_model %>% fit(
+ train_x_scale,
+ train_y,
+ epochs = 150,
+ batch_size = 10,
+ validation_split = 0.2,
+ verbose = 2,
+ callbacks = early_stop
+ )
Train on 492 samples, validate on 123 samples
Epoch 1/150
492/492 - 3s - loss: 0.6586 - accuracy: 0.6199 - val_loss: 0.6220 - val_accuracy: 0.6098
……
Epoch 51/150
492/492 - 1s - loss: 0.4017 - accuracy: 0.8211 - val_loss: 0.4330 - val_accuracy: 0.8130
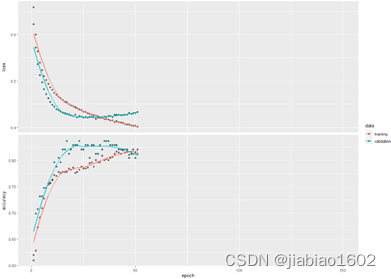
在经过51个训练周期后,模型停止了训练。因为回调函数参数restore_best_weights设置为TRUE, 则将会获得val_loss最小(epochs=31)的模型权重值。
> # 查看val_loss最小值的周期
> which.min(history$metrics$val_loss)
[1] 31
> # 查看val_loss最小值
> min(history$metrics$val_loss)
[1] 0.4188787
最后,利用训练好的模型对测试样本进行预测,并查看混淆矩阵。
> outcome.pred <- mlp_model %>%
+ predict_classes(test_x_scale)
> (t <- table('actual' = test_y,
+ 'forecast' = outcome.pred))
forecast
actual 0 1
0 84 7
1 23 39
从混淆矩阵结果可知,在153个测试样本中,有7位非糖尿病患者被误预测为糖尿病患者,有23位糖尿病患者被误预测为非糖尿病患者。