论文标题:FP2VEC:a new molecular featurizer for learning molecular properties
代码: GitHub - wsjeon92/FP2VEC
预测化合物性质最成功的方法之一是定量结构-活性关系(QSAR)方法。
Mol2vec使用分子子结构表将分子结构表示为类似于分子指纹向量的向量表示。SMILES2VEC模型引入了从SMILES表示到embedding向量的直接转换。
引入了一种新的分子特征FP2VEC,它将化合物表示为一组可训练的embedding向量。这项工作的动机是化合物和自然语言之间存在明显的类比
一、模型方法
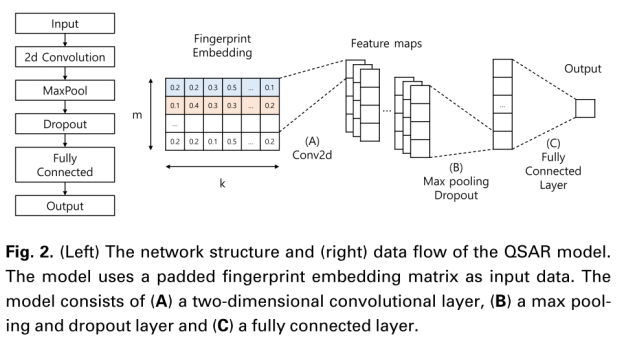
使用CNN架构构建了一个QSAR模型。采用了一个简单的CNN架构,该架构已经成功地用于NLP分类任务,如句子分类
1、Benchmark featurizers and datasets:
MoleculeNet(Tox21, HIV, BBBP and SIDER + Malaria, CEP, ESOL, FreeSolv and Lipophilicity)
2、Featurizer and QSAR model
Fingerprint embedding featurizer
一种化合物可以表示为一组分子子结构(分子指纹),每个子结构表示为一个向量(指纹嵌入向量),假设一个化合物可以由一组指纹embedding向量表示,就像文本可以由一组word embedding向量表示一样。
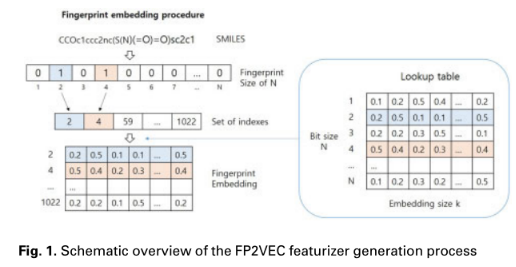
首先从化合物的smiles表示中提取分子的子结构,即 使用RDKit生成半径为2的1024位Morgan(或圆形)指纹。已经尝试了2048位或全尺寸(“展开”)指纹,但发现指纹向量的大小不影响模型的性能。之后,我们收集指纹索引,在指纹向量中标记为“1”。然后将分子结构的特征表示为一个整数列表,其中每个整数代表一个特定的分子子结构。这些整数类似于文本的单词索引。
接下来构建查找表(Lookup table),将每个整数索引表示为有限大小的向量(嵌入大小)。查找表是一个二维矩阵,其大小是bit size乘以embedding size。查找表的每一行都提供一个唯一的嵌入向量,对应于Morgan指纹的每个整数。在初始状态下,查找表使用随机值初始化。通过训练过程,对查找表的值进行微调,以最大限度地实现训练的特定目标。
通过这个过程,得到了特定任务的化合物向量表示,称为指纹embedding矩阵。这个特定于任务的查找表可以提供比传统圆形指纹本身更有用的信息。
Structure of the QSAR model using a simple CNN architecture
Multi-task learning
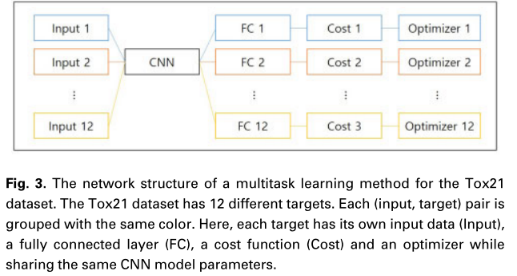
在Tox21和SIDER数据集中,一个化合物与多个目标相关联。在这种情况下,多任务学习模型比单任务学习模型显示出更好的预测效果。在单任务学习的情况下,每个目标都有单独的CNN模型。例如,对于tox21数据集,针对12个不同的目标有12个单独的CNN模型。因此,每个CNN模型都由不同的输入数据训练。然而,对于多任务学习,所有12个目标只有一个CNN模型(图3)。在多任务学习方案中,12个不同的目标共享CNN模型体系结构的参数。通过共享参数,CNN模型可以捕获目标化合物的一般特征。
每个目标的分离的完全连接层然后学习每个目标化合物的特定特征。同时使用单一CNN模型和分离的全连接层,多任务学习模型可以提高预测精度。
二、Results and discussion
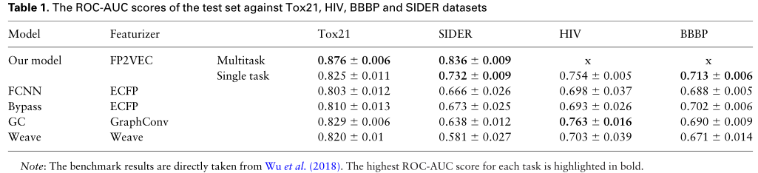
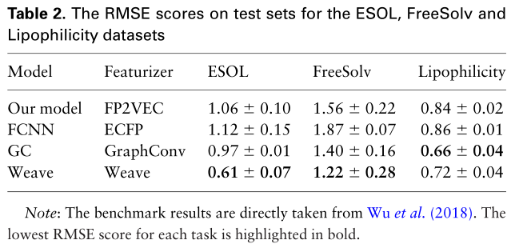
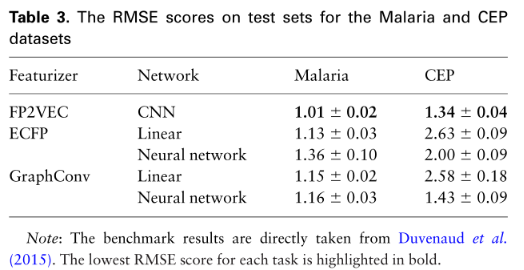
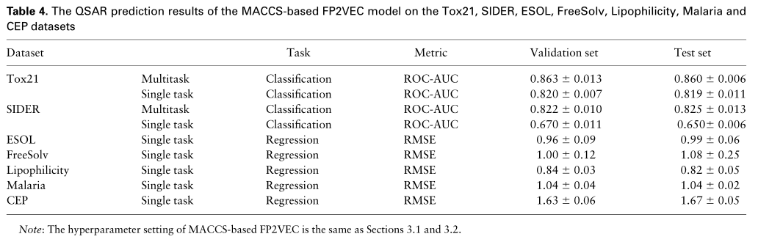
特别注意的是(证明了一点:分子指纹不适合单独作为分子性质预测的输入):
圆形指纹模型的预测结果比FP2VEC的预测结果差很多。圆形指纹向量稀疏;因此,小窗口的卷积滤波器不能适当地捕捉分子特征。当窗口大小为11时,预测精度略有提高(ROC-AUC评分为0.624)。大于11的窗口大小没有显著差异。这些结果清楚地表明,与原始圆形指纹相比,FP2VEC特征器提高了QSAR任务中的预测性能。
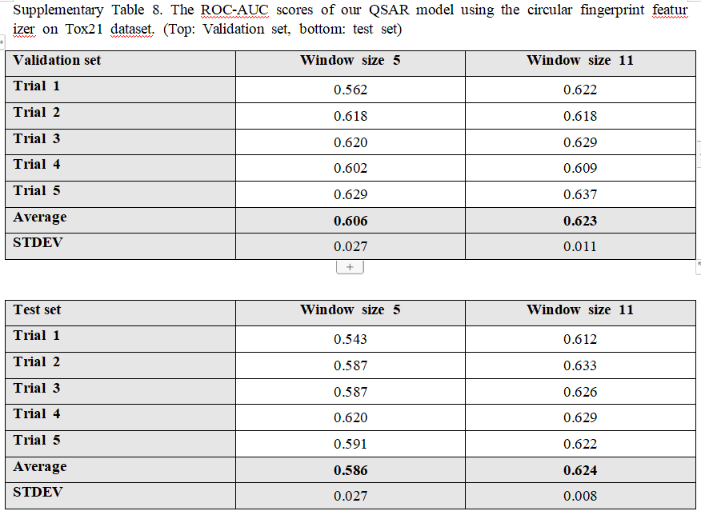
很新颖,把分子指纹的index映射为一个learnable table。虽然是19年的论文,思想、网络也很简单,但是工作扎实。



![[附源码]java毕业设计疫情背景下社区公共卫生服务系统](https://img-blog.csdnimg.cn/15054854ad2540da850cb2a800f4eebb.png)