方差分析这部分内容还不是很理解,在这里先做一个笔记,以后有时间再回过头来改一改。
用到的数据集 → \rightarrow →Iris
什么是假设检验?
假设检验就是利用样本数据对某个事先做出的统计假设,再按照某种方法去检验,最后判断此假设是否正确。
怎么去假设检验?
假设检验的目的是为了推断总体。首先对总体的未知参数或分布做出某种假设
H
0
H_{0}
H0,然后在
H
0
H_0
H0成立的条件下,若通过抽样分析发现“小概率事件”竟然在一次实验中发生了(在这里我们是不希望小概率事件发生的),则表明
H
0
H_0
H0很可能不成立,从而拒绝
H
0
H_0
H0;相反,若这个“小概率事件”没有发生,则没有理由拒绝
H
0
H_0
H0,从而接收
H
0
H_0
H0。
⭐️ 检验的显著性水平(
α
\alpha
α):要求“小概率事件”发生的概率小于等于某一给定的临界概率。通常
α
\alpha
α的取值为较小的数0.05、0.01、0.001。
⭐️ P值:假定原假设
H
0
H_0
H0为真时,“拒绝原假设
H
0
H_0
H0”这件事犯错的可能性。当P值
<
α
<\alpha
<α时,表示“拒绝原假设
H
0
H_0
H0”这件事犯错误的可能性很小,即可以认为原假设
H
0
H_0
H0是错误的,从而拒绝
H
0
H_0
H0;否则,应接收原假设
H
0
H_0
H0。
🌕 数据分布检验
数据分布的假设检验是重要的非参数检验,它不是针对具体的参数,而是根据样本值来判断总体是否服从某种指定的分布。
🌗 数据准备
# 导入相关的库
import numpy as np
from matplotlib import pyplot as plt
from scipy import stats
# 中文显示问题
import matplotlib
matplotlib.rcParams['axes.unicode_minus']=False
import seaborn as sns
sns.set(font="Kaiti",style="ticks",font_scale=1.4)
np.random.seed(123) # 设置随机数种子
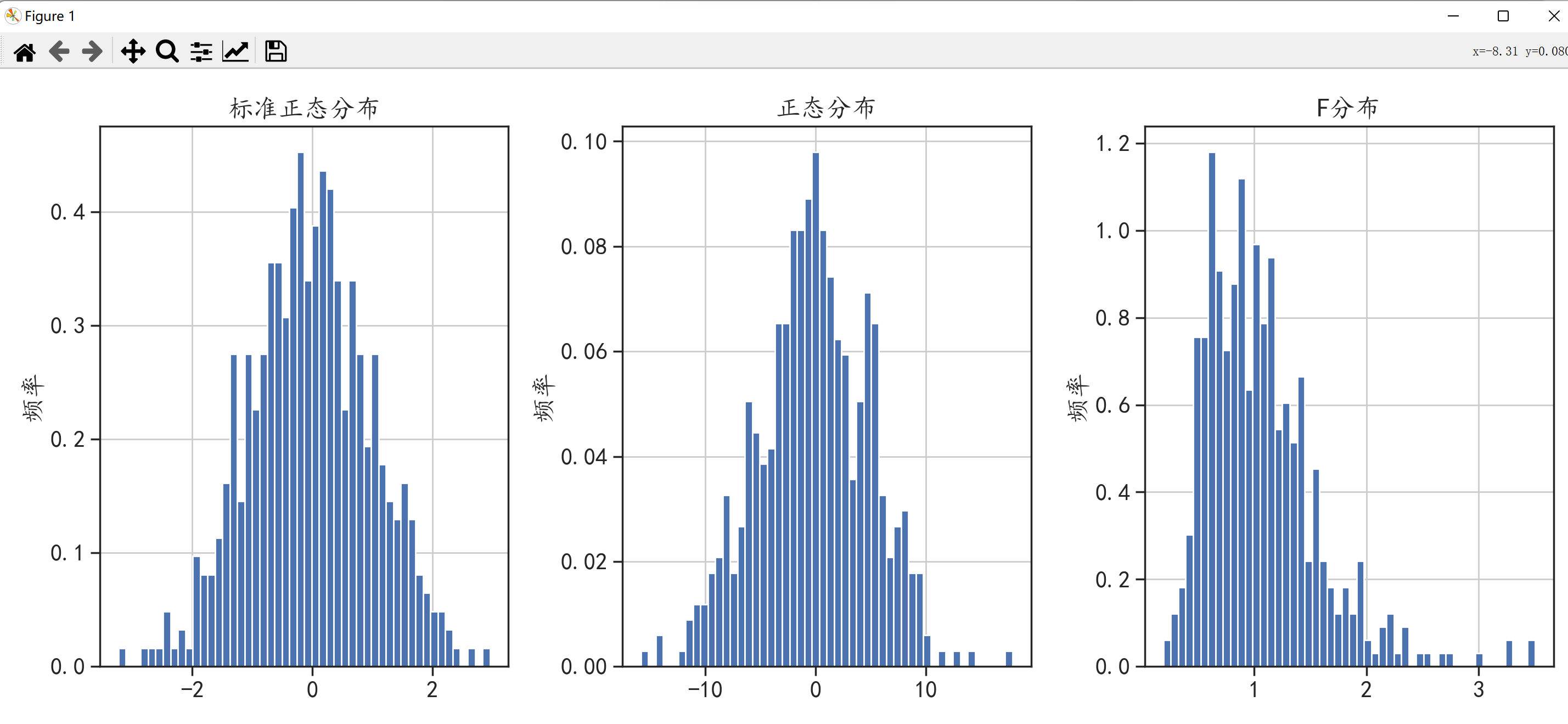
X1 = stats.norm.rvs(loc = 0,scale = 1,size = 500) # X1为服从均值为0方差为1的标准正态分布数据
X2 = stats.norm.rvs(loc = 0,scale = 5,size = 500) # X2为服从均值为0方差为5的正态分布数据
X3 = stats.f.rvs(15,30,size = 500) # X3为服从均值为15方差为30的非正态分布数据
plt.figure(figsize = (15,6))
plt.subplot(1,3,1) # 将画布分为一行三列(三部分),现在对从左至右从上至下第一个子图操作
plt.hist(X1,bins = 50,density = True) # X1的频率直方图(density=True表示频率,False表示频数。默认为频数)
plt.grid()
plt.ylabel("频率")
plt.title("标准正态分布")
plt.subplot(1,3,2) # 对第二个子图操作
plt.hist(X2,bins = 50,density = True)
plt.grid()
plt.ylabel("频率")
plt.title("正态分布")
plt.subplot(1,3,3) # 对第三个子图操作
plt.hist(X3,bins = 50,density = True)
plt.grid()
plt.ylabel("频率")
plt.title("F分布")
plt.tight_layout() # 防止子图间轴域的标签叠在一起
plt.show()
🌗 利用Q-Q图检验数据是否符合正态分布
什么是Q-Q图?
Q-Q中的两个Q都是quantile(分位数)的缩写,也就是说Q-Q是横纵坐标都关于分位数的一个图。那么说到这里,
什么又是分位数?
分位数也称分位点,是指将一个随机变量的概率分布范围分为几个等份的点,常用的有中位数(二分位数)、四分位数、百分位数等。
Q-Q图的作用即标题,是用来检验数据是否符合正态分布的,它就是先将两列数据的分位点画成散点图,然后比较两列数据的分位点是否分布在y=x的直线上,若是,则接收数据来自正态总体的假设,否则就拒绝原假设。
在这里我们可以使用statsmodels.api模块中的qqplot()函数来绘制Q-Q图。
import numpy as np
from matplotlib import pyplot as plt
from scipy import stats
import statsmodels.api as sm # 导入Q-Q图要用的模块
import matplotlib
matplotlib.rcParams['axes.unicode_minus']=False
import seaborn as sns
sns.set(font="Kaiti",style="ticks",font_scale=1.4)
np.random.seed(123)
X1 = stats.norm.rvs(loc = 0,scale = 1,size = 500)
X2 = stats.norm.rvs(loc = 0,scale = 5,size = 500)
X3 = stats.f.rvs(15,30,size = 500)
fig = plt.figure(figsize = (15,5))
ax = fig.add_subplot(1,3,1) # add_subplt与subplot是一个东西,没有太大的区别,只是调用的方式不一样
sm.qqplot(X1,line = "45",ax = ax) # 绘制X1的Q-Q图,line=45是绘制45°斜直线
plt.grid()
plt.title("X1正态检验Q-Q图")
ax = fig.add_subplot(1,3,2)
sm.qqplot(X2,loc = 0,scale = 5,line = "45",ax = ax) # 绘制X2的Q-Q图
plt.grid()
plt.title("X2正态检验Q-Q图")
ax = fig.add_subplot(1,3,3)
sm.qqplot(X3,line = "45",ax = ax) # 绘制X3的Q-Q图
plt.grid()
plt.title("X3正态检验Q-Q图")
fig.tight_layout()
plt.show()
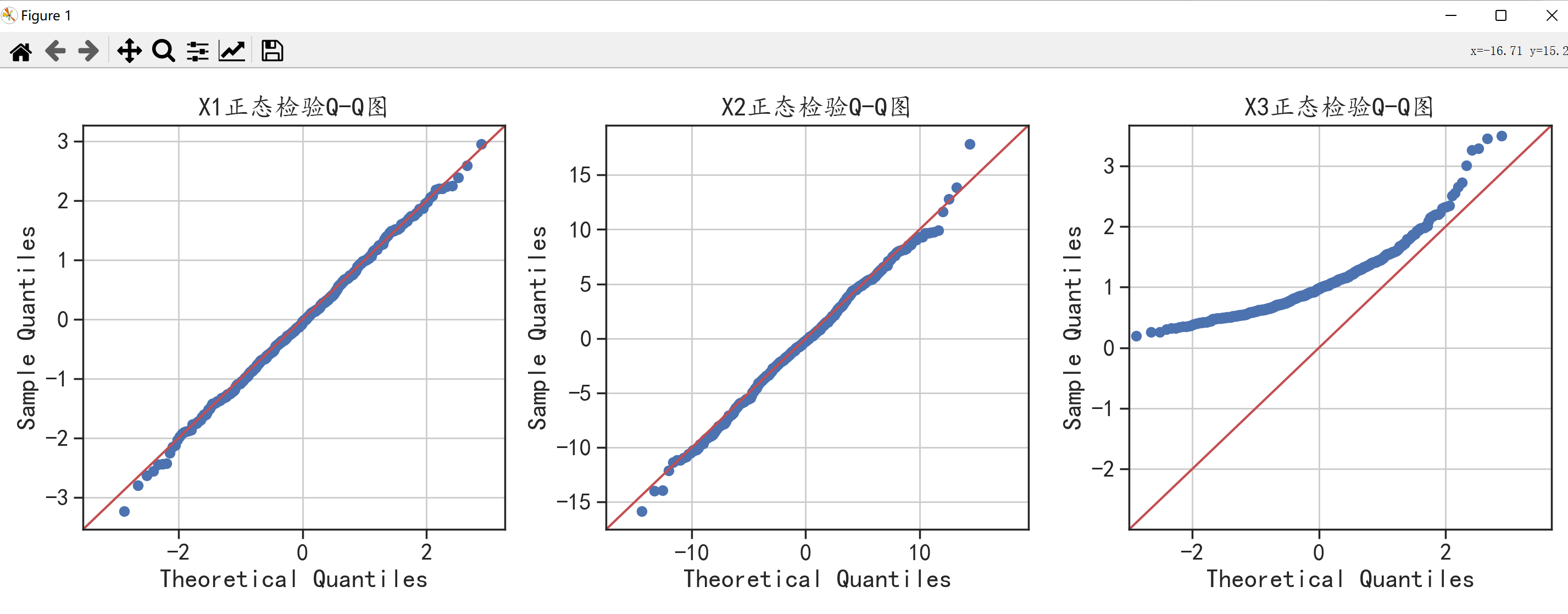
可以发现,前两个图的散点图都能很好地拟合参考线,表示X1和X2是正态分布,而第三个图的散点图没有在参考线上,表示X3不是正态分布。
🌗 利用K-S拟合优度检验来检验数据是否符合正态分布
K-S检验可以使用stats.kstest()函数完成,默认情况下会验证数据是否符合标准正态分布,同时可以指定cdf参数所要验证的分布类型,还可以利用args参数指定符合参数的特定分布。
# 这里还是用随机数种子“123”
print(stats.kstest(X1,cdf = "norm")) # X1的标准正态分布检验
print(stats.kstest(X2,cdf = "norm")) # X2的标准正态分布检验
print(stats.kstest(X2,cdf = "norm",args = (0,5))) # X2的正态分布检验
print(stats.kstest(X3,cdf = "norm")) # X3的标准正态分布检验
print(stats.kstest(X3,cdf = "f",args = (15,30))) # X3的非正态分布检验
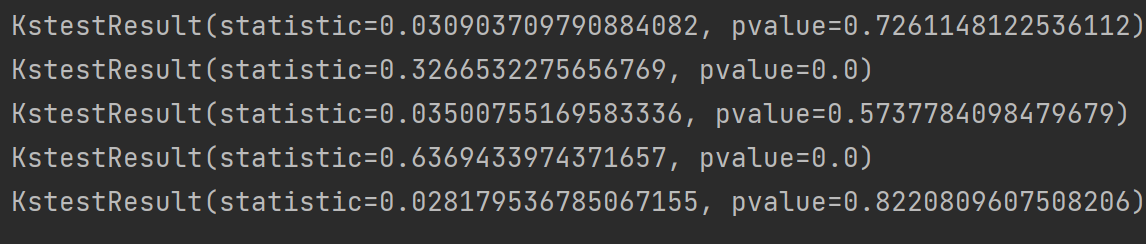
从结果中我们可以看到,在对X1的标准正态分布检验中,P值大于0.05(在前面回顾一下P值的概念),就说明了“拒绝原假设”这件事犯错误的可能性比较大了,也就是说拒绝原假设会犯错误,那么我们就应该接收原假设,即X1是服从标准正态分布的。(0.05是检验的显著性水平,是人为取值的)
又如在X2的标准正态分布检验中,它的P值为0,但本质上是不为0的,因为这个P值很小很小(5.2306133517677304 × 1 0 − 48 \times10^{-48} ×10−48),所以程序就直接省略了。那就是这个P值它是小于0.05,说明“拒绝原假设”这件事犯错误的可能性比较小,那么我们就应该拒绝原假设,即X2不服从标准正态分布。
后面几个结果也是同样的分析。
K-S检验还可以用stats.ks_2samp()函数检验两个随机变量的分布是否相同
print(stats.ks_2samp(X1,X2))
print(stats.ks_2samp(X2,X3))
print(stats.ks_2samp(X3,X1))
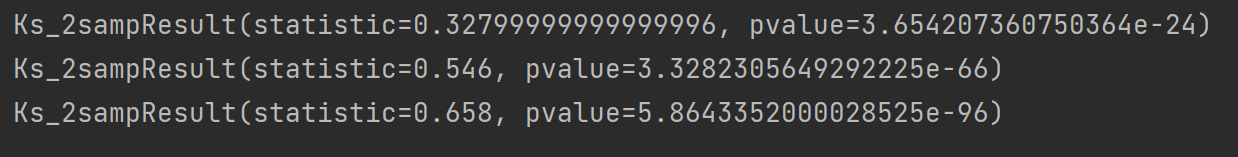
同样,也是根据P值来判断它们是否具有相同的分布。
🌕 t检验
t检验分为单样本t检验和两独立样本t检验。
⭐️ 单样本t检验:检验来自正态分布的样本的期望值(均值)是否为某一实数。
⭐️ 两独立样本t检验:判断两个来自正态分布(方差相同)的独立样本的期望值之差是否为某一实数。
两种检验的原假设都是差等于指定值。
🌗 单样本t检验
import numpy as np
from scipy import stats
np.random.seed(123)
X1 = stats.norm.rvs(loc = 0,scale = 1,size = 500) # 标准正态分布随机数X1
X2 = stats.norm.rvs(loc = 0,scale = 5,size = 500) # 正态分布随机数X2
X3 = stats.norm.rvs(loc = 5,scale = 5,size = 500) # 正态分布随机数X3
print(stats.ttest_1samp(X1,0)) # 检验X1的均值是否为0
print(stats.ttest_1samp(X2,5)) # 检验X2的均值是否为5
print(stats.ttest_1samp(X3,5)) # 检验X3的均值是否为5
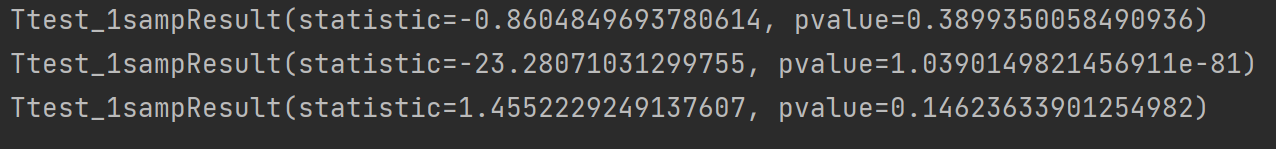
从第一组的检验结果可以发现,它的P值大于0.05,也就是说“拒绝原假设”这件事犯错误的可能性比较大,所以应该接受原假设,即X1的均值为0,剩下两组的判断也是如此。
🌗 两独立样本的检验
print(stats.ttest_ind(X1,X2)) # 检验X1和X2的均值之差是否等于0
print(stats.ttest_ind(X2,X3)) # 检验X2和X3的均值是否相等
print(stats.ttest_ind(X3,X1)) # 检验X3和X1的均值是否相等
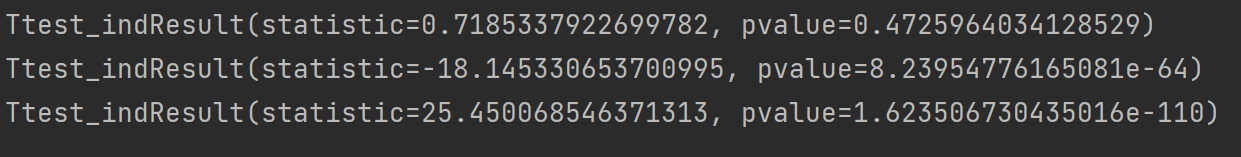
从结果分析,我们可以知道X1和X2的均值相等,X2和X3的均值不相等,X3和X1的均值也不相等。
🌕 方差分析
方差分析是分析实验数据的一种方法。
对于抽样测得的实验数据,由于观测条件不同或随机因素的干扰造成的差异使得实验结果不同,将前者造成的差异称为系统差异和偶然差异。
方差分析的目的:从实验数据中分析出各个因素及它们之间产生的影响,确定各个因素作用的大小,进而把两种差异区分开来,以确定在实验中有没有系统的因素在起作用。
方差分析根据因素数量,可分为单因素方差分析、双因素方差分析等。
🌗 单因素方差分析
使用sm.stats.anova_lm()函数完成方差分析(l是小写L),使用pairwise_tukeyhsd()函数对数据方差分析结果进行多重检验。
import pandas as pd
import statsmodels.api as sm
import statsmodels.formula.api as smf
a = pd.read_csv("D:/Pycharm/机器学习数据/program/data/chap2/Iris.csv") # 读取数据
model = smf.ols("SepalLengthCm~Species",data = a).fit()
b = sm.stats.anova_lm(model,typ = 2) # 方差分析
print(b)
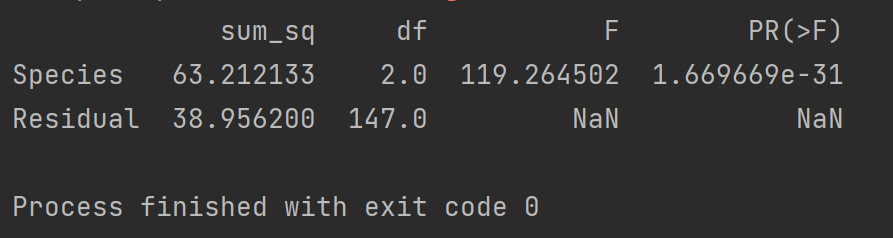 最后一个PR是P值,远小于0.05,则应该拒绝原假设,说明不用类别的SepalLengthCm特征均值不完全相同。那么我们可以使用多重比较对比哪些类别之间的均值不同。
最后一个PR是P值,远小于0.05,则应该拒绝原假设,说明不用类别的SepalLengthCm特征均值不完全相同。那么我们可以使用多重比较对比哪些类别之间的均值不同。
import pandas as pd
from statsmodels.stats.multicomp import pairwise_tukeyhsd
a = pd.read_csv("D:/Pycharm/机器学习数据/program/data/chap2/Iris.csv")
b = pairwise_tukeyhsd(endog = a.SepalLengthCm,groups = a.Species,alpha = 0.05)
print(b)
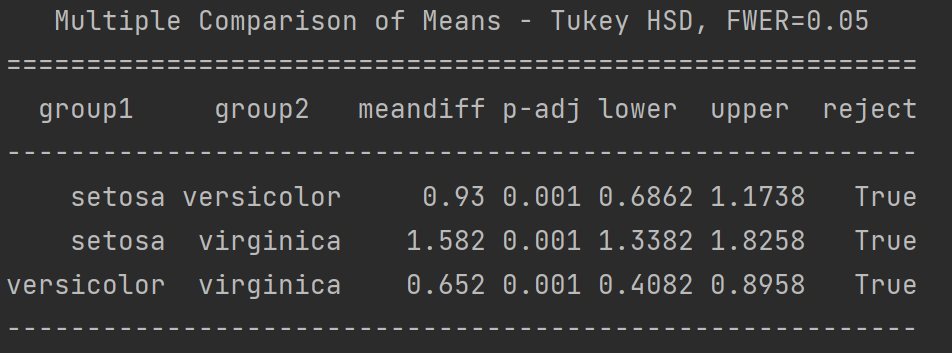
针对于获得的多重比较的结果,可以使用plot_simultaneous()函数可视化出多重比较的图像。
b.plot_simultaneous()
plt.grid()
plt.show()
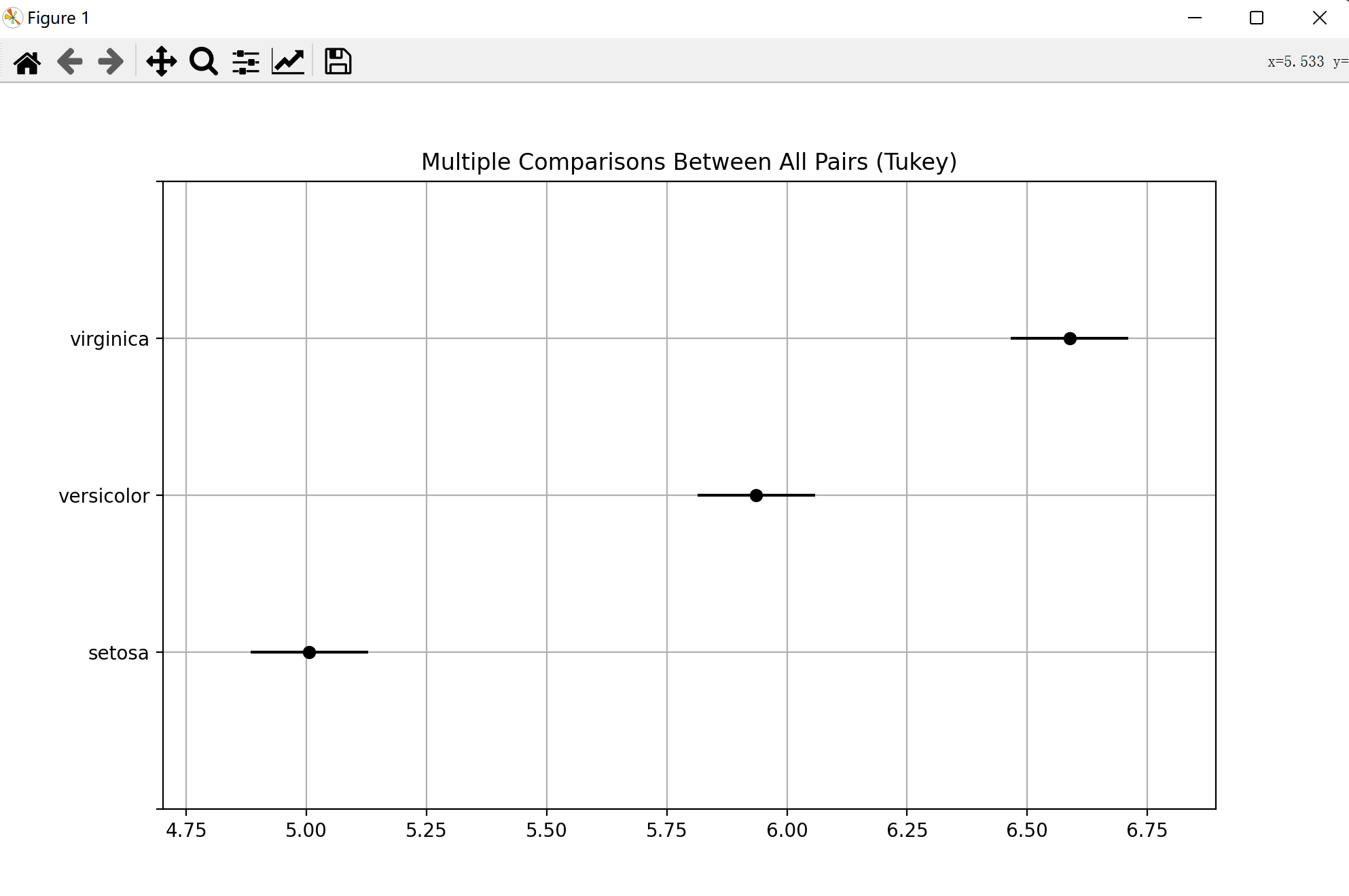
可以直观的看出,三个类别的均值都不相同或,且setosa的均值最小,virginica的均值最大。
🌗 双因素方差分析
双因素方差分析分为两种情况,分别是不考虑交互作用和考虑交互作用的情况。
import numpy as np
import pandas as pd
import statsmodels.api as sm
import statsmodels.formula.api as smf
a = pd.read_csv("D:/Pycharm/机器学习数据/program/data/chap2/Iris.csv")
np.random.seed(123)
a["Group"] = np.random.choice(["A","B"],size = 150) # 生成新的分类因素Group
model = smf.ols("SepalLengthCm~Species * Group",data = a).fit()
b = sm.stats.anova_lm(model,typ = 2)
print(b)

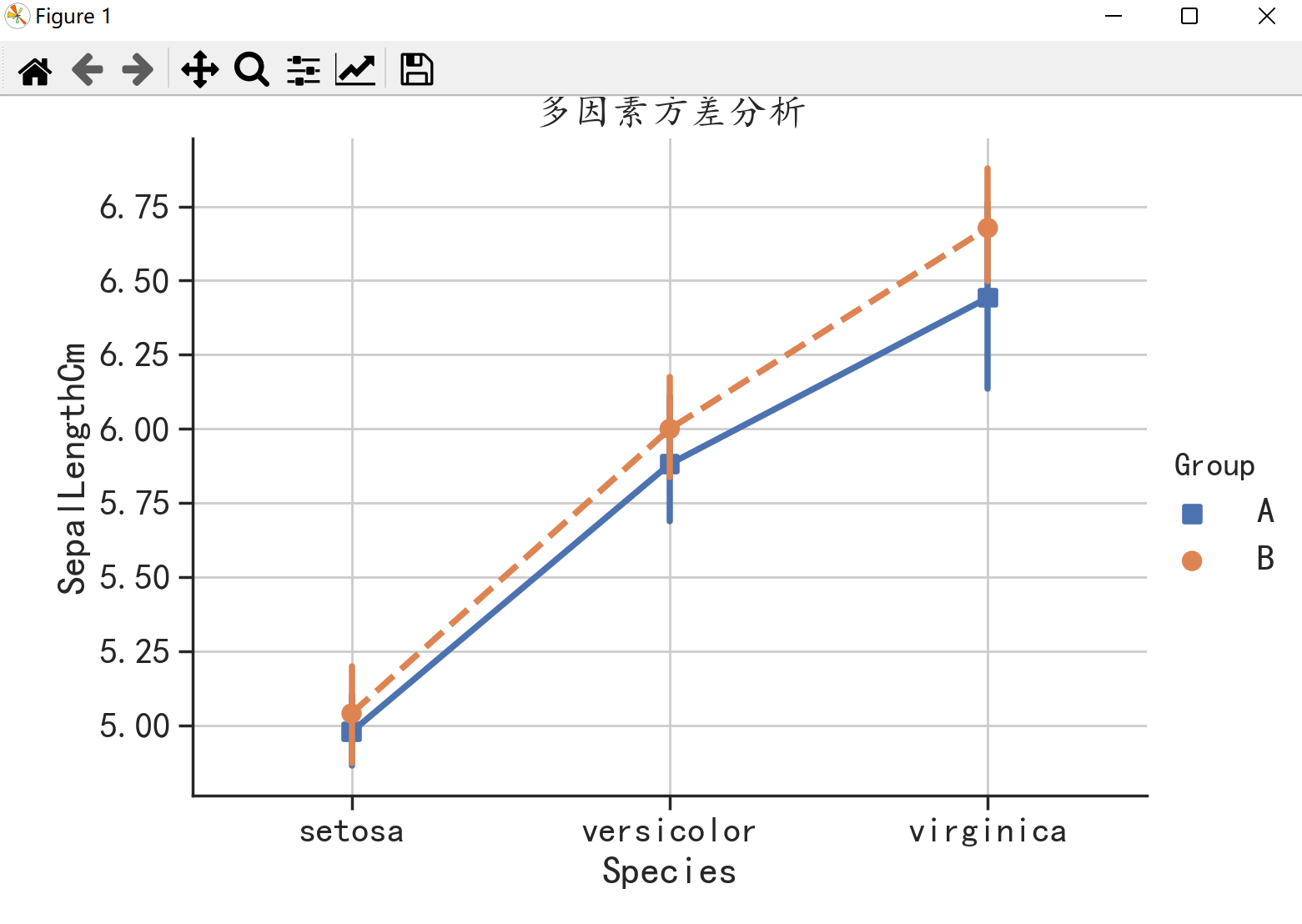
从结果可以看出,Species因素下差异是显著的。Group因素和Species:Group(Species和Group的交互作用)因素下差异是不显著的。
针对双因素影响的数据,可以使用sns.catplot()函数将其可视化。
import numpy as np
import pandas as pd
import statsmodels.api as sm
import statsmodels.formula.api as smf
from matplotlib import pyplot as plt
import seaborn as sns
import matplotlib
matplotlib.rcParams['axes.unicode_minus']=False
sns.set(font="Kaiti",style="ticks",font_scale=1.4)
a = pd.read_csv("D:/Pycharm/机器学习数据/program/data/chap2/Iris.csv")
np.random.seed(123)
a["Group"] = np.random.choice(["A","B"],size = 150)
model = smf.ols("SepalLengthCm~Species * Group",data = a).fit()
b = sm.stats.anova_lm(model,typ = 2)
sns.catplot(x = "Species",y = "SepalLengthCm",hue = "Group",markers = ["s","o"],linestyles = ["-","--"],data = a,kind = "point",aspect = 1.4)
plt.grid()
plt.title("多因素方差分析")
plt.show()