03、K-means聚类实现步骤与基于K-means聚类的图像压缩(1)
03、K-means聚类实现步骤与基于K-means聚类的图像压缩(1)
03、K-means聚类实现步骤与基于K-means聚类的图像压缩(2)
开始学习机器学习啦,已经把吴恩达的课全部刷完了,现在开始熟悉一下复现代码。对这个手写数字实部比较感兴趣,作为入门的素材非常合适。
K-means聚类实现步骤
1、K-means基础
K-means算法是一种常用的聚类算法,它的实现步骤如下:
STEP1:从数据集中随机选择k个样本作为初始聚类中心。
STEP2:计算每个样本到各聚类中心的距离,并将样本归入最近的聚类中心。
STEP3:重新计算每个聚类的中心,该中心为该类所有样本的平均值。
STEP4:重复步骤2和3,直到满足以下条件之一:
聚类中心不再变化。
达到预设的最大迭代次数。
最小平方误差SSE(误差的平方和)达到预设的阈值。
2、K-means的底层代码实现
STEP0:调用numpy和绘图库:
import numpy as np
from matplotlib import pyplot as plt
STEP1:从数据集中随机选择k个样本作为初始聚类中心:
# 随机初始化聚类初始优化点
def kMeans_init_centroids(X, K):
# 随机重新排序样本的索引
randidx = np.random.permutation(X.shape[0])
# 取前K个样本作为聚类中心
centroids = X[randidx[:K]]
return centroids
STEP2:计算每个样本到各聚类中心的距离,并将样本归入最近的聚类中心:
def find_closest_centroids(X, centroids):
# 获取聚类中心的数量,也即K值
K = centroids.shape[0]
# 初始化一个数组用于存储每个样本所属的聚类中心的索引
idx = np.zeros(X.shape[0], dtype=int)
# 遍历数据集中的每个样本
for i in range(X.shape[0]):
# 初始化一个列表用于存储当前样本到每个聚类中心的距离
distance = []
# 计算当前样本到每个聚类中心的距离
for j in range(centroids.shape[0]):
# 使用欧几里得距离公式计算样本i与聚类中心j之间的距离
norm_ij = np.linalg.norm(X[i] - centroids[j])
distance.append(norm_ij)
# 找出距离列表中的最小值,该最小值对应的索引就是当前样本所属的聚类中心
idx[i] = np.argmin(distance)
# 返回每个样本所属的聚类中心的索引数组
return idx
STEP3:重新计算每个聚类的中心,该中心为该类所有样本的平均值:
def compute_centroids(X, idx, K):
# 获取数据集X的行数m和列数n
# m表示样本数量,n表示每个样本的特征数量
m, n = X.shape
# 初始化一个K x n的零矩阵,用于存储K个聚类中心
# K表示聚类数量,n表示特征数量
centroids = np.zeros((K, n))
# 遍历每个聚类中心
for k in range(K):
# 从数据集X中选择属于当前聚类k的所有样本
# idx是一个长度为m的数组,存储了每个样本所属的聚类中心的索引
points = X[idx == k]
# 计算属于当前聚类k的所有样本的平均值,得到聚类中心
# axis=0表示按列计算平均值
centroids[k] = np.mean(points, axis=0)
# 返回计算得到的K个聚类中心
return centroids
STEP4:重复步骤2和3,直到满足以下条件之一:
聚类中心不再变化。
达到预设的最大迭代次数。
最小平方误差SSE(误差的平方和)达到预设的阈值。
此处直接以达到预设的最大迭代次数作为停止条件
def run_kMeans(X, initial_centroids, max_iters=10):
# 获取数据集X的行数m和列数n
# m表示样本数量,n表示每个样本的特征数量
m, n = X.shape
# 获取初始聚类中心的数量K
K = initial_centroids.shape[0]
# 将初始聚类中心赋值给centroids变量
centroids = initial_centroids
# 将初始聚类中心复制给previous_centroids变量,用于后续比较聚类中心是否发生变化
previous_centroids = centroids
# 初始化一个长度为m的零数组,用于存储每个样本所属的聚类中心的索引
idx = np.zeros(m)
# 开始运行K-means算法,最多迭代max_iters次
for i in range(max_iters):
# 输出当前迭代进度
print("K-Means iteration %d/%d" % (i, max_iters - 1))
# 调用find_closest_centroids函数,为数据集X中的每个样本找到最近的聚类中心,并返回索引数组
idx = find_closest_centroids(X, centroids)
# 调用compute_centroids函数,根据每个样本所属的聚类中心和索引数组,计算新的聚类中心
centroids = compute_centroids(X, idx, K)
# 返回最终的聚类中心和每个样本所属的聚类中心的索引
return centroids, idx
3、K-means的底层代码案例
此处直接使用吴恩达的案例,非常简洁直观嘞:
import numpy as np
import matplotlib.pyplot as plt
def load_data():
X = np.load("K_means_data/ex7_X.npy")
return X
def draw_line(p1, p2, style="-k", linewidth=1):
plt.plot([p1[0], p2[0]], [p1[1], p2[1]], style, linewidth=linewidth)
def plot_data_points(X, idx):
# plots data points in X, coloring them so that those with the same
# index assignments in idx have the same color
plt.scatter(X[:, 0], X[:, 1], c=idx)
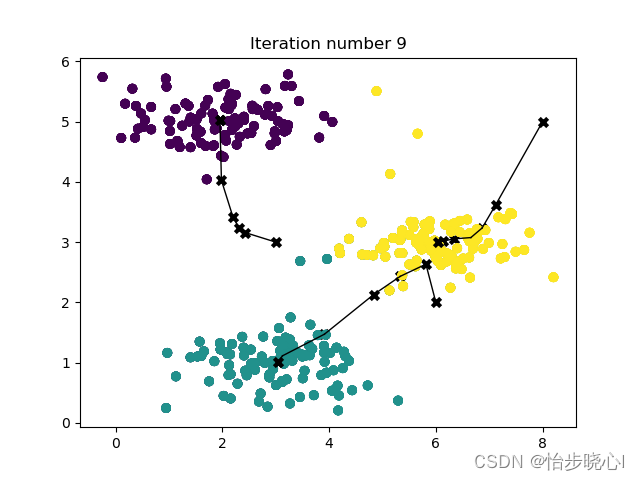
def plot_progress_kMeans(X, centroids, previous_centroids, idx, K, i):
# Plot the examples
plot_data_points(X, idx)
# Plot the centroids as black 'x's
plt.scatter(centroids[:, 0], centroids[:, 1], marker='x', c='k', linewidths=3)
# Plot history of the centroids with lines
for j in range(centroids.shape[0]):
draw_line(centroids[j, :], previous_centroids[j, :])
plt.title("Iteration number %d" % i)
def find_closest_centroids(X, centroids):
"""
Computes the centroid memberships for every example
Args:
X (ndarray): (m, n) Input values
centroids (ndarray): k centroids
Returns:
idx (array_like): (m,) closest centroids
"""
# Set K
K = centroids.shape[0]
# You need to return the following variables correctly
idx = np.zeros(X.shape[0], dtype=int)
for i in range(X.shape[0]):
# Array to hold distance between X[i] and each centroids[j]
distance = []
for j in range(centroids.shape[0]):
norm_ij = np.linalg.norm(X[i] - centroids[j])
distance.append(norm_ij)
idx[i] = np.argmin(distance)
return idx
# GRADED FUNCTION: compute_centpods
def compute_centroids(X, idx, K):
"""
Returns the new centroids by computing the means of the
data points assigned to each centroid.
Args:
X (ndarray): (m, n) Data points
idx (ndarray): (m,) Array containing index of closest centroid for each
example in X. Concretely, idx[i] contains the index of
the centroid closest to example i
K (int): number of centroids
Returns:
centroids (ndarray): (K, n) New centroids computed
"""
# Useful variables
m, n = X.shape
# You need to return the following variables correctly
centroids = np.zeros((K, n))
for k in range(K):
points = X[idx == k]
centroids[k] = centroids[k] = np.mean(points, axis=0)
return centroids
# You do not need to implement anything for this part
def run_kMeans(X, initial_centroids, max_iters=10, plot_progress=False):
"""
Runs the K-Means algorithm on data matrix X, where each row of X
is a single example
"""
# Initialize values
m, n = X.shape
K = initial_centroids.shape[0]
centroids = initial_centroids
previous_centroids = centroids
idx = np.zeros(m)
# Run K-Means
for i in range(max_iters):
# Output progress
print("K-Means iteration %d/%d" % (i, max_iters - 1))
# For each example in X, assign it to the closest centroid
idx = find_closest_centroids(X, centroids)
# Optionally plot progress
if plot_progress:
plot_progress_kMeans(X, centroids, previous_centroids, idx, K, i)
previous_centroids = centroids
# Given the memberships, compute new centroids
centroids = compute_centroids(X, idx, K)
plt.show()
return centroids, idx
# Load an example dataset
X = load_data()
# Set initial centroids
initial_centroids = np.array([[3,3],[6,2],[8,5]])
K = 3
# Number of iterations
max_iters = 10
centroids, idx = run_kMeans(X, initial_centroids, max_iters, plot_progress=True)
运行结果: