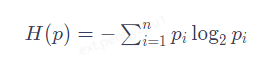文章目录
- 集成学习
- 随机森林
- 随机森林回归填补缺失值
- 实例:随机森林在乳腺癌数据上的调参
- 附录参数
集成学习
集成学习(ensemble learning)是时下非常流行的机器学习算法,它本身不是一个单独的机器学习算法,而是通过在数据上构建多个模型,集成所有模型的建模结果。
集成算法会考虑多个评估器的建模结果,汇总之后得到一个综合的结果,以此来获取比单个模型更好的回归或分类表现。
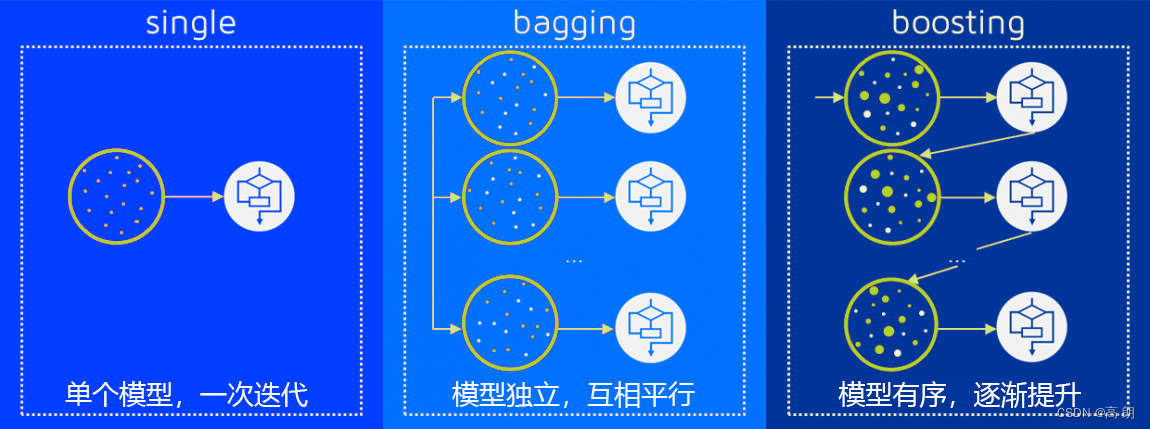
多个模型集成成为的模型叫做集成评估器(ensemble estimator),组成集成评估器的每个模型都叫做基评估器(base estimator)。通常来说,有三类集成算法:装袋法(Bagging),提升法(Boosting)和stacking。
Bagging的核心思想是构建多个相互独立的评估器,然后对其预测进行平均或多数表决原则来决定集成评估器的结果。装袋法的代表模型就是随机森林。Boosting,基评估器是相关的,是按顺序一一构建的。其核心思想是结合弱评估器的力量一次次对难以评估的样本进行预测,从而构成一个强评估器。提升法的代表模型有Adaboost和梯度提升树。
如何得到若干个个体学习器:
- 所有的个体学习器都是一个种类的,或者说是同质的。比如都是决策树个体学习器,或者都是神经网络个体学习器。
- 所有的个体学习器不全是一个种类的,或者说是异质的。比如我们有一个分类问题,对训练集采用支持向量机个体学习器,逻辑回归个体学习器和朴素贝叶斯个体学习器来学习,再通过某种结合策略来确定最终的分类强学习器。
如何选择一种结合策略:
比较常用的集成策略有直接平均、加权平均等。最直接的集成学习策略就是直接平均,即“投票”。我们先从讨论最常见的基于多数票机制的集成方法。简单来说,多数票机制就是选择多数分类器所预测的分类标签,也就是那些获得50%以上支持的预测结果。
可以看一下scikit-learn库中有关集成学习的类:
| 类 | 类的功能 |
|---|---|
| ensemble.AdaBoostClassifier | AdaBoost分类 |
| ensemble.AdaBoostRegressor | Adaboost回归 |
| ensemble.BaggingClassifier | 装袋分类器 |
| ensemble.BaggingRegressor | 装袋回归器 |
| ensemble.ExtraTreesClassifier | Extra-trees分类(超树,极端随机树) |
| ensemble.ExtraTreesRegressor | Extra-trees回归 |
| ensemble.GradientBoostingClassifier | 梯度提升分类 |
| ensemble.GradientBoostingRegressor | 梯度提升回归 |
| ensemble.IsolationForest | 隔离森林 |
| ensemble.RandomForestClassifier | 随机森林分类 |
| ensemble.RandomForestRegressor | 随机森林回归 |
| ensemble.RandomTreesEmbedding | 完全随机树的集成 |
| ensemble.VotingClassifier | 用于不合适估算器的软投票/多数规则分类器 |
随机森林
随机森林算法可以简单概况为以下四个步骤:
- 随机提取一个规模为n的bootstrap样本(从训练集中有放回的随机选择n个样本)
- 基于提取的bootstrap样本生成决策树。在每个结点上完成以下任务:
- 不放回的选取d个特征;
- 根据目标函数的要求,例如信息增益最大化,使用选定的最佳特征来分裂结点。
- 把步骤1和2重复k次
- 聚合每棵树的预测结果,并且以多数票机制确定标签的分类。
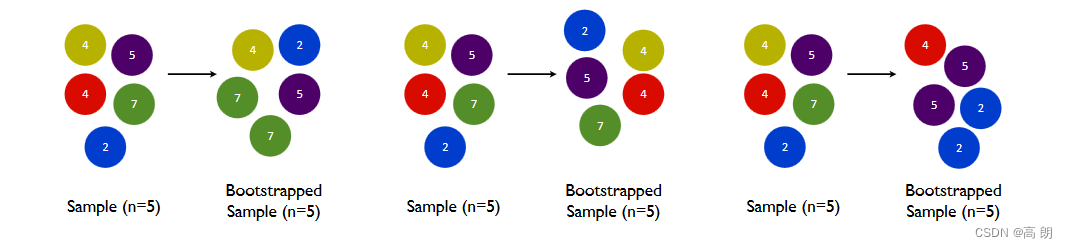
关于bootstrap样本不放回说明:
在一个含有n个样本的原始训练集中,我们进行随机采样,每次采样一个样本,并在抽取下一个样本之前将该样本放回原始训练集,也就是说下次采样时这个样本依然可能被采集到,这样采集n次,最终得到一个和原始训练集一样大的,n个样本组成的自助集。
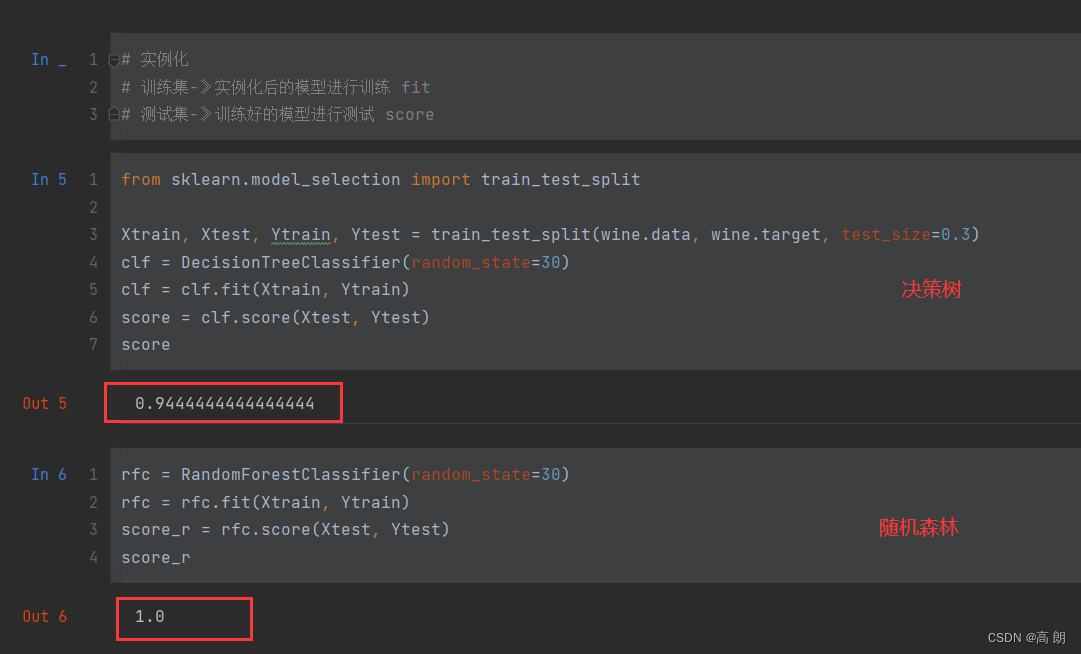 我们用交叉验证再次比较单颗决策树和随机森林:
我们用交叉验证再次比较单颗决策树和随机森林:
# 交叉验证:
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
rfc = RandomForestClassifier(n_estimators=30)
rfc_s = cross_val_score(rfc, wine.data, wine.target, cv=10)
clf = DecisionTreeClassifier()
clf_s = cross_val_score(clf, wine.data, wine.target, cv=10)
plt.plot(range(1,11), rfc_s, label='RandomForest')
plt.plot(range(1,11), clf_s, label='DecisionTree')
plt.legend()
plt.show()
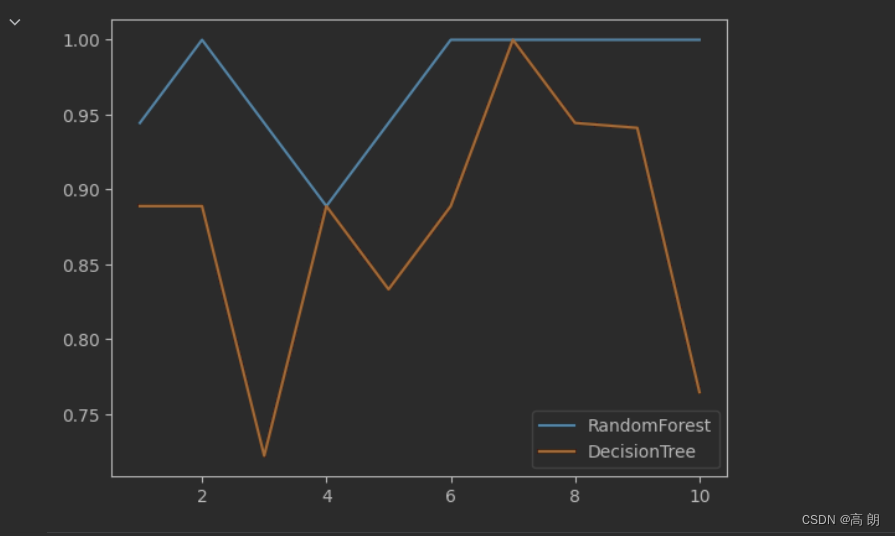 随机森林始终大于等于单科决策树。
随机森林始终大于等于单科决策树。
关于随机森林scikit-learn里面的RandomForestClassifier类参数详解:
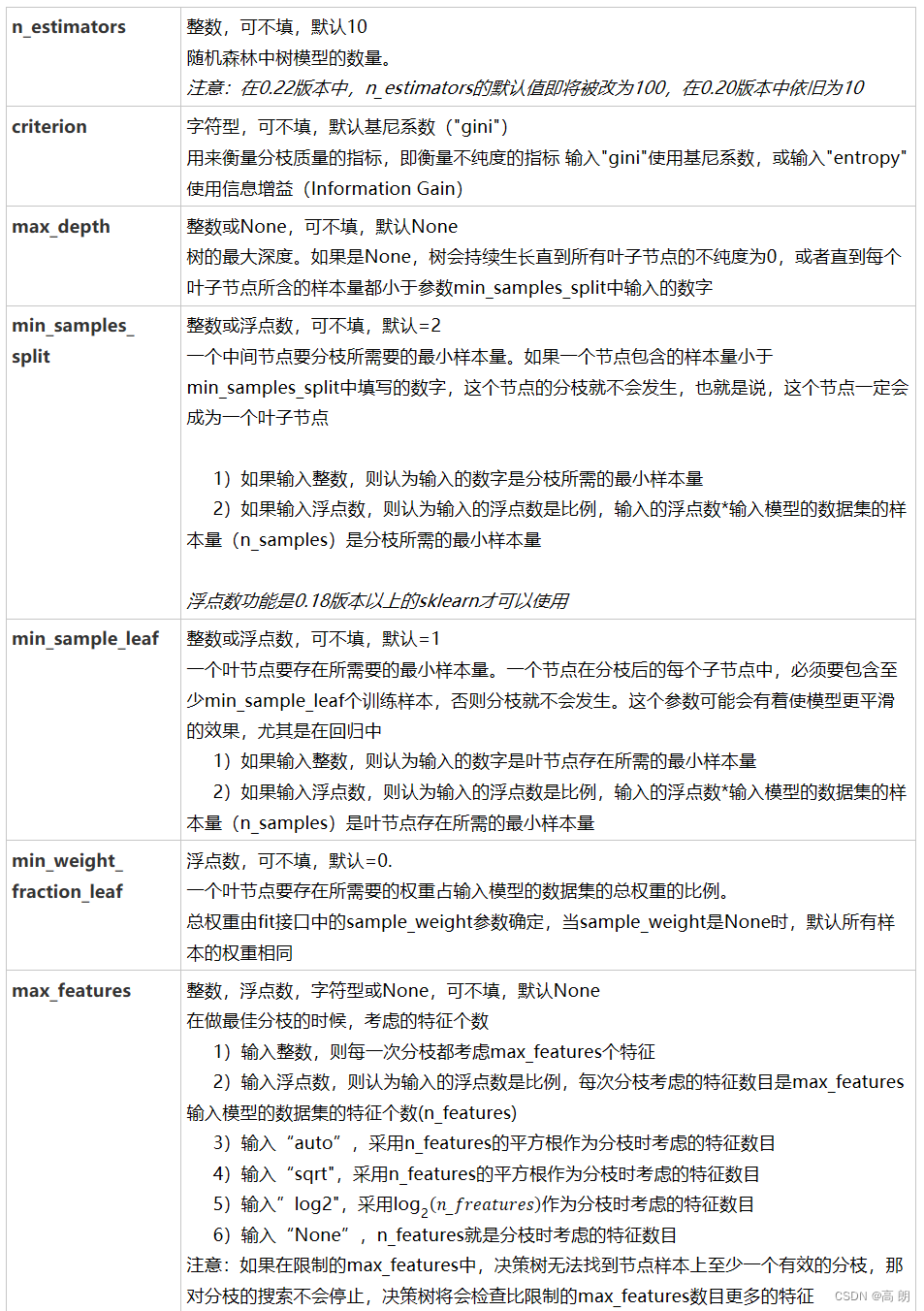
n_estimators:这是森林中树木的数量,即基评估器的数量。
这个参数对随机森林模型的精确性影响是单调的,n_estimators越
大,模型的效果往往越好。但是相应的,任何模型都有决策边界,n_estimators达到一定的程度之后,随机森林的精确性往往不在上升或开始波动,并且,n_estimators越大,需要的计算量和内存也越大,训练的时间也会越来越长。对于这个参数,我们是渴望在训练难度和模型效果之间取得平衡。
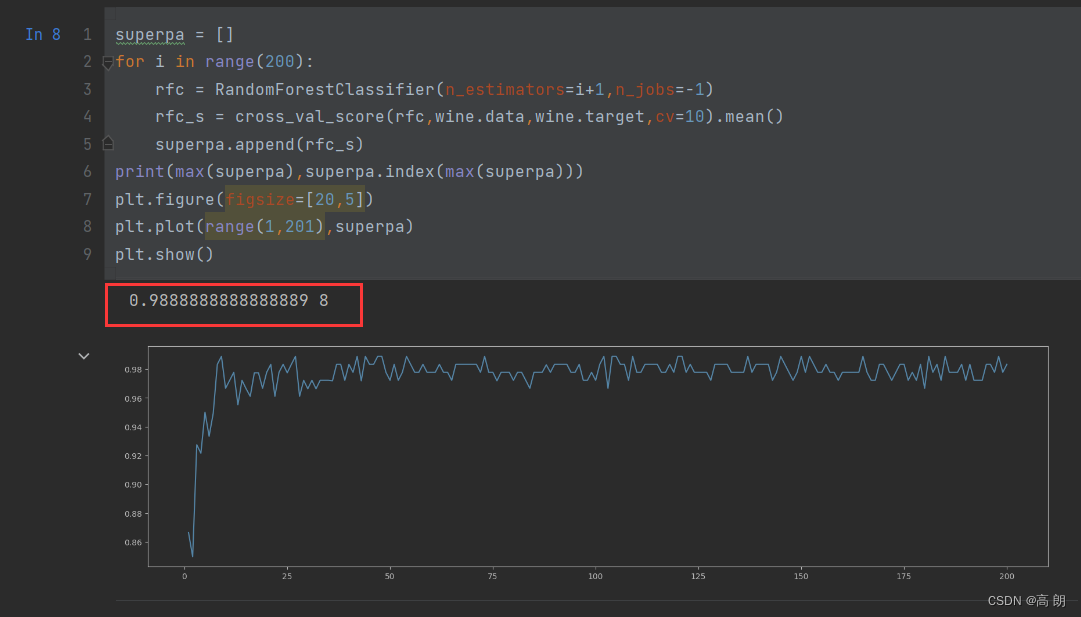
superpa = []
for i in range(200):
rfc = RandomForestClassifier(n_estimators=i+1,n_jobs=-1)
rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10).mean()
superpa.append(rfc_s)
print(max(superpa),superpa.index(max(superpa)))
plt.figure(figsize=[20,5])
plt.plot(range(1,201),superpa)
plt.show()
可以通过这个来找到最佳的n_estimators值
2. random_state
随机森林中其实也有random_state,用法和分类树中相似,只不过在分类树中,一个random_state只控制生成一棵树,而随机森林中的random_state控制的是生成森林的模式,而非让一个森林中只有一棵树。
 当
当random_state固定时,随机森林中生成是一组固定的树,但每棵树依然是不一致的,这是
用”随机挑选特征进行分枝“的方法得到的随机性。并且我们可以证明,当这种随机性越大的时候,袋装法的效果一
般会越来越好。用袋装法集成时,基分类器应当是相互独立的,是不相同的。
- 其他:
随机森林回归填补缺失值
基本思想:
数据集=特征数据+标签,因为是有监督的学习,我们的标签数据肯定是完整的,假设特征数据中特征A有缺失值需要填充,可以这样做:
- 特征数据 = 特征数据(除去特征A) + 标签
- 标签=特征A
- 现在数据集的特点是:特征数据完整,标签数据有缺失值
- 拿标签数据不缺失的数据进行训练,训练完成后,拿模型对缺失的数据进行预测填充,这就完成了我们数据集的填充。
上面的情况只是一列缺失,也就是一个特征缺失,对于多个特征都有缺失的情况该怎么处理:
- 遍历所有的特征,从缺失最少的开始进行填补(因为填补缺失最少的特征所需要的准确信息最少)。
- 填补一个特征时,先将其他特征的缺失值用0代替,每完成一次回归预测,就将预测值放到原本的特征矩阵中,再继续填补下一个特征。每一次填补完毕,有缺失值的特征会减少一个,所以每次循环后,需要用0来填补的特征就越来越少。当进行到最后一个特征时(这个特征应该是所有特征中缺失值最多的),已经没有任何的其他特征需要用0来进行填补了,而我们已经使用回归为其他特征填补了大量有效信息,可以用来填补缺失最多的特征。
- 遍历所有的特征后,数据就完整,不再有缺失值了。
X_missing_reg = X_missing.copy() # 对原始数据进行copy
sortindex = np.argsort(X_missing_reg.isnull().sum(axis=0)).values # 对缺失特征的数目进行排序并返回索引下标顺序
for i in sortindex:
#构建我们的新特征矩阵和新标签
df = X_missing_reg
fillc = df.iloc[:,i] # 要填充的特征=》标签
df = pd.concat([df.iloc[:,df.columns != i],pd.DataFrame(y_full)],axis=1) # 除去要需要填充的特征的特征数据+标签=》特征数据
#在新特征矩阵中,对含有缺失值的列,进行0的填补
df_0 =SimpleImputer(missing_values=np.nan,
strategy='constant',fill_value=0).fit_transform(df)
#找出我们的训练集和测试集
Ytrain = fillc[fillc.notnull()] # 新标签里面没有缺失的值
Ytest = fillc[fillc.isnull()] # 新标签里面缺失的值,后面进行预测填充
Xtrain = df_0[Ytrain.index,:] # 训练集
Xtest = df_0[Ytest.index,:] # 测试集
#用随机森林回归来填补缺失值
rfc = RandomForestRegressor(n_estimators=100)
rfc = rfc.fit(Xtrain, Ytrain)
Ypredict = rfc.predict(Xtest)
#将填补好的特征返回到我们的原始的特征矩阵中
X_missing_reg.loc[X_missing_reg.iloc[:,i].isnull(),i] = Ypredict
实例:随机森林在乳腺癌数据上的调参
- 导入所需要的库
from sklearn.datasets import load_breast_cancer # 乳腺癌的数据集
from sklearn.ensemble import RandomForestClassifier # 随机森林分类树
from sklearn.model_selection import GridSearchCV # 网格搜索 调参
from sklearn.model_selection import cross_val_score # 交叉验证
import matplotlib.pyplot as plt # 画图
import pandas as pd
import numpy as np
- 数据处理
data = load_breast_cancer()
# jupyter 查看数据信息
data
data.data.shape
data.target
- 简单建模
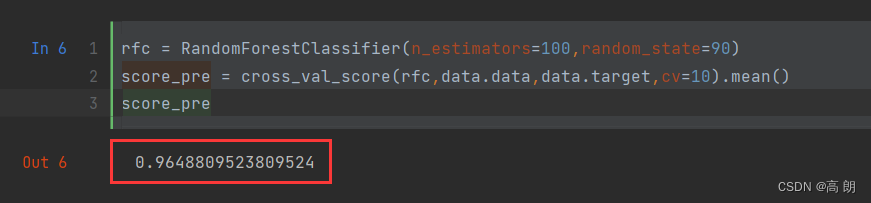
rfc = RandomForestClassifier(n_estimators=100,random_state=90)
score_pre = cross_val_score(rfc,data.data,data.target,cv=10).mean()
score_pre
- 调参优化
在机器学习中,我们用来衡量模型在未知数据上的准确率的指标,叫做泛化误差(Genelization error)
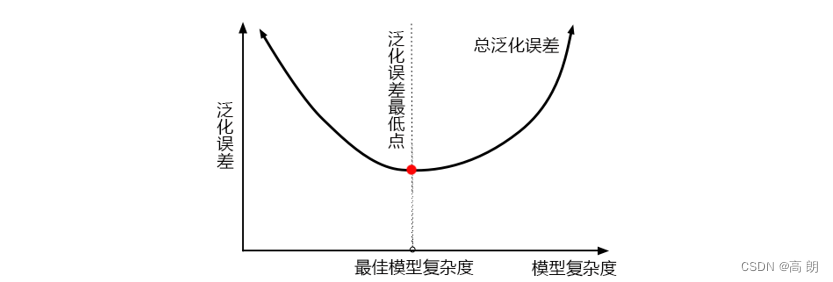 最佳模型是泛化误差最低的点,左边是欠拟合,模型不够复杂,右边是过拟合,模型太过复杂。
最佳模型是泛化误差最低的点,左边是欠拟合,模型不够复杂,右边是过拟合,模型太过复杂。
对树模型来说,树越茂盛,深度越深,枝叶越多,模型就越复杂。
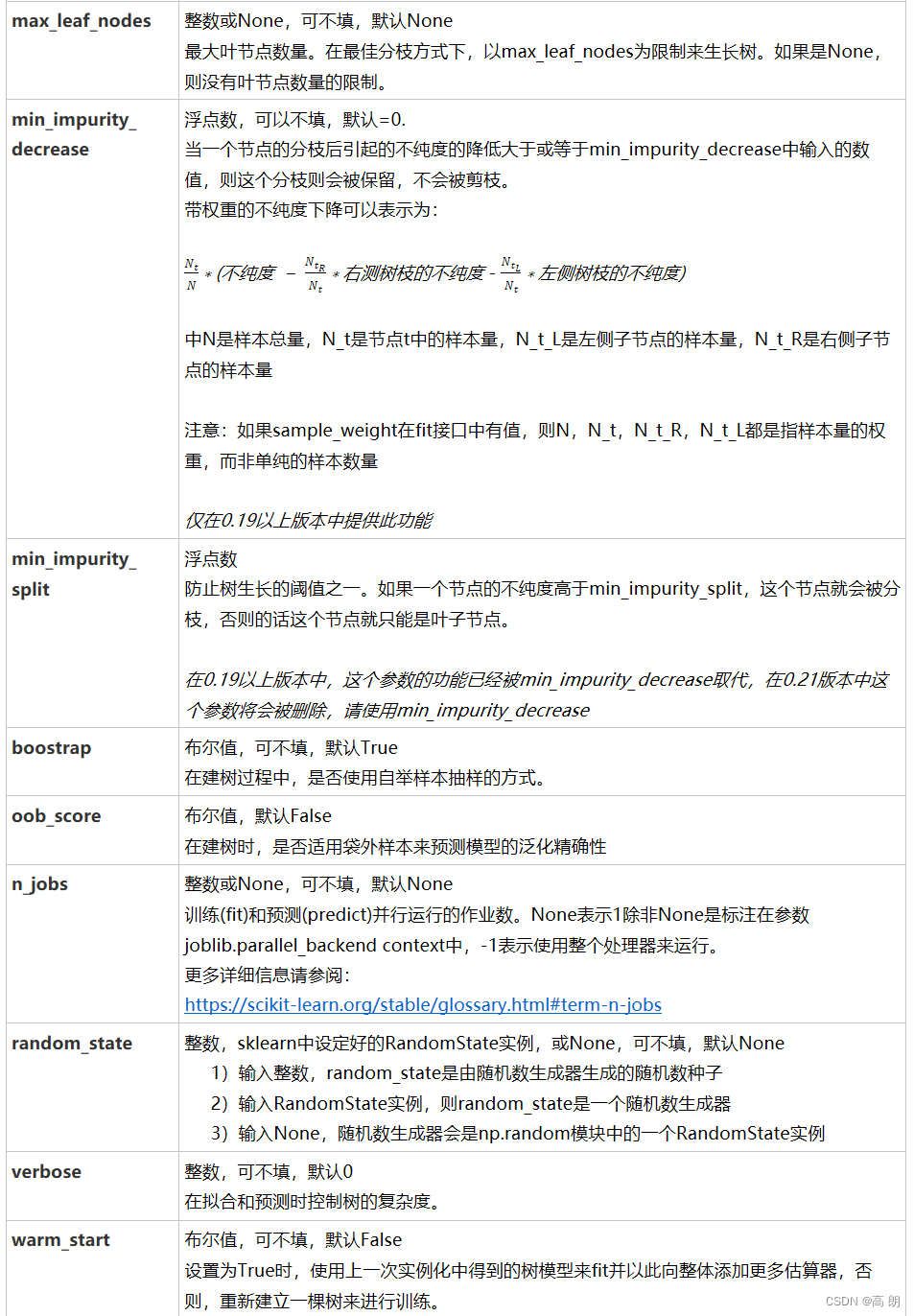
对于随机森林最终的几个参数:
| 参数 | 对模型在未知数据上的评估性能的影响 | 影响程度 |
|---|---|---|
| n_estimators | 提升至平稳,n_estimators↑,不影响单个模型的复杂度 | ⭐⭐⭐⭐ |
| max_depth | 有增有减,默认最大深度,即最高复杂度,向复杂度降低的方向调参max_depth↓,模型更简单,且向图像的左边移动 | ⭐⭐⭐ |
| min_samples _leaf | 有增有减,默认最小限制1,即最高复杂度,向复杂度降低的方向调参min_samples_leaf↑,模型更简单,且向图像的左边移动 | ⭐⭐ |
| min_samples _split | 有增有减,默认最小限制2,即最高复杂度,向复杂度降低的方向调参min_samples_split↑,模型更简单,且向图像的左边移动 | ⭐⭐ |
| max_features | 有增有减,默认auto,是特征总数的开平方,位于中间复杂度,既可以向复杂度升高的方向,也可以向复杂度降低的方向调参max_features↓,模型更简单,图像左移max_features↑,模型更复杂,图像右移max_features是唯一的,既能够让模型更简单,也能够让模型更复杂的参数,所以在调整这个参数的时候,需要考虑我们调参的方向 | ⭐ |
| criterion | 有增有减,一般使用gini | 看具体情况 |
1)n_estimators
初步大范围的调:
scorel = []
for i in range(0,200,10):
rfc = RandomForestClassifier(n_estimators=i+1,
n_jobs=-1,
random_state=90)
score = cross_val_score(rfc,data.data,data.target,cv=10).mean()
scorel.append(score)
print(max(scorel),(scorel.index(max(scorel))*10)+1)
plt.figure(figsize=[20,5])
plt.plot(range(1,201,10),scorel)
plt.show()
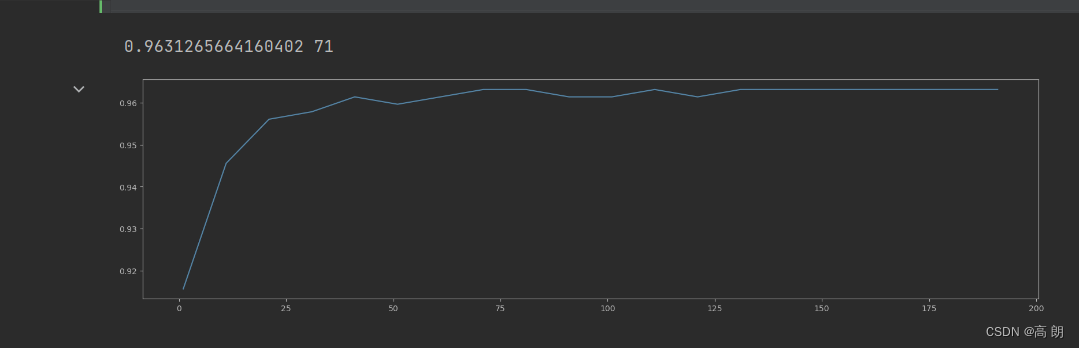 大范围确定
大范围确定n_estimators 在71附近,缩小范围再次调整:
scorel = []
for i in range(65,75):
rfc = RandomForestClassifier(n_estimators=i,
n_jobs=-1,
random_state=90)
score = cross_val_score(rfc,data.data,data.target,cv=10).mean()
scorel.append(score)
print(max(scorel),([*range(65,75)][scorel.index(max(scorel))]))
plt.figure(figsize=[20,5])
plt.plot(range(65,75),scorel)
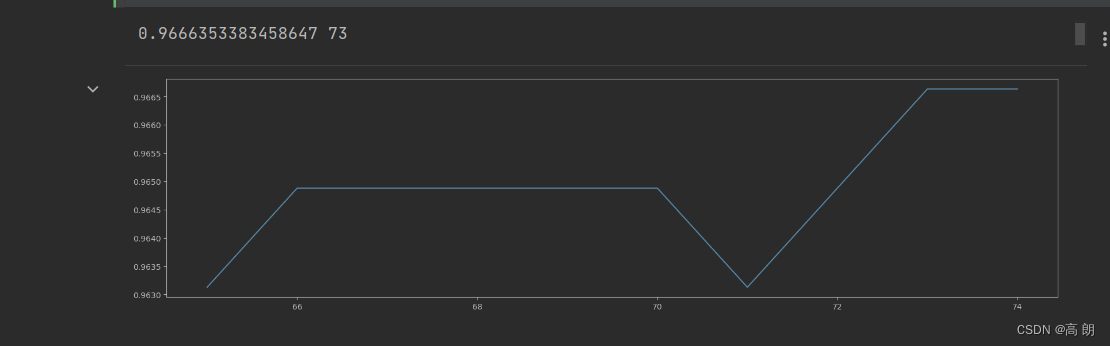 可以确定
可以确定n_estimators=73,相比之前的准确率是有提升的。
2)max_depth
采用网格搜索:
# 调整max_depth
param_grid = {'max_depth':np.arange(1, 20, 1)}
# 一般根据数据的大小来进行一个试探,乳腺癌数据很小,所以可以采用1~10,或者1~20这样的试探
# 但对于像digit recognition那样的大型数据来说,我们应该尝试30~50层深度(或许还不足够
# 更应该画出学习曲线,来观察深度对模型的影响
rfc = RandomForestClassifier(n_estimators=73
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)
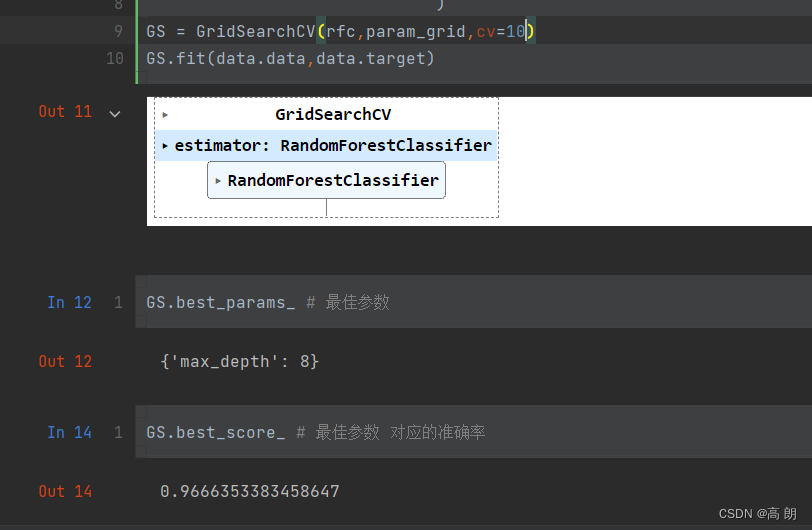 可以发现准确率没有变化,可以不设置这个参数。
可以发现准确率没有变化,可以不设置这个参数。
3)其他参数也可以采用网格搜索来找出:
"""
有一些参数是没有参照的,很难说清一个范围,这种情况下我们使用学习曲线,看趋势
从曲线跑出的结果中选取一个更小的区间,再跑曲线
param_grid = {'n_estimators':np.arange(0, 200, 10)}
param_grid = {'max_depth':np.arange(1, 20, 1)}
param_grid = {'max_leaf_nodes':np.arange(25,50,1)}
对于大型数据集,可以尝试从1000来构建,先输入1000,每100个叶子一个区间,再逐渐缩小范围
有一些参数是可以找到一个范围的,或者说我们知道他们的取值和随着他们的取值,模型的整体准确率会如何变化,这
样的参数我们就可以直接跑网格搜索
param_grid = {'criterion':['gini', 'entropy']}
param_grid = {'min_samples_split':np.arange(2, 2+20, 1)}
param_grid = {'min_samples_leaf':np.arange(1, 1+10, 1)}
param_grid = {'max_features':np.arange(5,30,1)}
"""
附录参数