最新一届的蛋白质结构预测奥林匹克大赛,即15届CASP比赛(CASP15),在日前拉下了帷幕。这正值谷歌团队AlphaFold2在上一届CASP大赛给该领域带来革命性冲击后两周年。两年后,该领域的状况如何,蛋白质结构预测该何去何从?为此,Nature杂志发表了Ewen Callaway博士关于CASP15的专题文章。该文章对CASP大赛的组织者John Moult博士,以及蛋白质结构预测专家Yang Zhang(张阳)教授,Mohammed AlQuraishi博士,Arne Elofsson教授和 CASP大赛的评审员Ezgi Karaca博士进行了采访。
后AlphaFold时代的蛋白质结构预测
两年前,计算生物学家John Moult博士宣布:“从某种意义上说,问题已经解决了。”总部位于伦敦的公司 DeepMind 刚刚横扫了由Moult共同创立的两年一度的竞赛。该竞赛测试了各代表团队预测蛋白质结构的能力,这是生物学中最重大的挑战之一。其中DeepMind的AlphaFold凭借其革命性的人工智能 (AI) 算法在该项比赛独占鳌头。
两年后,Moult 的竞赛,结构预测的关键评估(CASP),仍然走在 AlphaFold 的长长阴影中。今年即本周末在土耳其安塔利亚举办的CASP15的结果表明,目前由氨基酸序列预测蛋白质结构的最成功方法都可以看到AlphaFold的影子。“每个人都在使用 AlphaFold,”安娜堡密歇根大学的计算生物学家张阳教授说。
然而,AlphaFold 的进展也为蛋白质结构预测领域带来了新的挑战。超越现有蛋白质结构预测的精度需要新的技术和算法。“唾手可得的果实已经摘下,”纽约市哥伦比亚大学的计算生物学家 Mohammed AlQuraishi博士说。“接下来的一些问题会更难。”
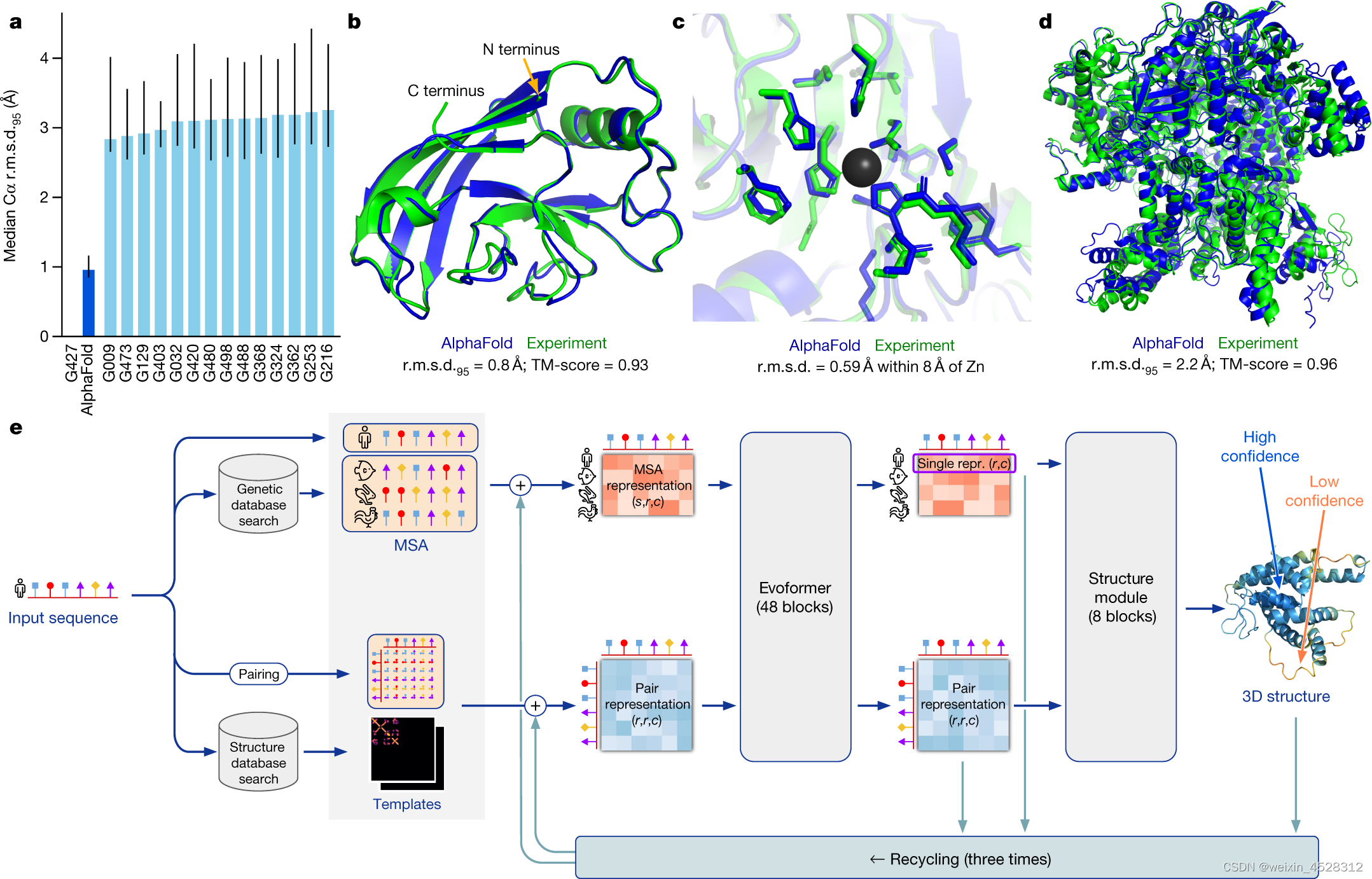
DeepMind团队2021年在Nature上发表的AlphaFold2 算法流程图
挑战AlphaFold
CASP大赛始于 1994 年,旨在为蛋白质结构预测领域带来最为严谨的评估,以此来推动计算生物学领域加速理解细胞构建模块和推进药物发现的努力。在每次比赛的过程中,各个计算预测团队的任务是使用计算工具来预测已使用 X 射线晶体学和低温电子显微镜等实验方法确定但尚未发布的蛋白质结构。然后,由独立的评审科学家团队,通过把计算机预测模型和实验结构直接比较,来对各预测团队的模型进行评估打分。上届CASP14的结果表明,许多蛋白质的计算机预测结构和实验解出的结构没有多大差别。这是CASP比赛历史上第一次达到这样的准确性。
自从CASP14之后,AlphaFold 在生命科学研究中变得无所不在。DeepMind在 2021 年发布了该软件的底层代码,以便任何人都可以运行该程序。今年更新的 AlphaFold 数据库保存了基因组数据库中代表的几乎所有生物体的几乎每一种蛋白质的预测结构。虽然预测质量参差不齐,但是其结构总数超过 2 亿条蛋白质。
AlphaFold 的成功和新发现的普遍性对马里兰大学的John Moult博士和他的同事们提出了挑战。他们正在计划今年的 CASP15。“人们说,'哦,我们不再需要 CASP,问题已经解决了。' 我认为这是完全错误的方式。”Moult博士说。
在 CASP15 上,最成功的团队是那些以各种方式采用和推广AlphaFold算法的团队,同时也取得了相当不错的精度提高。“但是AlphaFold的准确度已经足够高,很难取得比它好很多的精度”,Moult博士说。
蛋白质复合物结构预测
为了使比赛在后 AlphaFold 世界中更具相关性,Moult博士 和他的团队增加了新的测试并调整了一些现有的测试范畴。新的测试包括确定蛋白质如何与药物等其他分子相互作用,以及预测某些蛋白质可能呈现的多种形状。Moult 说,在过去的十年中,CASP 已经包含了多种相互作用的蛋白质相互作用的“复合物”,但今年对准确预测此类分子的结构提出了更高的要求。
“这是正确的做法,”张阳教授说,“因为预测单个蛋白质或结构域的结构——过去 CASP 比赛的奶油和面包——很大程度上已经被 AlphaFold 解决了”。斯德哥尔摩大学蛋白质生物信息学家 Arne Elofsson 教授同意这种说法。 他说,“确定蛋白质复合物的形状尤其代表了该领域的一项重要新挑战,因为还有很大的改进空间。”
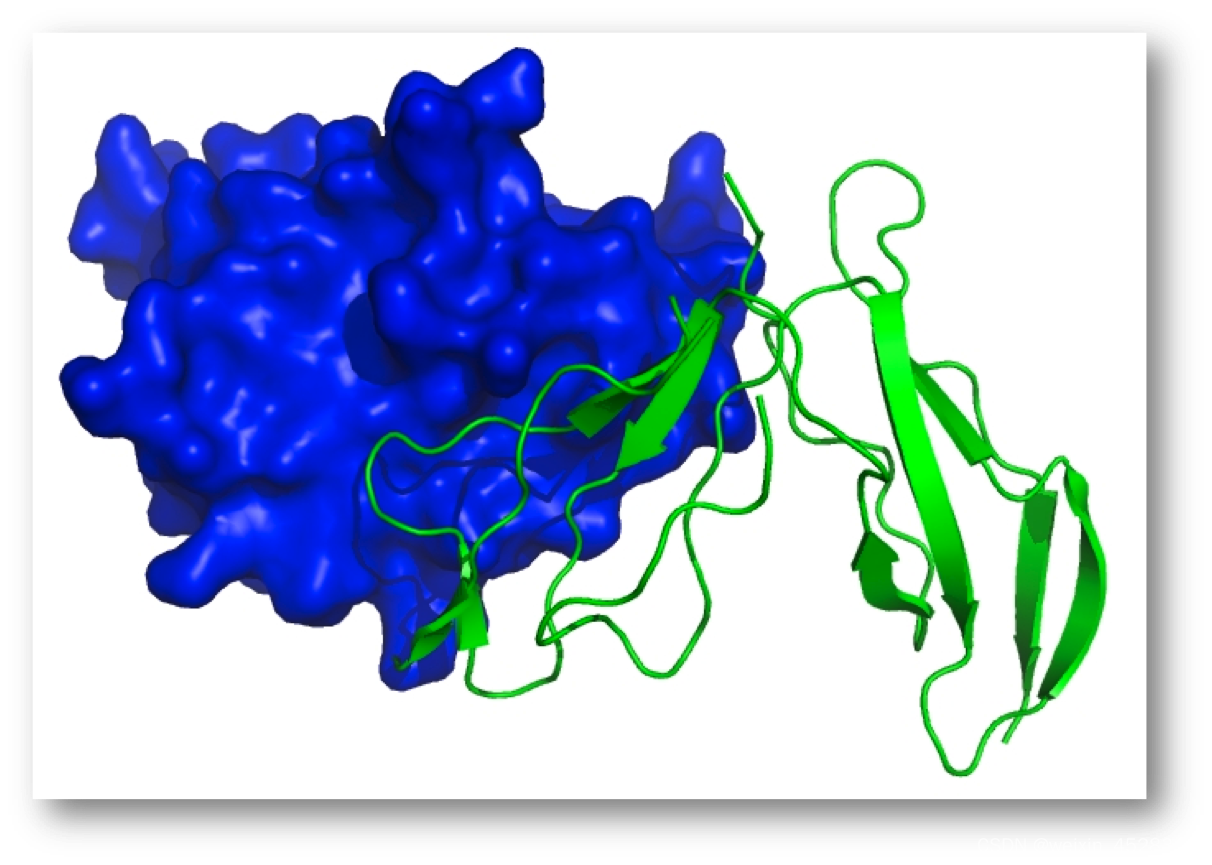 蛋白质复合物结构
蛋白质复合物结构
AlphaFold 最初设计的版本是用于预测单个蛋白质的形状。但是,在其公开发布的几天内,其他科学家表明该软件可以被改装,用来模拟蛋白质之间的相互作用。从此后的几个月里,研究人员想出了无数种方法来提高 AlphaFold 处理复合物的能力。考虑到这个目标,DeepMind 甚至发布了一个名为 AlphaFold-Multimer 的新版本。
这样的努力似乎得到了回报,因为与之前的比赛相比,CASP15 的准确复合物数量显着增加。这可能主要是由于采用了 AlphaFold 的方法。“这对我们来说是一个接近复合物实验精度的新游戏。” Moult博士说。“我们也有一些失败。”
例如,团队对由两种相同的交织蛋白质组成的功能未知的病毒分子做出了惊人准确的预测。土耳其伊兹密尔生物医学和基因组中心的计算结构生物学家 Ezgi Karaca 博士评估了蛋白质复合物结构的预测。“这种形状让 AlphaFold 之前的工具无所适从。” Karaca 博士说,“AlphaFold 的标准版本未能准确模拟一个巨大的 20 链细菌酶的形状,但一些参赛团队通过对AlphaFold的推广对这些蛋白质复合物产生了正确的预测模型。
与此同时,这些团队努力预测涉及称为抗体的免疫分子的复合物——包括一些附着在 SARS-CoV-2 蛋白上的分子。 Karaca 博士说,“一些团队的预测已经取得了成功,这表明改编和推广AlphaFold 将有助于预测这些医学上重要的分子的形状。
下一步的方向
人们也注意到,DeepMind没有参加今年的 CASP 比赛。该公司没有说明不参与的原因,但在 CASP15 会议期间发布了一份简短声明,祝贺参赛的团队。与此同时,它预计推出对 AlphaFold2 更新的版本,以帮助研究人员根据新的算法进行基准测试。
其他研究人员表示,参加 CASP 比赛是一项非常耗时费力的努力,该公司可能觉得将这些时间花在其他科研挑战上会更加有益。“如果他们参与进来,对我们来说会很好,”Moult博士说。但他补充说,“因为这个算法已经非常精确,他们无法实现另一个大飞跃”。
研究人员表示,对 AlphaFold 进行重大改进需要时间,并且可能需要在机器学习和蛋白质结构预测方面进行新的创新。正在开发的一个领域是“语言模型”的应用,例如预测文本工具中使用的那些模型,以预测蛋白质结构。但是这些方法——包括由社交网络巨头 Meta 开发的方法——在 CASP15 上的表现,并不如基于 AlphaFold 的工具。
然而,此类工具可能有助于预测突变如何改变蛋白质的结构——这是由于 AlphaFold 的成功而出现的蛋白质结构预测中的几个关键挑战之一。AlQuraishi 博士说,由于这一点,该领域不再专注于单一目标。“有许多这样的问题值得研究。”
参考文献:
After AlphaFold: protein-folding contest seeks next big breakthrough