目录
- 分组条件期望(GroupedCE)解释程序
- 创建虚拟环境
- 导包
- 加载数据集
- 训练模型
- 计算独立条件期望ICE
- plot_ice_explanation
- 计算分组条件期望 (GCE)
- plot_gce_explanation
记录一下学习过程,官方的代码在https://github.com/Trusted-AI/AIX360/tree/master/examples/gce。
分组条件期望(GroupedCE)解释程序
分组条件期望(GroupedCE)解释器是一个本地的、与模型无关的解释器,它为给定的实例和一组功能生成分组条件期望图。特征集可以是用户定义的输入协变量的子集,也可以是基于全局解释器(例如SHAP)提供的重要性的前K个特征。解释器生成三维图,包含特征对同时变化时的模型输出。如果提供了单个特征,则解释器生成标准的2D ICE图,其中一次仅扰动一个特征。
创建虚拟环境
为了避免环境污染,为aix360的实验创建一个虚拟环境。
https://blog.csdn.net/weixin_45735391/article/details/133197625
导包
import os
# to suppress unrelated tensorflow warnings while importing aix360.datasets
os.environ["TF_CPP_MIN_LOG_LEVEL"] = "3"
import numpy as np
import pandas as pd
加载数据集
数据来自这里。该数据集的标准化版本可以在 sklearn找到。如来源链接中所述,该数据集由10个基线变量组成,年龄、性别、体重指数、平均血压,为n=442名糖尿病患者中的每一位获得了6个血清测量值,以及感兴趣的反应,即基线后一年疾病进展的定量测量值。
划分训练集和测试集:
- test_size=0.2:测试机占比20%
- random_state=42:随机数的种子
随机数种子:其实就是该组随机数的编号,在需要重复试验的时候,保证得到一组一样的随机数。比如你每次都填1,其他参数一样的情况下你得到的随机数组是一样的。但填0或不填,每次都会不一样。 - return_only_numerical=True:把SEX这一列特征去掉
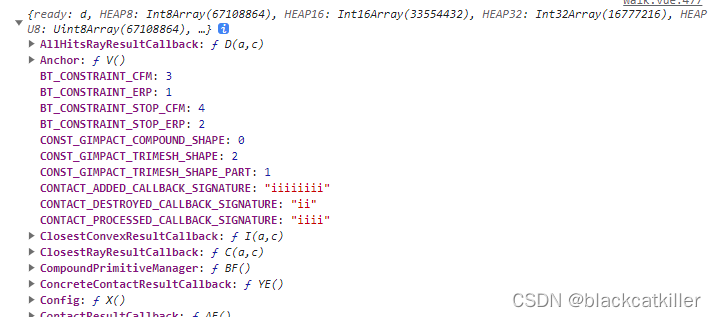
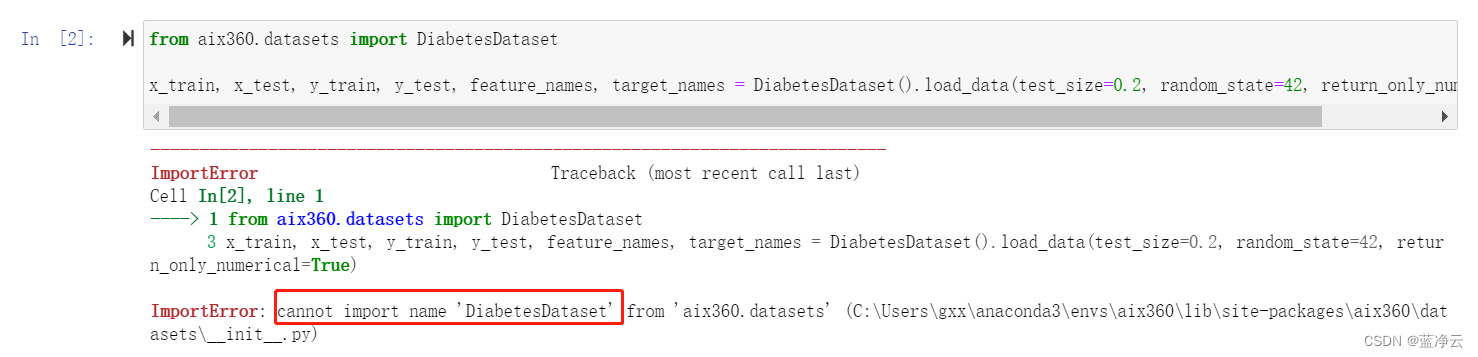
from aix360.datasets import DiabetesDataset
x_train, x_test, y_train, y_test, feature_names, target_names = DiabetesDataset().load_data(test_size=0.2, random_state=42, return_only_numerical=True)
刚运行就报错,确实有点烦。。。
根据提示找到__init__.py文件

运行一下diabetes_dataset.py里面的代码试试

没有requests模块,问题不大,pip install就好了
pip install requests

再次运行diabetes_dataset.py里面的代码,已经不报错了。
好像有点顺利,然后再次运行刚才的代码,还是那个错误,咦?重启一下notebook,OK了,确实有点顺利,哈哈哈。。。
它这个报错只是告诉你不能正常加载数据集,而不是直接告诉你缺少requests模块,有点坑。。。
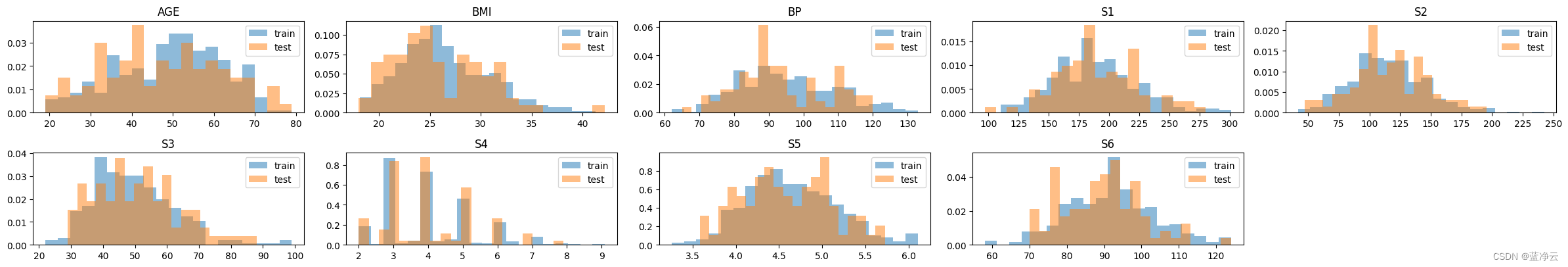
Diabetes数据集有10个特征,前面把SEX去掉了,所以还剩下age、bmi、bp、s1、s2、s3、s4、s5、s6这九个特征。
下面是这九个特征的直方图。
import matplotlib.pyplot as plt
plt.figure(figsize = (24,20))
for i in np.arange(x_train.shape[1]):
plt.subplot(10,5,i+1)
plt.title(feature_names[i])
plt.hist(x_train[:,i], 20, label = 'train',density = True,alpha = 0.5)
plt.hist(x_test[:,i], 20, label = 'test',density = True,alpha = 0.5)
plt.legend()
plt.tight_layout()
plt.show()
运行结果:
训练模型
用scikit-learn的RandomForestRegressor模型进行训练。
from sklearn.ensemble import RandomForestRegressor
clf = RandomForestRegressor().fit(x_train, y_train)
print('Score on train data :',clf.score(x_train, y_train))
print('Score on test data :',clf.score(x_test, y_test))
运行结果:
Score on train data : 0.9225497874165971
Score on test data : 0.40015247074701676
计算独立条件期望ICE
独立条件期望图(Individual Conditional Expectation Plot,ICE),描述的是每个个体的预测值与单一变量之间的关系,为每个实例显示一条曲线,显示当特征发生变化时实例的预测如何变化,可视化预测对每个实例的特征的关系。
ICE图的优点在于易于理解,能够避免数据异质的问题;缺点在于只能反映单一特征变量与目标之间的关系,受制于变量独立假设的要求,同时ICE图像往往由于个体过多导致图像看起来过于冗杂,不容易获取解释信息。
pip install shap
又报错,奇奇怪怪的毛病真多。。。

运行下面的命令,安装成功,重启一下notebook。
pip install --user -i https://pypi.tuna.tsinghua.edu.cn/simple shap
from aix360.algorithms.gce.gce import GroupedCEExplainer
n_test_samples = 25 # number of instances to explain
n_samples = 100 # number of samples to generate for selected feature
ice_explanations = {}
for i, feature_col in enumerate(feature_names):
ice_explainer = GroupedCEExplainer(model=clf.predict,
data=x_train,
feature_names=feature_names,
n_samples=n_samples,
features_selected=[feature_col],
random_seed=22
)
ice_explanations[feature_col] = []
for i in range(n_test_samples):
ice_explanations[feature_col].append(ice_explainer.explain_instance(instance=x_test[[i], :]))
一步一个bug,刺激。。。

首先,在base环境下安装widgetsnbextension:
conda activate base
pip install widgetsnbextension
然后,回到运行环境,安装ipywidgets:
conda activate aix360
pip install ipywidgets
装完再重启一下notebook。
有个警告。。。

本来想着再找一下源码,额,还是太天真了。。。没找着tqdm在哪,,,干脆在前面加上。。。然后就没这个提示了。。。
from tqdm import tqdm
plot_ice_explanation
plots.prot_ice_explanation具有用于绘制ICE解释的辅助代码。对于不同的数据集或绘图变体,可以更新代码“plots.py”。
pip install plotly
from plots import plot_ice_explanation
plot_ice_explanation(ice_explanations, title="GroupedCE (Individual Conditional Expectation) Explanation Plot for {}-{} Test Instances.".format(0, n_test_samples-1))
运行结果:
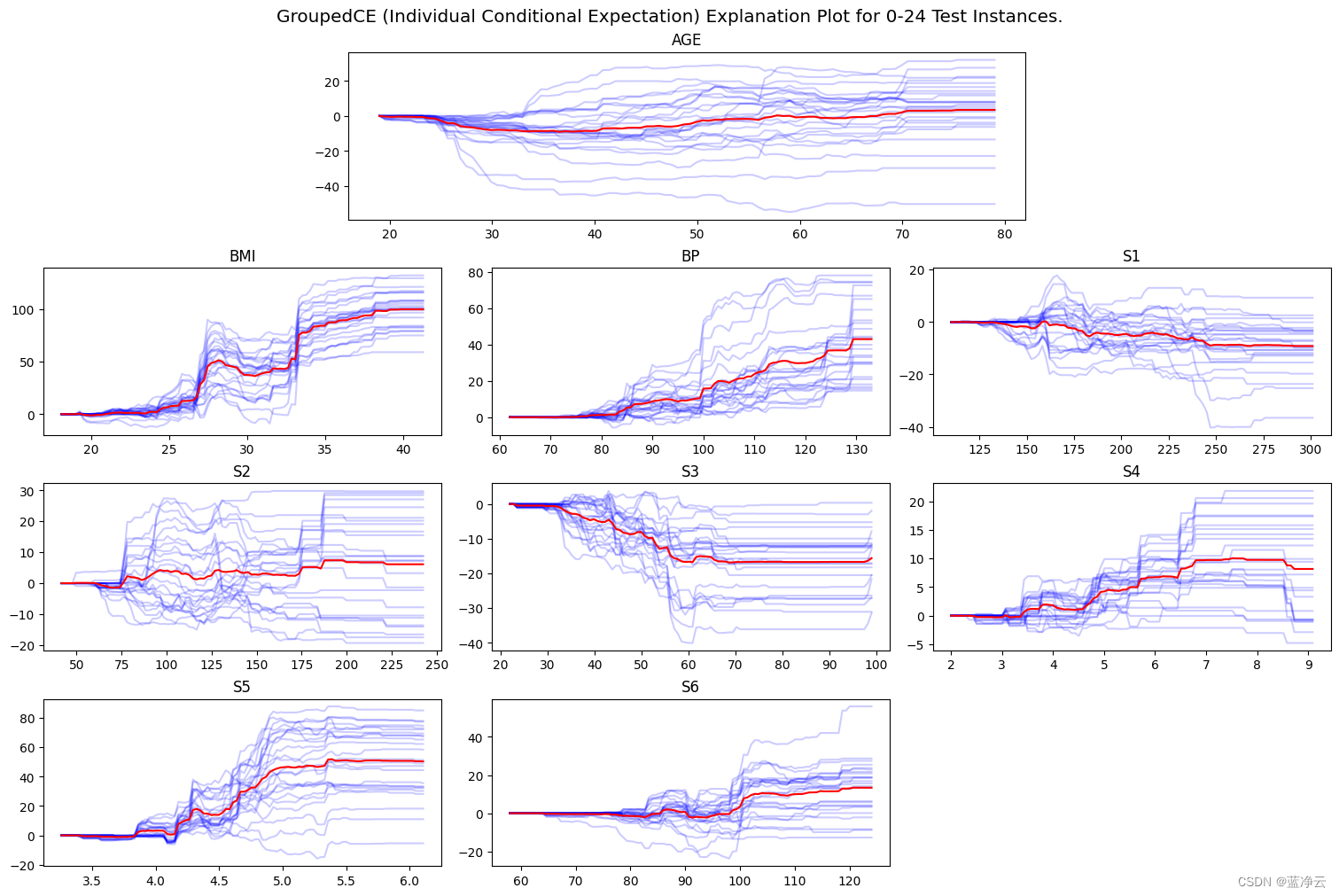
根据以上ICE图,模型预测随着BMI和BP特征的增加而线性增加。
计算分组条件期望 (GCE)
初始化GroupedCEExplainer以计算每个特征的GCE分数。该解释是为单个实例计算的局部解释。结果网格中的每个单元格都解释了特征值的组合(例如BPM和BP)如何影响模型预测。
from aix360.algorithms.gce.gce import GroupedCEExplainer
n_samples = 100
top_k_features = 4
# initialization
groupedce_explainer = GroupedCEExplainer(model=clf.predict,
data=x_train,
feature_names=feature_names,
n_samples=n_samples,
top_k_features=top_k_features,
random_seed=22)
运行结果:
Considering Top 4 features according to SHAP: ['S5', 'S3', 'BP', 'BMI']
i = 0
x_instance = x_test[[i], :]
# compute explanation
groupedce_explanation = groupedce_explainer.explain_instance(instance=x_instance)
plot_gce_explanation
plots.plot_gce_explanation具有用于绘制GroupedCE(gce)解释的辅助代码。对于不同的数据集或绘图变体,可以更新代码“plots.py”。
pip install -U kaleido
from IPython.display import Image
from plots import plot_gce_explanation
title="Grouped Conditional Expectation (GCE) Plots for Test Instance {} and Selected Features {}".format(i, groupedce_explanation['selected_features'])
fig = plot_gce_explanation(groupedce_explanation, title=title)
dataset_plot_bytes = fig.to_image(format="png", width=1000, height=1000)
Image(dataset_plot_bytes)
运行结果:
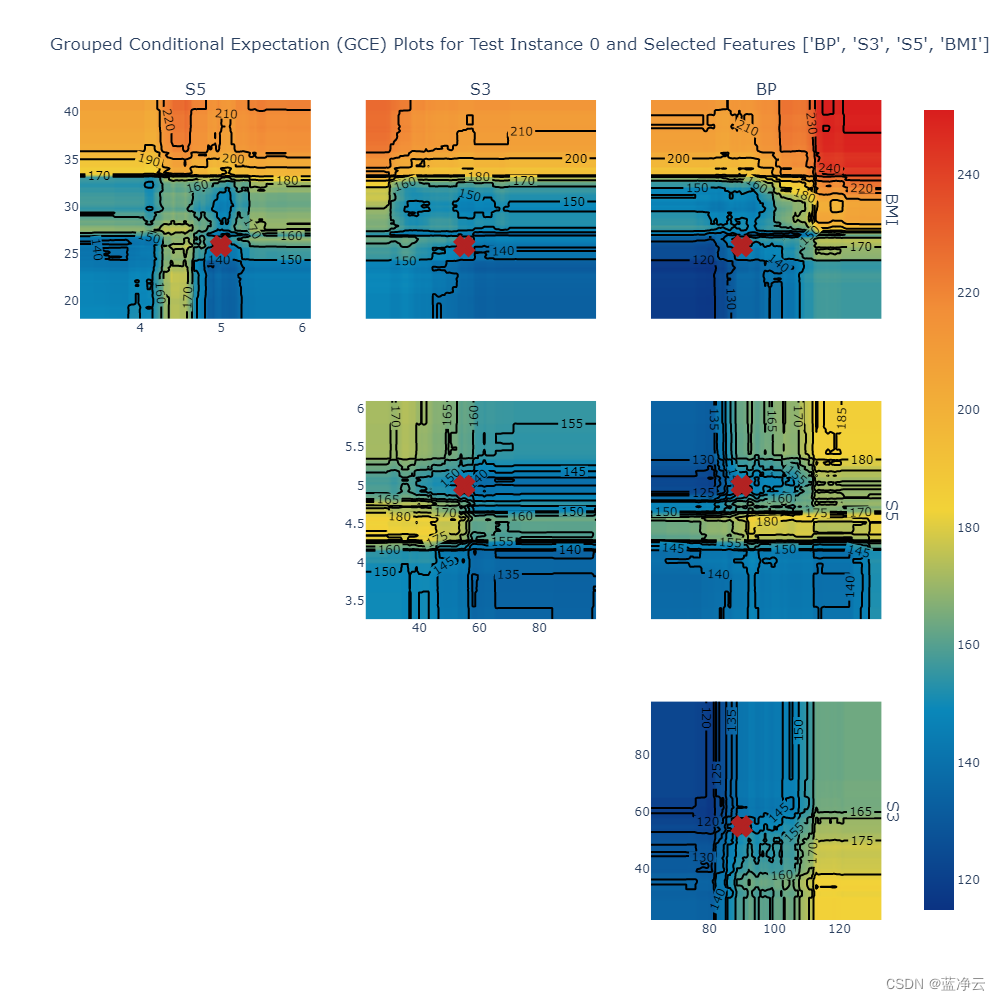
上述GCE图显示,与BP、S3和S5相比,BMI对模型预测的影响更大。