生物信息学概述
以基因组DNA序列信息为源头,识别基因组序列中代表蛋白质和RNA基因的编码区,阐明非编码区的信息特征,破译隐藏在DNA序列中的遗传语言规律
生物信息学研究的内容与方法
研究主要内容
⚫ 生物学数据的收集、存储、管理与提供
⚫ 基因组序列信息的提取和分析
⚫ 功能基因组相关信息分析
⚫ 生物大分子结构模拟和药物设计
⚫ 生物信息分析的技术与方法研究
⚫ 生物信息学方法的应用研究
一般方法
⚫ 确立研究的生物学体系。例如:特定脑血管疾病的发生和发展;蛋白质三级结构与功能;
⚫ 确定研究的问题。是否需要实验的支持?之前哪些计算方面的相关工作?
⚫ 构建生物学/数学模型,例如:蛋白质功能预测,构建模型->序列结构区域特定的功能氨基酸。
⚫ 计算方法的选择:KNN,HMM,SVM,CNN,ANN等。
⚫ 计算结果分析,构建相应的计算工具/数据库/软件/在线网站,并与同类工具做比较。
⚫ 计算工具的应用。有哪些用处?用户的反馈?
基于数据挖掘、模拟分析、机器学习
- 统计方法:外在数量表现推断事物可能的规律性
- 搜集数据(采样、实验设计)
- 分析数据(建模、知识发现)
- 推理(预测分类)
- 回归分析
- 多元回归
- 自回归
- 判别分析
- 线性判别分析
- 非线性判别分析
- 聚类分析
- 系统聚类分析
- 动态聚类分析
- 探索性分析
- 主元分析
- 相关性分析
聚类分析:对象的集合分组为由类似的对象组成的多个类的分析过程
线性判别分析LDA:监督学习的降维技术,即它的数据集的每个样本是有类别输出的
给定训练样例集
样例投影到一条直线上 -> 同类样例的投影点尽可能接
近
“投影后类内方差最小,类间方差最大”
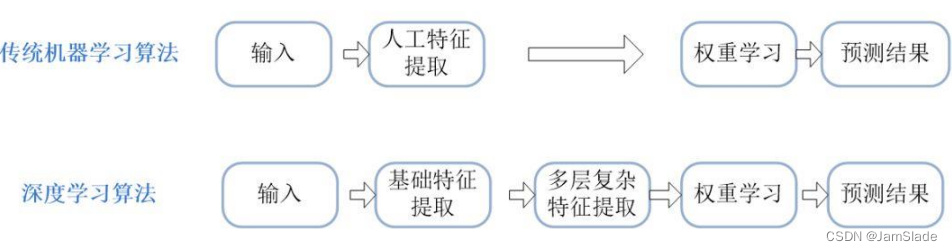
机器学习:通过执行某种过程而改进它的性能

有监督学习:问题需要答案,模型让碰到原始数据的时候贴近标准答案
分类:针对离散变量
回归:针对连续变量
应用:查找某两个指标之间的关联性
无监督学习:答案不存在或不易定义。让机器自动去发掘原有数据中间的一些特别特征或结构
聚类:样本根据某个特征分成若干群体
降维:多特征数据找寻并去除冗余性
应用:脑电信号的分析,上百组转换成十几组时序信号
深度学习 无监督学习的一种
模仿人脑神经网络的结构和某些工作机制,利用大量的神经元连成网络来实现大规模并行计算
序列分析中的主要算法
模式识别、学习算法 – 结构、功能的基于知识预测
——判别分析方法
——人工神经网络算法
——随机过程方法(HMM)
——语言学(形式语法)
——演化、遗传算法
聚类算法 – 生物大分子分类
——层次聚类算法
——模糊聚类算法
——快速动态聚类算法
——神经网络方法
系统水平
✓ 1.静态研究:路径计算、二元关系和演绎…(离散数学方法)
✓ 2.动态研究:微分方程组、Petri网模型…(网络的时间依赖演化)
难点
- 蛋白质折叠 – 氨基酸序列? 氨基酸序列预测蛋白质天然的三维结构,然而氨基酸序列并不包含结构的全部信息
- 物种重建 – DNA序列? 根据全基因组序列构建生物体的功能系统,然而基因组只是细胞中分子之间相互作用的整个网络中的一部分,可能并不包含其它的重要信息



![【精选】ARMv8/ARMv9架构入门到精通-[前言]](http://assets.processon.com/chart_image/604719347d9c082c92e419de.png)