2022-LCLR-DIFFDOCK: DIFFUSION STEPS, TWISTS, AND TURNS FOR MOLECULAR DOCKING

Paper: https://arxiv.org/abs/2210.01776
Code: https://github.com/gcorso/DiffDock
预测小分子配体与蛋白质的结合结构(称为分子对接)是药物设计的关键。最近的深度学习方法将对接视为一个回归问题,与传统的基于搜索的方法相比,它减少了运行时间,但在准确性方面还没有得到实质性的改进。相反,将分子对接构建为生成建模问题,并开发了DIFFDOCK,这是配体姿态的非欧几里得流形上的扩散生成模型。为此,将此流形映射到涉及对接的自由度(平移、旋转和扭转)的积空间,并在此空间上开发有效的扩散过程。举例来说,DIFFDOCK在PDB- Bind上获得了38%的top-1成功率(RMSD<2 a),显著优于之前最先进的传统对接(23%)和深度学习(20%)方法。此外,DIFFDOCK具有快速推断时间和提供高选择精度的置信度估计。
蛋白质的生物学功能可以通过与之结合的小分子配体(如药物)来调节。因此,计算药物设计中的一个关键任务是分子对接,预测配体与目标蛋白结合时的位置、方向和构象,从而推断配体的作用(如果有的话)。传统的对接方法[Trott & Olson, 2010;Halgren等人,2004]依赖于估计所提议结构或姿态正确性的评分函数,以及搜索评分函数的全局最大值的优化算法。然而,由于搜索空间很大,评分函数的范围很广,这些方法往往太慢,而且不准确,特别是对于高通量工作流。
近期作品[St ark et al., 2022;Lu等人,2022]开发了深度学习模型,一次性预测绑定姿势,将对接视为回归问题。虽然这些方法比传统的基于搜索的方法快得多,但它们还没有证明在准确性方面有显著提高。,这可能是因为基于回归的范式与分子对接的目标不完全对应,这反映在这样一个事实中,即标准精度指标类似于预测模型下数据的可能性,而不是回归损失。因此,将分子对接构建为给定配体和目标蛋白结构的生成建模问题,学习配体姿势的分布。
因此,作者开发了一种用于分子对接的配体空间上的扩散生成模型(DIFFDOCK)。定义了一个涉及到对接的自由度的扩散过程:配体相对于蛋白质的位置(定位结合袋),它在口袋中的方向,以及描述其构象的扭转角。DIFFDOCK样品姿态通过运行学习到的(反向)扩散过程,该过程迭代地将配体姿态上未知的、有噪声的先验分布转换为学习到的模型分布, r如下图所示
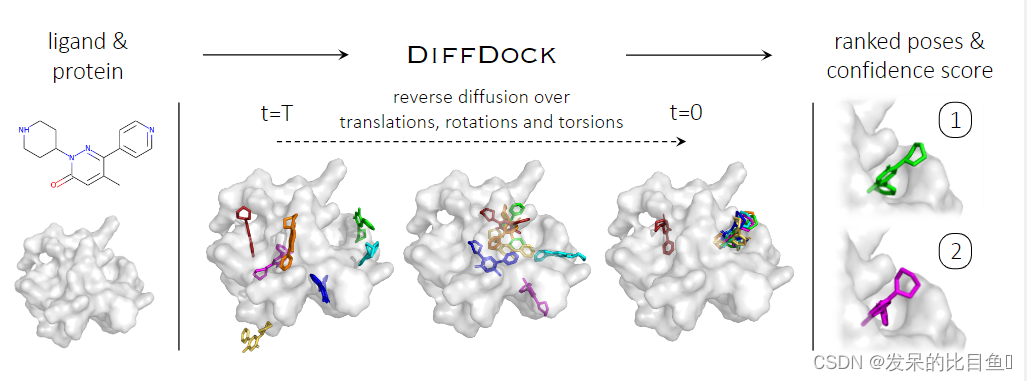 直观地说,这个过程可以被看作是通过更新它们的平移、旋转和扭转角度来逐步优化随机姿势。
直观地说,这个过程可以被看作是通过更新它们的平移、旋转和扭转角度来逐步优化随机姿势。
虽然DGMs已经应用于分子机器学习中的其他问题,但现有的方法不适用于分子对接,其中配体的空间是一个(m+6)维的子流形
M
∈
R
3
n
M \in R^{3n}
M∈R3n,其中n和M分别是原子数和扭转角。为了开发DIFFDOCK,对接自由度将M定义为通过一组允许的配体位姿转换可访问的位姿空间。使用这种思想将M中的元素映射到与这些转换相对应的组的积空间,在其中可以有效地开发和训练DGMs。由于对接模型的应用通常只需要固定数量的预测和这些预测的置信度分数,训练一个置信度模型,为从DGM中采样的姿态提供置信度估计,并挑选出最有可能的样本。
这个两步过程可以被视为蛮力搜索和一次性预测之间的一种中间方法:保留了考虑和比较多个姿势的能力,而不会遇到高维搜索的困难。
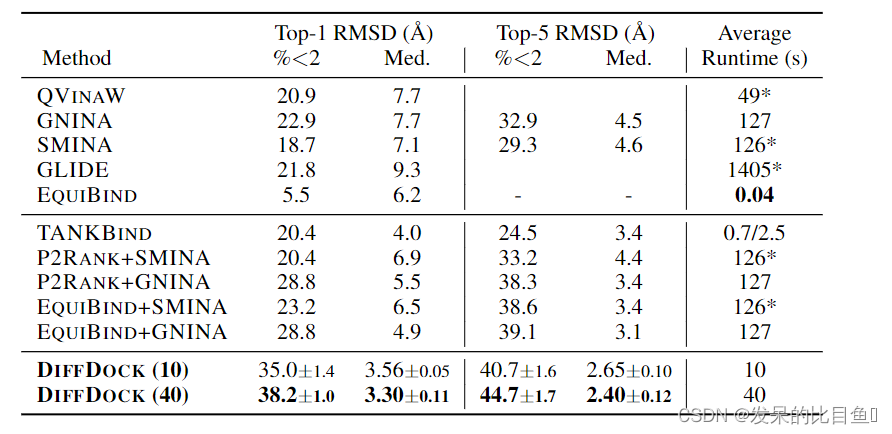
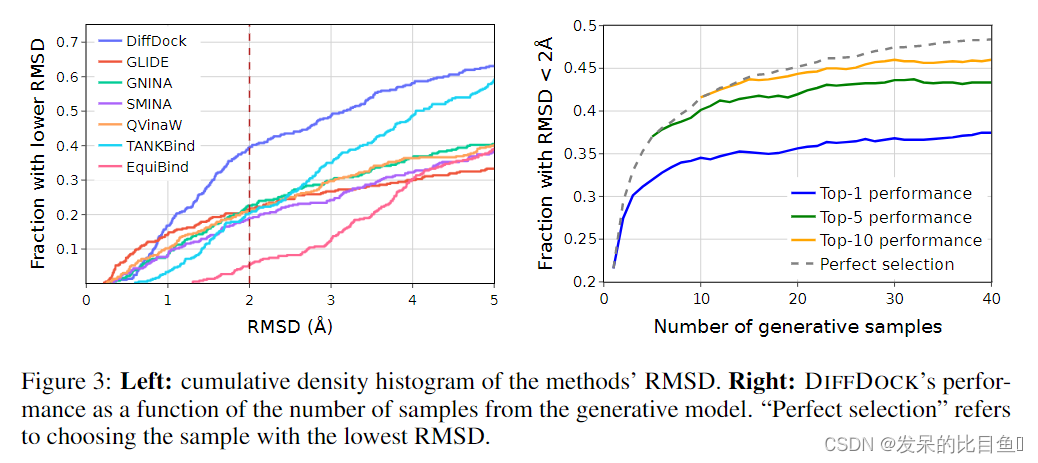
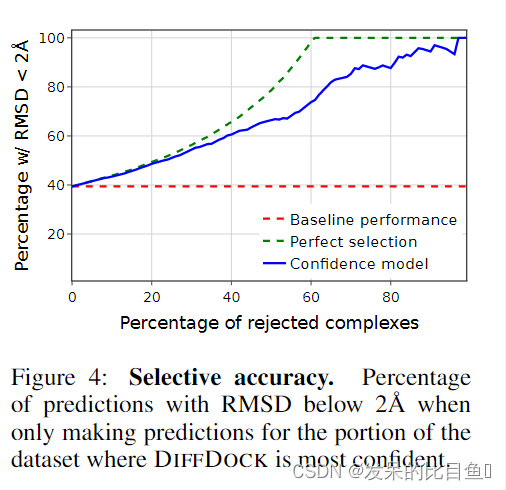
根据经验,在标准盲对接基准PDBBind上,在配体均方根距离(RMSD)低于2 A的情况下,DIFFDOCK实现了38%的top-1预测,几乎是之前最先进的深度学习模型(20%)的两倍。DIFFDOCK的性能甚至超过了最先进的基于搜索的方法(23%),同时在GPU上仍然快3到12倍。此外,它提供了其预测的准确置信度评分,在其最自信的三分之一之前未见的复合体上获得83% RMSD<2 A。
综上所述,本工作的主要贡献是:
- 将分子对接任务构建为一个生成问题,并强调了之前深度学习方法的问题。
- 提出了一个新的扩散过程的配体姿态对应的自由度涉及分子对接
- 在PDBBind盲对接基准上, R M S D < 2 A RMSD<2 A RMSD<2A,实现了最新的38%的top-1预测,大大超过了之前最好的基于搜索的方法(23%)和深度学习方法(20%)。
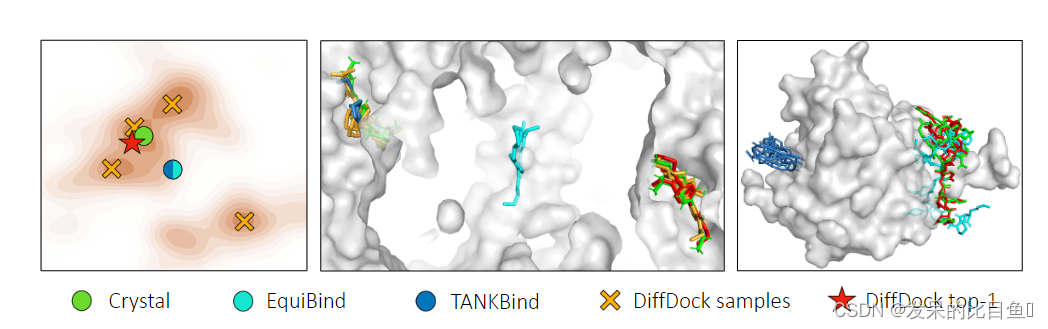
Molecular docking.









![[附源码]Python计算机毕业设计红旗家具城管理系统Django(程序+LW)](https://img-blog.csdnimg.cn/648ac3698618429da7dd4cf79e195644.png)
