DrugBAN+:Interpretable bilinear attention network with domain adaptation improves drug–target prediction
论文:Interpretable bilinear attention network with domain adaptation improves drug–target prediction | Nature Machine Intelligence
代码:https://github.com/peizhenbai/DrugBAN
数据集BioSNAP:https://github.com/kexinhuang12345/MolTrans/tree/master/dataset/BIOSNAP/full_data
一、问题
传统的基于结构和基于配体的虚拟筛选方法因其相对有效的性能而得到了广泛的研究。然而,基于结构的虚拟筛选需要分子对接模拟,如果目标蛋白的三维结构未知,则不适用。此外,基于配体的虚拟筛选基于同一蛋白质的已知活性预测新的活性分子,但当已知活性数量不足时,性能较差
DTI本质上是由药物化合物中重要分子亚结构与蛋白质序列结合位点之间的相互作用决定的。
问题1:许多以前的模型使用它们单独的编码器学习全局表示,而没有明确地学习局部交互。因此,首先学习整个结构的药物和蛋白质表示,而相互信息仅在黑箱解码模块中隐式学习。药物与靶标之间的相互作用尤其与其关键的亚结构有关; 因此,单独的全局表示学习往往会限制建模能力和预测性能。此外,如果没有对局部相互作用的明确学习,即使预测是准确的,预测结果也很难解释
问题2:跨领域推广预测性能,超越学习分布。由于化学和基因组空间的广阔区域,在实际应用中需要预测的药物靶标对通常是不可见的,并且与训练数据中的任何对都不相似。它们具有不同的分布,因此需要跨域建模。一个健壮的模型应该能够将学习到的知识转移到一个只有未标记数据的新领域。在这种情况下,需要通过学习可转移表征来对齐分布并提高跨域泛化性能
二、模型方法
1、Problem formulation
蛋白质序列:P=(a1,a2,...,an),每一个ai是23种氨基酸之一,药物分子是使用graph表示���=(V,E)。
2、DrugBAN framework

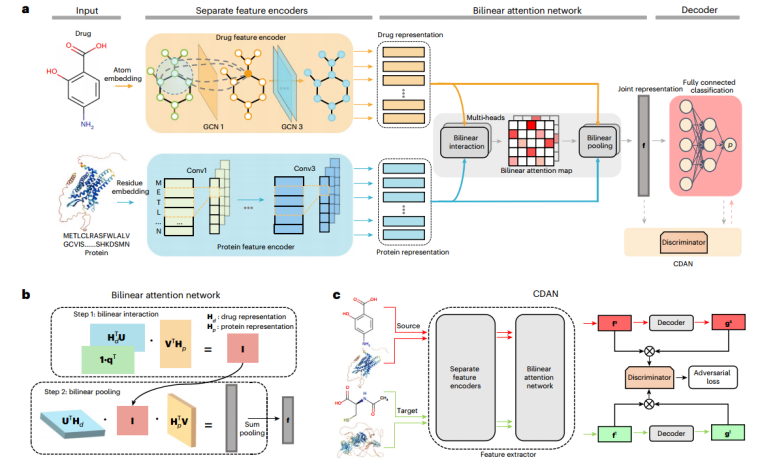
给定drug–target pair,首先分别使用GCN和1D CNN分别编码分子图和蛋白质序列信息。然后,使用双线性注意网络模块来学习编码drug和蛋白质(target)表征之间的局部相互作用。
双线性注意网络由双线性attention step和双线pooling step组成,以生成联合表示,如b所示。其次,MLP预测分数,表示交互的概率。为了提高跨域药物-目标对的模型泛化性能,进一步将CDAN嵌入到框架中,以适应表示,以便更好地对齐源和目标分布,如图c所示
Domain adaptation
减少源域和目标域之间的域分布偏移(在源域中使用标记数据,在目标域中使用未标记数据),CDAN将对抗网络与多线性条件作用相结合,用于可转移表征学习。
CNN for protein sequence
蛋白质特征编码器由三个连续的1D卷积层组成,将输入的蛋白质序列转换为潜在特征空间中的矩阵表示。氨基酸有23种,将其nn.Embedding得到初始特征矩阵Xp。超过max_length(实验中1200)的截断,短于max_length的0填充。蛋白质编码器从蛋白质特征矩阵Xp中提取局部残基模式。第一个卷积层用于捕获kernel=3的3-mer残差级特征,后两层(kernel_size=6、9,channel=128)继续扩大感受野,学习局部蛋白质片段的更抽象的特征,CNN:


GCN for molecular graph
将每个SMILES字符串转换为其二维分子图���。首先根据其化学性质初始化每个原子节点,74维,7种性质:原子类型、原子度、隐含Hs数、形式电荷、自由基电子数、原子杂化、总Hs数以及原子是否芳香族。设置了最大允许的节点数Θd(论文中290),具有较少节点的分子将包含带有零填充的虚拟节点。特征矩阵设为Md,使用简单的线性变换变成实值密集矩阵Xd,使用三层GCN来有效地学习的图表示,通过聚合节点相应的由化学键连接的邻近原子集来更新原子特征向量。这种传播机制自动捕获分子的子结构信息。因此GCN:


Pairwise interaction learning
应用双线性注意网络模块来捕捉药物和蛋白质之间的成对局部相互作用。它由两层组成: 一层是a bilinear interaction map,用于捕获两两注意权重; 另一层是interaction map上的a bilinear pooling layer,用于提取联合药物-靶标表示。
CNN和GCN得到不同的特征:

step1双线性注意层,heads=2,hidden_dim k设为768,通过变换矩阵U和V,低秩双线性交互建模得到双线性注意映射矩阵I,以测量子结构级交互强度:

可以将双线性相互作用视为首先将表示hid和hjp映射到具有权重矩阵U和V的公共特征空间,然后学习Hadamard积和向量q的权重的相互作用。这样,两两相互作用为子结构对预测结果贡献提供可解释性。
step2池化层没有新的可学习参数(pooling size s设为3)。权重矩阵U和V与前一交互映射层共享,以减少参数量,缓解过拟合:


将联合表示f输入解码器,解码器是一个完全连接的分类层(hidden size设为512),后面是一个sigmoid函数:

并使用交叉熵损失+L2正则化:

Cross-domain adaptation for better generalization
特征提取器F(⋅), 解码器 G(⋅) and 域判别器 D(⋅)
F(⋅)表示单独的feature encoders和bilinear attention network,生成输入域数据的联合表示
G(⋅)表示Decoder,全连接层:

应用一个多线性映射将联合表示F和分类器预测G嵌入到一个联合条件表示,并且Flatten:

通过在h上调节域判别器D(⋅),同时对齐联合表示并预测源域和目标域的分类分布。由三层全连接网络组成的域判别器D(⋅)学习区分联合条件表示h是来自源域还是来自目标域。相反,训练特征提取器F(⋅)和解码器G(⋅),利用源标记信息最小化源域交叉熵损失(cross-entropy loss),同时生成不可区分的表示h,混淆鉴别器D(⋅):

判别器:


看论文的话会很懵(如果没有对域适配有概念的话),判别器D(.)如何实现域适配的?
答:首先论文中的特征提取器F(.)是提取feature以供解码器G(.)和域判别器D(.)使用。对于G,最小化损失完成DTI任务;对于D,最大化域分类对抗损失(损失越大,说明D无法判别,以达到域适配的目的)
如何实现最大化对抗损失呢?向负的梯度方向优化就是最大化目标函数。

三、实验
1、Experimental setting
三个公共DTI数据集(BindingDB、BioSNAP和Human)上评估
2、Evaluation strategies and metrics
随机分为训练集、验证集和测试集,比例为7:1:2。
跨域评估:基于聚类的对分割策略(clustering-based pair split strategy):首先使用单链接算法分别通过ECFP4指纹和伪氨基酸组成(PSC)聚类药物和蛋白质。之后,从聚类结果中随机选择60%的药物簇和60%的蛋白质簇,并将所选药物和蛋白质之间的所有药物-靶标对作为源域数据。其余簇中药物和蛋白质之间的所有对都被认为是靶域数据。

评估使用AUROC、AUPRC
3、In-domain performance comparison(没有CDAN)
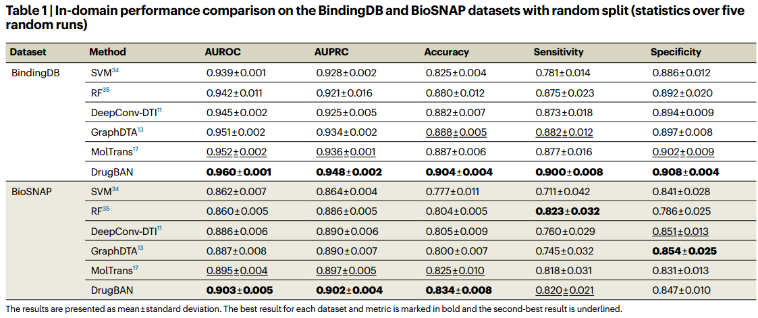
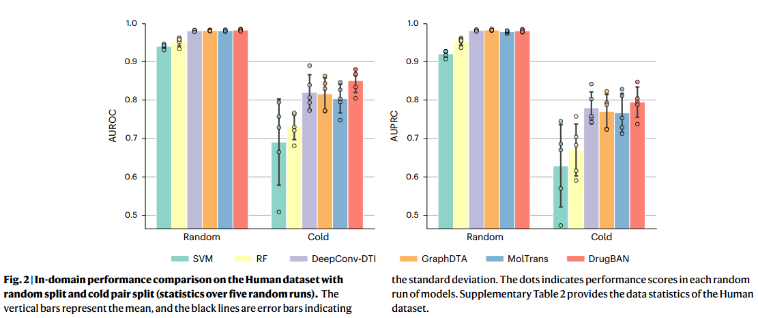
冷对分割策略保证了在训练过程中不会观察到所有的测试药物和蛋白质,从而使得对测试数据的预测不能仅仅依赖于已知药物或蛋白质的特征。所有模型的性能都有明显的下降,特别是SVM和RF,在其他最先进的深度学习基线上,DrugBAN仍然取得了最好的性能
4、Cross-domain performance comparison(+CDAN模块)
训练测试数据有不同的分布,RF在BindingDB数据集上取得了良好的性能,甚至持续优于其他深度学习基线(DeepConv, GraphDTA和MolTrans)

5、Ablation study
以探讨双线性注意和域适应模块对DrugBAN的影响:

将DrugBAN中的双线性注意替换为one-side attention,生成两种变体。
研究了两种变体:带有域对抗神经网络(DANN)的DrugBAN(即DrugBANDANN)和带有CDAN的MolTrans(即MolTransCDAN)
6、Interpretability with bilinear attention visualization
利用双线性注意图的组成部分,可视化每个子结构对最终预测结果的贡献,使分子水平的洞察和解释对药物设计工作至关重要。研究来自蛋白质数据库(PDB)的共结晶配体的前三个预测(PDB id: 6QL2,5W8L和4N6H)
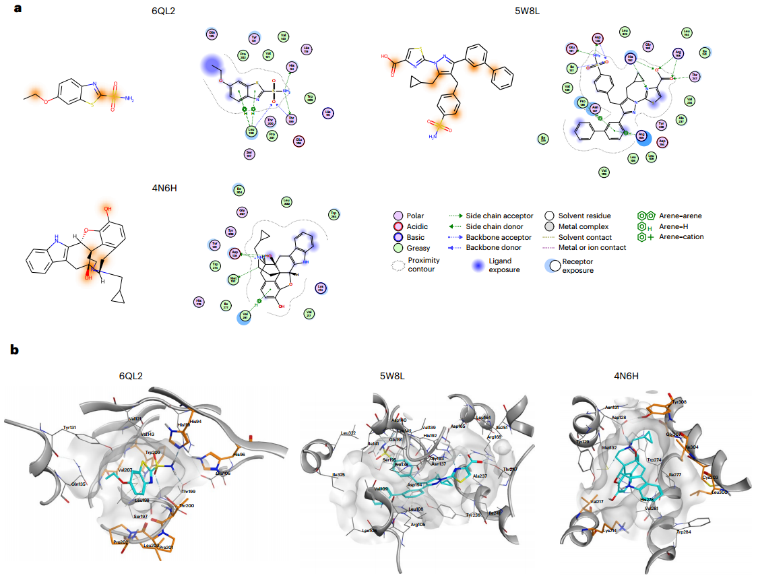
可视化结果如图4a所示,并附有相应x射线结构产生的配体-蛋白质相互作用图。对于每个分子,我们将双线性注意力图中前20%的加权原子涂成橙色。
尽管许多被预测可能参与配体结合的氨基酸残基实际上距离各自的化合物很远,但形成结合位点的一些氨基酸残基被正确预测(图4b)。例如,在6QL2复合物中,突出显示了以下残基:His94, His96, Thr200, Pro201, Pro202, Leu203, Val207和Trp209。其中,只有His94与乙氧唑胺形成特异性相互作用。在5W8L中,没有一个构成配体-蛋白结合位点的残基被突出显示。然而,在4N6H中,在结合位点内有几个正确预测的残基:Lys214、Val217、Leu300、Cys303、Ile304、Gly307和Tyr308。不幸的是,没有任何残基参与与配体的特定相互作用。
最近从100万个物种中生成了20亿个蛋白质3D结构预测。这一进展为利用3D结构信息进行基于化学基因组学的DTI预测打开了大门。(有计算资源,这就是新方向,没有计算资源,图个乐)