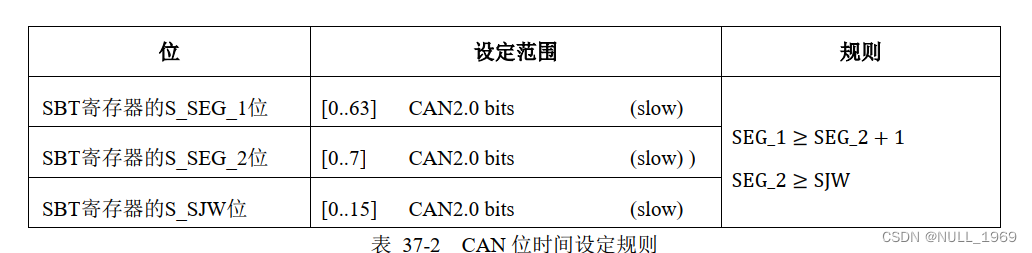
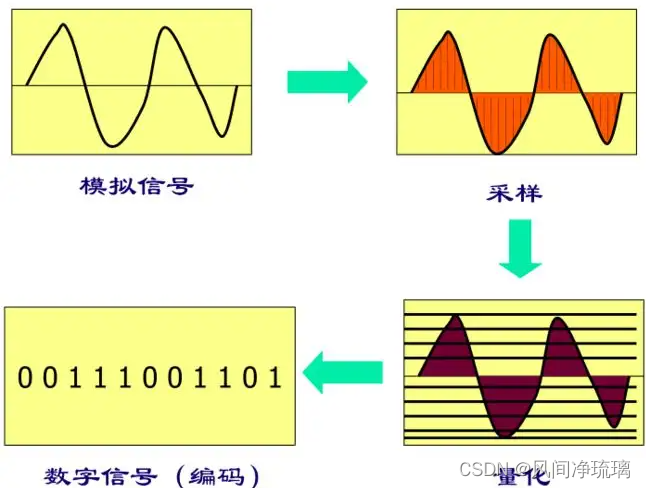
通过分析坐标轴中样本和样本间的距离可看到 2 个样本或 2 组样本间的差异性。若2个样本或2组样本之间的直线距离较近,则表示这2个样本或2组样本差异性较小;相反则表示差异性较大。因此PCA和PCoA 具有直观性(直接看两点之间的距离)和完整性(呈现所有样本),数据易于分析解读。
PCA(Principal Components Analysis),主成分分析,主分量分析或主成分回归分析法,
利用线性变换,将数据变换到新的坐标系统中,再利用降维的思想,使得任何数据投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上。减少数据集维数,并保持数据集中方差最大的特征,使数据直观呈现在二维坐标系。
PCoA(Principal Co-ordinates Analysis),主坐标分析,体现数据相似性或差异性的可视化坐标,是一种非约束性的数据降维分析方法,可研究样本组成的相似性或相异性。与PCA类似,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,其没有改变样本点之间的相互位置关系,只是改变了坐标系统。
区别:
PCA是基于样本的相似系数矩阵(如:欧式距离)检索主成分;
PCoA是基于距离矩阵(欧式距离以外的其他距离)构成主坐标。
通过生物学的样例理解PCA:
假如有3个实验样本,共有1个物种x,用物种 x 的相对丰度表示样本和样本之间的差异。画一个一维坐标轴,将这3个样本的物种 x 的丰度表示在一维轴线上,如下图所示:

此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异(样本A和B间的距离即为A中的物种 x 的丰度与 B 中物种 x 的丰度的差值)。
假如有3个实验样本,共有2个物种:x 和 y。用物种 x 和物种 y 的相对丰度来在二维坐标系中定位样本。A=(x1,y1), B=(x2,y2),C=(x3,y3),如下图所示:
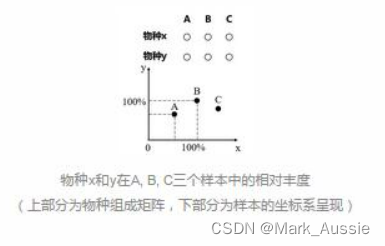
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
假如有3个实验样本,共有k个物种: x, y, z…………k。可用物种x, y, z…………k的丰度来定位样本A=(x1,y1,z1……………k1)。样本B与C也可以用这种形式表示。A=(x1,y1,z1……………k1)是一组向量,而且是k维向量(A=(x1)是一维向量,A=(x1,y1)是二维向量,A=(x1,y1,z1)是三维向量)。但是k维向量无法在二维坐标系(平面)中表示(一维和二维向量可以,如上a和b两种情况)。此时将K维向量作出一些取舍,削去一些不重要的向量仅保留2个关键向量。
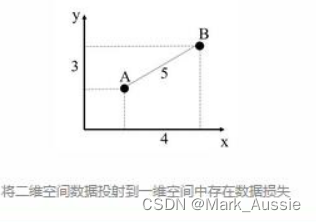
因降维处理,数据发生损失,样本和样本之间的距离代表样本之间的物种丰度差异。
通过生物学的样例理解PCoA:
假如有2个实验样本,有很多物种,用某中算法计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了2个样本的物种相对丰度的差异。画一个一维坐标轴,将这2个样本表示在一维轴线上,如下图所示:
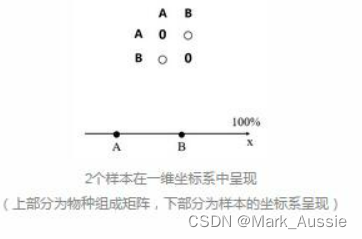
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
假如有3个实验样本,用某种算法计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了每2个样本的物种相对丰度的差异。画一个二维坐标轴(三点组成一个面),将这3个样本表示在二维轴线上,如下图所示:
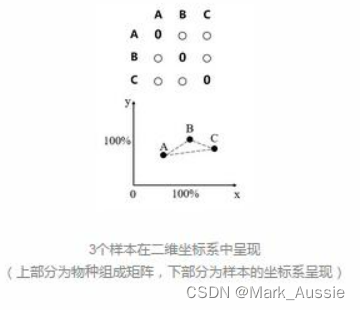
此时数据不发生偏移,样本和样本之间的距离代表样本之间的物种丰度差异。
假如有n个实验样本,用某种算法计算每个样本的物种组成差异度(用一个数值表示物种相对丰度),数值之间的差异就代表了每2个样本的物种相对丰度的差异。画一个n-1维坐标轴,将这n个样本表示在n-1维空间中。但是n-1维空间无法在平面上表示(一维和二维除外,三维勉强可以),因此只能利用矩阵呈现,如下图所示:
若要将n-1维的数据在二维坐标系中呈现,需降维处理,即将n-1维的数据投影到二维空间当中,方法与思路同PCA类似。此时,2个坐标轴的贡献率均小于100%,如下图所示:
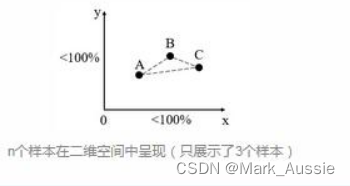
因降维处理,数据发生损失,样本和样本之间的距离代表样本之间的物种丰度差异。
“PCA是基于样本的相似系数矩阵(如欧式距离)来寻找主成分,而PCoA是基于距离矩阵(欧式距离以外的其他距离)来寻找主坐标”,其实浅显地来理解,就是上面这么回事。
参考:
三文读懂PCA和PCoA(一)




![【GO】 K8s 管理系统项目[API部分--Pod]](https://img-blog.csdnimg.cn/cc8f77562e8a4658809f8312c769b20a.png)