本文实在ubuntu下操作
DDB1-CRBN.pdb 、brd4.pdb 是需要对接的两个pdb蛋白文件
1、PatchDock
网页版:https://bioinfo3d.cs.tau.ac.il/PatchDock/php.php
PatchDock有网页版,如下;软件版需要申请获取下载链接
参考:https://bioinfo3d.cs.tau.ac.il/PatchDock/php.php
https://blog.csdn.net/weixin_42357472/article/details/127363166
下载解压后:
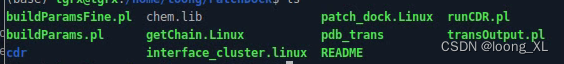
使用:
1、生成params.txt
./PatchDock/buildParams.pl DDB1-CRBN.pdb brd4.pdb
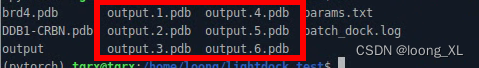
2、对接,output是对接结果
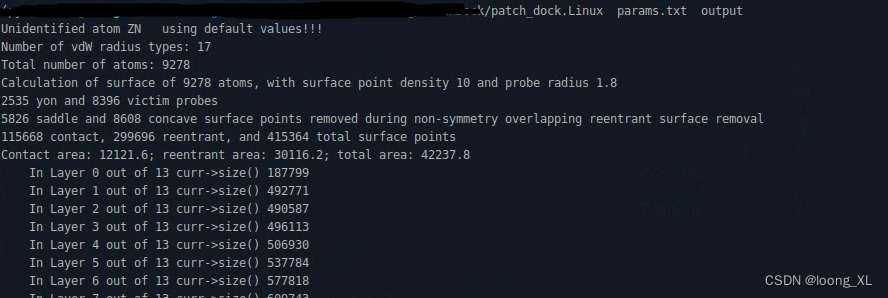
./PatchDock/patch_dock.Linux params.txt output
3、提取对接结果,从output提取前6ge
./PatchDock/transOutput.pl output 1 6
2、LightDock(python版本dock方法)
网页版:https://server.lightdock.org/job/quick/step/1
参考:https://lightdock.org/tutorials/0.9.2/simple_docking
安装: pip install lightdock
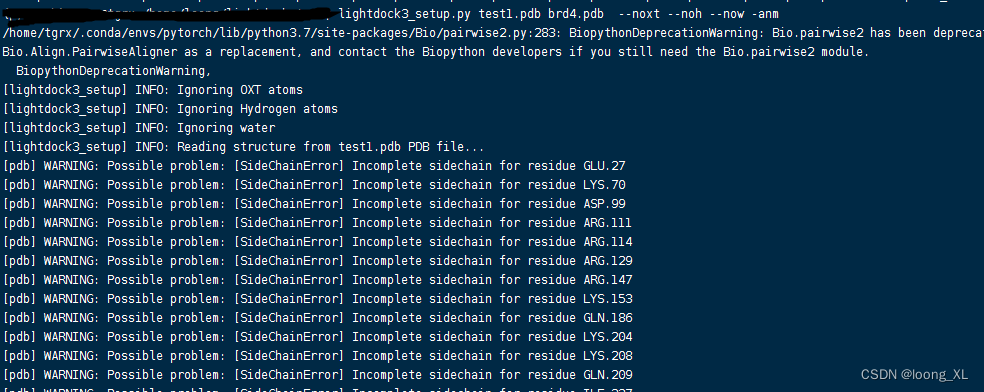
注意:LightDock软件对于输入的 pdb文件需要提取处理 删除掉含有的杂原子 HETATM;可以通过pymol处理
1、蛋白预处理,生成setup.json文件
lightdock3_setup.py test1.pdb brd4.pdb --noxt --noh --now -anm
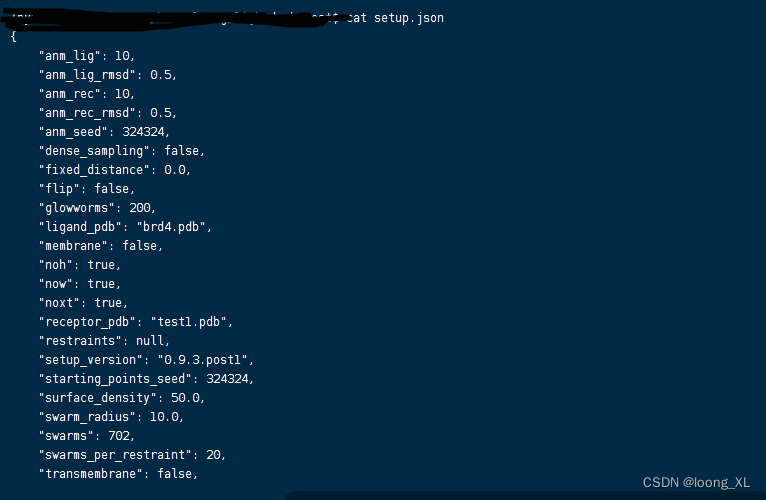
2、对接,100 step,6是cpu数量;we will only perform 100 steps on the swarm 0 with the following command;结果在swarm_0文件下
lightdock3.py setup.json 100 -c 6 -l 0
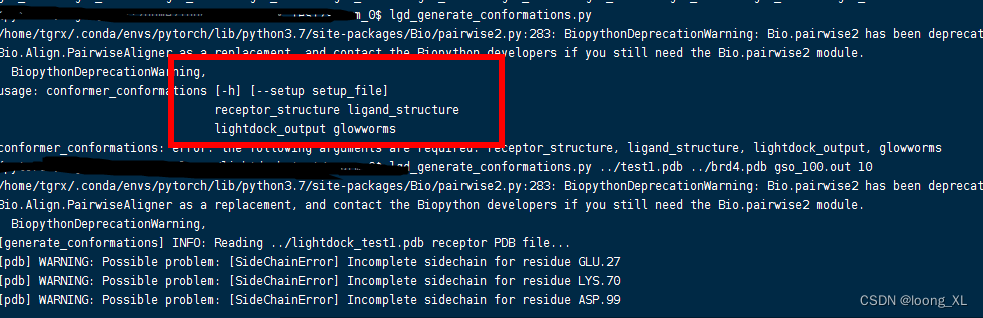
3、提取结果;从 gso_100.out 提取前10
cd swarm_0
lgd_generate_conformations.py ../test1.pdb ../brd4.pdb gso_100.out 10

![[附源码]Node.js计算机毕业设计仿咸鱼二手物品交易系统Express](https://img-blog.csdnimg.cn/c8eedbf4b6254d28b35985c2cc948e4c.png)