写在前面
今天在GitHub看到一个博主写的RNASeqTool的ShinApp,里面包含了PCA、DESeq2、volcano、NormEnrich、GSEA、Gene tred analysis和WGCNA分析。使用后还是很方便的,就此推荐给大家。感兴趣可以自己操作即可。
GitHub网址
https://github.com/ChaoXu1997/RNAseqTool
若你无法下载此安装包,可回复:20230804获得

使用方法
直接安装到服务器R环境中
# Install devtools
install.packages("devtools")
# Install BiocManager
install.packages("BiocManager")
# Install dependent R packages from Bioconductor
pkgs <- c('clusterProfiler', 'DESeq2', 'Mfuzz')
lapply(pkgs,function(pkg){
if (!require(pkg, quietly = TRUE))
BiocManager::install(pkg,update = F)
})
# Install dependent R packages from GitHub
devtools::install_github("vqv/ggbiplot")
devtools::install_github("junjunlab/GseaVis")
# Install RNAseqTool
devtools::install_github("ChaoXu1997/RNAseqTool")
# Run ShinyApp
RNAseqTool::run_app()
# Remove RNAseqTool
remove.packages("RNAseqTool")
本地运行
本地运行,安装类似
# Install RNAseqTool
devtools::install_github("ChaoXu1997/RNAseqTool")
# Run ShinyApp
RNAseqTool::run_app()
# Remove RNAseqTool
remove.packages("RNAseqTool")
或是直接下载安装包后,直接运行。
# Launch the ShinyApp (Do not remove this comment)
# To deploy, run: rsconnect::deployApp()
# Or use the blue button on top of this file
#install.packages("GseaVis")
#install.packages("ggbiplot")
setwd("E:\\小杜的生信筆記\\2023\\20230804_转录组下游分析\\RNAseqTool-master")
pkgload::load_all(export_all = FALSE,helpers = FALSE,attach_testthat = FALSE)
options( "golem.app.prod" = TRUE)
RNAseqTool::run_app() # add parameters here (if any)
若遇到缺少相关包,安装即可。
运行界面
R自动弹出界面
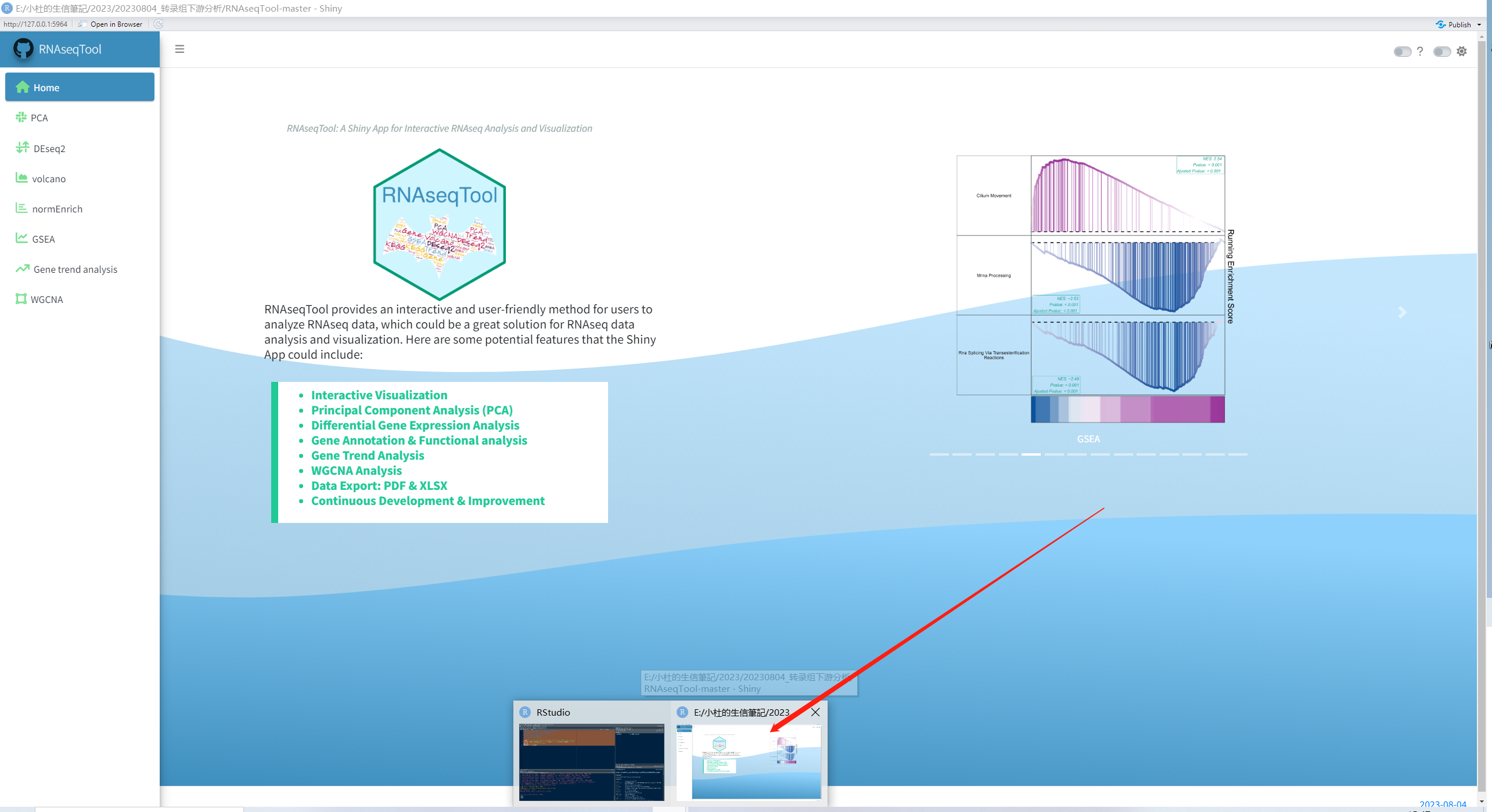
复制相应的网址,浏览器打开
运行
开发者,给了对应的事例数据。若使用时,不知数据类型,可以自己下载事例数据,进行数据准备。
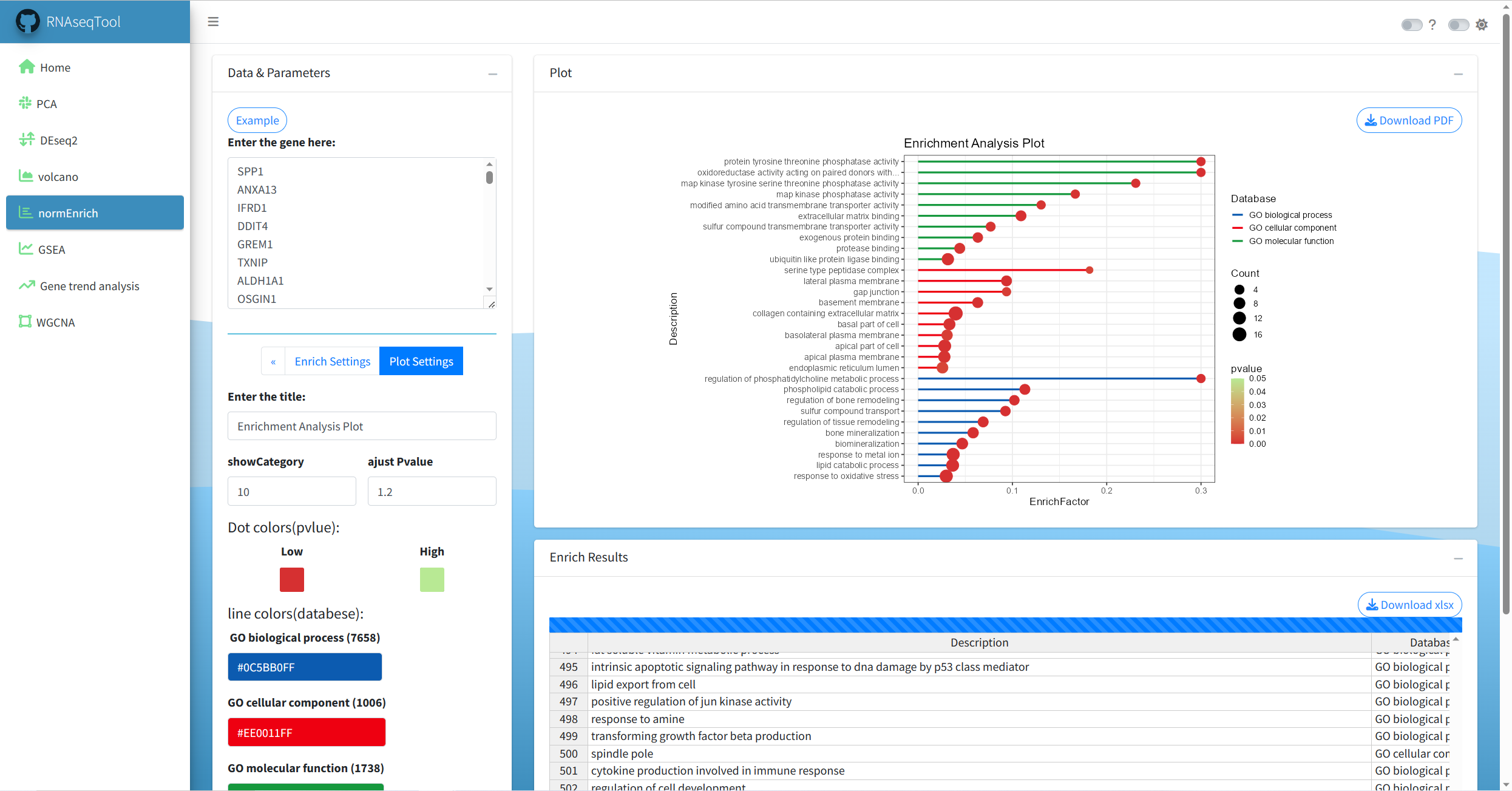
动态教程
开发者也给了几个动态了教程,但是时间较短,具体参数还需自己操作。
…等等(自己查看)
ENDING !!!
最后,感谢开发者的开发,对转录组下游分析的同学应该会有一定的帮助。希望后续,可以添加更多的分析。
往期文章:
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
-
WGCNA分析 | 全流程分析代码 | 代码一
-
WGCNA分析 | 全流程分析代码 | 代码二
-
WGCNA分析 | 全流程代码分享 | 代码三
2. 精美图形绘制教程
- 精美图形绘制教程
3. 转录组分析教程
腾讯课堂: https://bioinformatic.ke.qq.com/
CCtalk: https://www.cctalk.com/m/group/90864710
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!