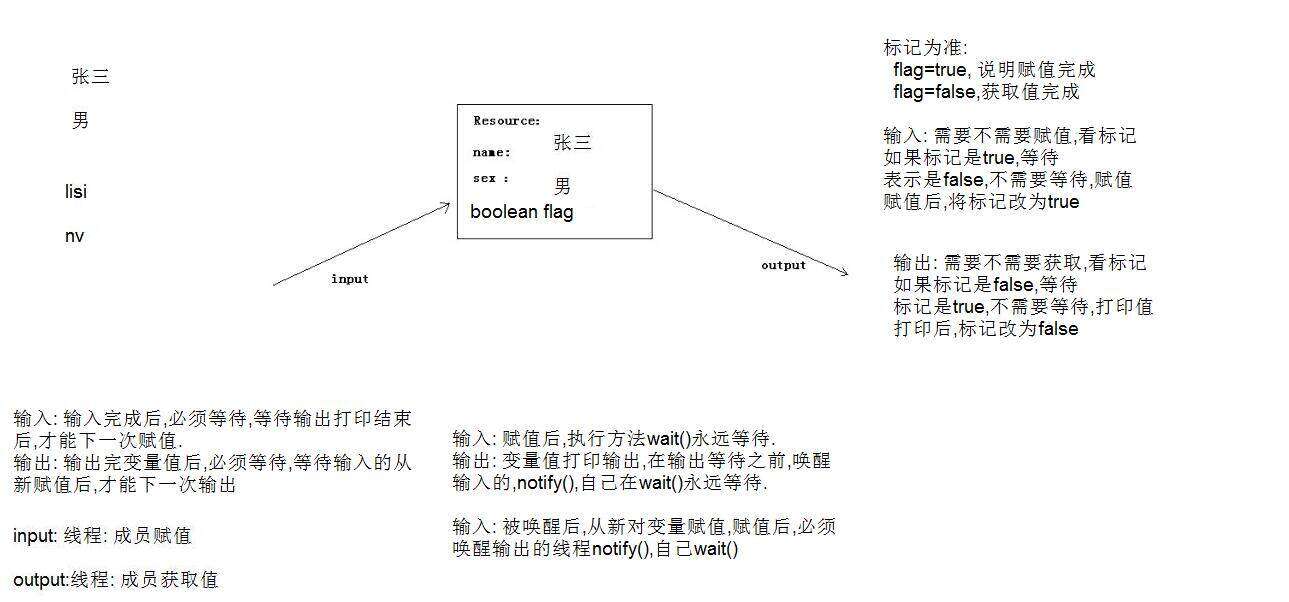
一、ChatGPT结合知识图谱
在本专栏的前面文章中构建 ChatGPT 本地知识库问答应用,都是基于词向量检索 Embedding 嵌入的方式实现的,在传统的问答领域中,一般知识源采用知识图谱来进行构建,但基于知识图谱的问答对于自然语言的处理却需要耗费大量的人力和物力,而 ChatGPT 本身就拥有强大的自然语言处理能力,如果将ChatGPT和知识图谱相结合岂不是更加强大,本文和下篇文章探索将 ChatGPT结合知识图谱,构建一个基于医疗问答场景的应用。
什么是知识图谱:
知识图谱是一种用于表示和组织结构化知识的图形化模型。它是一种利用图论和语义网络的技术,旨在捕捉信息之间的关联性和语义含义。在问答领域,知识图谱发挥着重要作用。
首先,知识图谱以实体和关系的形式存储知识。实体代表现实世界中的具体事物,例如人、地点、事件等,而关系则描述这些实体之间的联系,例如居住在、发生在等。通过将实体和关系连接为节点和边,知识图谱能够形成一个复杂的网络,反映出知识之间的复杂关系。
其次,知识图谱通过为每个实体和关系添加语义标签,使得计算机能够理解和推理这些知识。这意味着知识图谱能够帮助机器理解实体之间的语义关系,从而回答用户提出的问题。例如,当用户询问“谁是美国第一位总统?”时,知识图谱可以识别到实体“美国”和“总统”,并根据关系“第一位”来回答这个问题。
知识图谱采用经典的 neo4j 图数据来进行构建,在实验前请安装好 neo4j 环境:
二、医疗数据集
医疗数据集,采用 github 上 刘焕勇老师 分享的数据集,下载地址:
https://github.com/wangle1218/QASystemOnMedicalKG/blob/master/data/medical.json
数据为 JSON 格式,示例如下:
{
"_id":{
"$oid":"5bb578b6831b973a137e3ee6"
},
"name":"肺泡蛋白质沉积症",
"desc":"肺泡蛋白质沉积症(简称PAP),又称Rosen-Castle-man-Liebow综合征,是一种罕见疾病。该病以肺泡和细支气管腔内充满PAS染色阳性,来自肺的富磷脂蛋白质物质为其特征,好发于青中年,男性发病约3倍于女性。",
"category":[
"疾病百科",
"内科",
"呼吸内科"
],
"prevent":"1、避免感染分支杆菌病,卡氏肺囊肿肺炎,巨细胞病毒等。\n2、注意锻炼身体,提高免疫力。",
"cause":"病因未明,推测与几方面因素有关:如大量粉尘吸入(铝,二氧化硅等),机体免疫功能下降(尤其婴幼儿),遗传因素,酗酒,微生物感染等,而对于感染,有时很难确认是原发致病因素还是继发于肺泡蛋白沉着症,例如巨细胞病毒,卡氏肺孢子虫,组织胞浆菌感染等均发现有肺泡内高蛋白沉着。\n虽然启动因素尚不明确,但基本上同意发病过程为脂质代谢障碍所致,即由于机体内,外因素作用引起肺泡表面活性物质的代谢异常,到目前为止,研究较多的有肺泡巨噬细胞活力,动物实验证明巨噬细胞吞噬粉尘后其活力明显下降,而病员灌洗液中的巨噬细胞内颗粒可使正常细胞活力下降,经支气管肺泡灌洗治疗后,其肺泡巨噬细胞活力可上升,而研究未发现Ⅱ型细胞生成蛋白增加,全身脂代谢也无异常,因此目前一般认为本病与清除能力下降有关。",
"symptom":[
"紫绀",
"胸痛",
"呼吸困难",
"乏力",
"毓卓"
],
"yibao_status":"否",
"get_prob":"0.00002%",
"get_way":"无传染性",
"acompany":[
"多重肺部感染"
],
"cure_department":[
"内科",
"呼吸内科"
],
"cure_way":[
"支气管肺泡灌洗"
],
"cure_lasttime":"约3个月",
"cured_prob":"约40%",
"cost_money":"根据不同医院,收费标准不一致,省市三甲医院约( 8000——15000 元)",
"check":[
"胸部CT检查",
"肺活检",
"支气管镜检查"
],
"recommand_drug":[
],
"drug_detail":[
]
}
其中数据集中常用的字段解释:
| 字段 | 说明 |
|---|---|
| name | 疾病名称 |
| desc | 疾病简介 |
| category | 分类 |
| prevent | 预防措施 |
| cause | 疾病病因 |
| symptom | 疾病症状 |
| yibao_status | 是否支持医保 |
| get_prob | 发病率 |
| get_way | 传染性 |
| acompany | 并发症 |
| cure_department | 医疗科目 |
| cure_way | 治疗方式 |
| cure_lasttime | 治疗周期 |
| cured_prob | 治愈概率 |
| cost_money | 大概花费 |
| check | 诊断检查项目 |
| recommand_drug | 建议用药 |
| drug_detail | 药物详细信息 |
| easy_get | 疾病易感人群 |
| not_eat | 不适宜吃的食物 |
| recommand_eat | 建议吃的食物 |
| common_drug | 一般用药 |
三、知识图谱结构规划
由于数据集主要是围绕病症来衍生的,因此除了疾病的属性会多些其余均是为建立关系而创建。
3.1 实体规划
疾病实体(disease)
| 字段 | 说明 |
|---|---|
| name | 疾病名称 |
| desc | 疾病简介 |
| prevent | 预防措施 |
| cause | 疾病病因 |
| get_prob | 发病率 |
| get_way | 传染性 |
| cure_lasttime | 治疗周期 |
| cured_prob | 治愈概率 |
| cost_money | 大概花费 |
科室实体(department)
| 字段 | 说明 |
|---|---|
| name | 科室名称 |
疾病症状实体 (symptom)
| 字段 | 说明 |
|---|---|
| name | 疾病症状 |
治疗方式实体 (cureWay)
| 字段 | 说明 |
|---|---|
| name | 治疗方式 |
检查项目实体 (check)
| 字段 | 说明 |
|---|---|
| name | 检查项目 |
用药药物实体 (drug)
| 字段 | 说明 |
|---|---|
| name | 药物名称 |
易感染人群实体 (crowd)
| 字段 | 说明 |
|---|---|
| name | 感染人群 |
食物实体 (food)
| 字段 | 说明 |
|---|---|
| name | 食物 |
3.2 关系规划
| 开始实体 | 关系 | 结束实体 |
|---|---|---|
| 疾病(disease) | 疾病科室关系 (diseaseDepartmentRelations) | 科室实体(department) |
| 疾病(disease) | 疾病症状关系 (diseaseSymptomRelation) | 疾病症状实体 (symptom) |
| 疾病(disease) | 疾病治疗关系 (diseaseCureWayRelation) | 治疗方式实体 (cureWay) |
| 疾病(disease) | 疾病检查项目关系 (diseaseCheckRelation) | 检查项目实体 (check) |
| 疾病(disease) | 疾病用药关系 (diseaseDrugRelation) | 药物实体 (drug) |
| 疾病(disease) | 疾病易感染人群关系 (diseaseCrowdRelation) | 易感染人群实体 (crowd) |
| 疾病(disease) | 疾病宜吃食物关系 (diseaseSuitableFoodRelation) | 食物实体 (food) |
| 疾病(disease) | 疾病忌吃食物关系 (diseaseTabooFoodRelation) | 食物实体 (food) |
| 疾病(disease) | 疾病并发症关系 (diseaseDiseaseRelation) | 疾病(disease) |
四、知识图谱构建
在里采用 Python 语言构建,需要安装 py2neo 库:
pip install py2neo -i https://pypi.tuna.tsinghua.edu.cn/simple
from py2neo import Graph
import os
from tqdm import tqdm
import json
class CreateKG():
def __init__(self, kg_host, kg_port, kg_user, kg_password, data_path):
self.graph = Graph(
host=kg_host,
http_port=kg_port,
user=kg_user,
password=kg_password)
if not data_path or data_path == '':
raise Exception("数据集地址为空")
if not os.path.exists(data_path):
raise Exception("数据集不存在")
self.data_path = data_path
def saveEntity(self, label, data):
print("\n写入实体:", label)
for item in tqdm(data, ncols=80):
try:
property = []
for key, value in item.items():
value = value.replace("'", "")
property.append(key + ":" + "'" + value + "'")
if len(property) == 0:
continue
cql = "MERGE(n:" + label + "{" + ",".join(property) + "})"
self.graph.run(cql)
except Exception as e:
pass
def saveRelation(self, s_label, e_label, label, data):
print("\n写入关系:", label)
for item in tqdm(data, ncols=80):
try:
s_name = item["s_name"]
e_name = item["e_name"]
cql = "MATCH(p:" + s_label + "),(q:" + e_label + ") WHERE p.name='" + s_name + "' AND q.name='" + e_name + "' MERGE (p)-[r:" + label + "]->(q)"
self.graph.run(cql)
except Exception as e:
pass
def getValue(self, key, data):
if key in data:
return data[key]
return ""
def init(self):
# 实体
# 疾病
diseases = []
# 科室
departments = []
# 疾病症状
symptoms = []
# 治疗方式
cureWays = []
# 检查项目
checks = []
# 药物
drugs = []
# 易感染人群
crowds = []
# 食物
foods = []
# 关系
# 疾病科室
diseaseDepartmentRelations = []
# 疾病症状
diseaseSymptomRelations = []
# 疾病治疗
diseaseCureWayRelations = []
# 疾病检查
diseaseCheckRelations = []
# 疾病用药
diseaseDrugRelations = []
# 疾病易感染人群
diseaseCrowdRelations = []
# 疾病宜吃食物
diseaseSuitableFoodRelations = []
# 疾病忌吃食物
diseaseTabooFoodRelations = []
# 疾病并发症
diseaseDiseaseRelations = []
print("====数据抽取======")
with open(self.data_path, 'r', encoding='utf8') as f:
for line in tqdm(f.readlines(), ncols=80):
data = json.loads(line)
# 疾病实体
disease = {
"name": data["name"],
"desc": self.getValue("desc", data),
"prevent": self.getValue("prevent", data),
"cause": self.getValue("cause", data),
"get_prob": self.getValue("get_prob", data),
"get_way": self.getValue("get_way", data),
"cure_lasttime": self.getValue("cure_lasttime", data),
"cured_prob": self.getValue("cured_prob", data),
"cost_money": self.getValue("cost_money", data),
}
diseases.append(disease)
# 科室
if "cure_department" in data:
for department in data["cure_department"]:
# 疾病科室关系
diseaseDepartmentRelations.append({
"s_name": data["name"],
"e_name": department
})
# 科室实体
property = {
"name": department
}
if property not in departments:
departments.append(property)
# 症状
if "symptom" in data:
for symptom in data["symptom"]:
# 疾病科室关系
diseaseSymptomRelations.append({
"s_name": data["name"],
"e_name": symptom
})
# 症状实体
property = {
"name": symptom
}
if property not in symptoms:
symptoms.append(property)
# 治疗方式
if "cure_way" in data:
for cure_way in data["cure_way"]:
# 疾病科室关系
diseaseCureWayRelations.append({
"s_name": data["name"],
"e_name": cure_way
})
# 治疗方式实体
property = {
"name": cure_way
}
if property not in cureWays:
cureWays.append(property)
# 检查项目
if "check" in data:
for check in data["check"]:
# 疾病科室关系
diseaseCheckRelations.append({
"s_name": data["name"],
"e_name": check
})
# 检查项目实体
property = {
"name": check
}
if property not in checks:
checks.append(property)
# 一般用药
if "common_drug" in data:
for common_drug in data["common_drug"]:
# 疾病科室关系
diseaseDrugRelations.append({
"s_name": data["name"],
"e_name": common_drug
})
# 用药实体
property = {
"name": common_drug
}
if property not in drugs:
drugs.append(property)
# 易感染人群
if "easy_get" in data:
easy_get = data["easy_get"]
# 疾病科室关系
diseaseCrowdRelations.append({
"s_name": data["name"],
"e_name": easy_get
})
# 易感染人群实体
property = {
"name": easy_get
}
if property not in crowds:
crowds.append(property)
# 宜吃食物
if "recommand_eat" in data:
for recommand_eat in data["recommand_eat"]:
# 疾病科室关系
diseaseSuitableFoodRelations.append({
"s_name": data["name"],
"e_name": recommand_eat
})
# 食物实体
property = {
"name": recommand_eat
}
if property not in foods:
foods.append(property)
# 忌吃食物
if "not_eat" in data:
for not_eat in data["not_eat"]:
# 疾病科室关系
diseaseTabooFoodRelations.append({
"s_name": data["name"],
"e_name": not_eat
})
# 食物实体
property = {
"name": not_eat
}
if property not in foods:
foods.append(property)
# 并发症
if "acompany" in data:
for acompany in data["acompany"]:
# 疾病科室关系
diseaseDiseaseRelations.append({
"s_name": data["name"],
"e_name": acompany
})
# 疾病
self.saveEntity("disease", diseases)
# 科室
self.saveEntity("department", departments)
# 疾病症状
self.saveEntity("symptom", symptoms)
# 治疗方式
self.saveEntity("cureWay", cureWays)
# 检查项目
self.saveEntity("check", checks)
# 药物
self.saveEntity("drug", drugs)
# 易感染人群
self.saveEntity("crowd", crowds)
# 食物
self.saveEntity("food", foods)
# 关系
# 疾病科室
self.saveRelation("disease", "department", "diseaseDepartmentRelations", diseaseDepartmentRelations)
# 疾病症状
self.saveRelation("disease", "symptom", "diseaseSymptomRelation", diseaseSymptomRelations)
# 疾病治疗
self.saveRelation("disease", "cureWay", "diseaseCureWayRelation", diseaseCureWayRelations)
# 疾病检查
self.saveRelation("disease", "check", "diseaseCheckRelation", diseaseCheckRelations)
# 疾病用药
self.saveRelation("disease", "drug", "diseaseDrugRelation", diseaseDrugRelations)
# 疾病易感染人群
self.saveRelation("disease", "crowd", "diseaseCrowdRelation", diseaseCrowdRelations)
# 疾病宜吃食物
self.saveRelation("disease", "food", "diseaseSuitableFoodRelation", diseaseSuitableFoodRelations)
# 疾病忌吃食物
self.saveRelation("disease", "food", "diseaseTabooFoodRelation", diseaseTabooFoodRelations)
# 疾病并发症
self.saveRelation("disease", "disease", "diseaseDiseaseRelation", diseaseDiseaseRelations)
if __name__ == '__main__':
kg_host = "127.0.0.1"
kg_port = 7474
kg_user = "neo4j"
kg_password = "123456"
data_path = "./data/medical.json"
kg = CreateKG(kg_host, kg_port, kg_user, kg_password, data_path)
kg.init()
运行之后可以看到处理的进度:
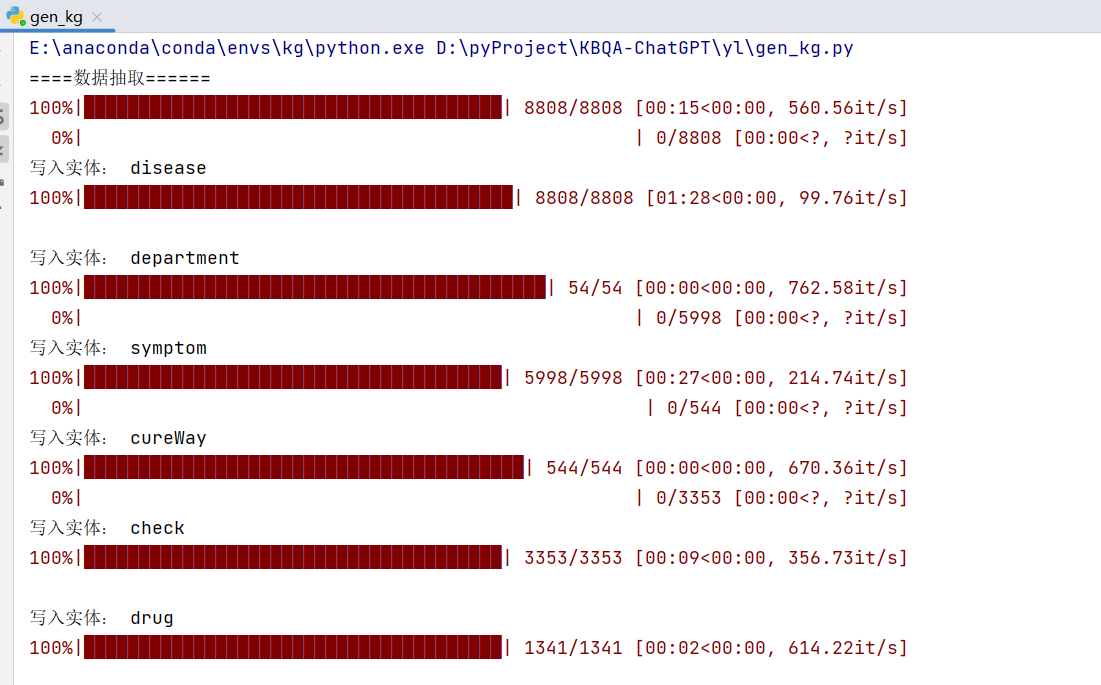
等待处理结束后就可以在图谱中看到构建后的效果了:
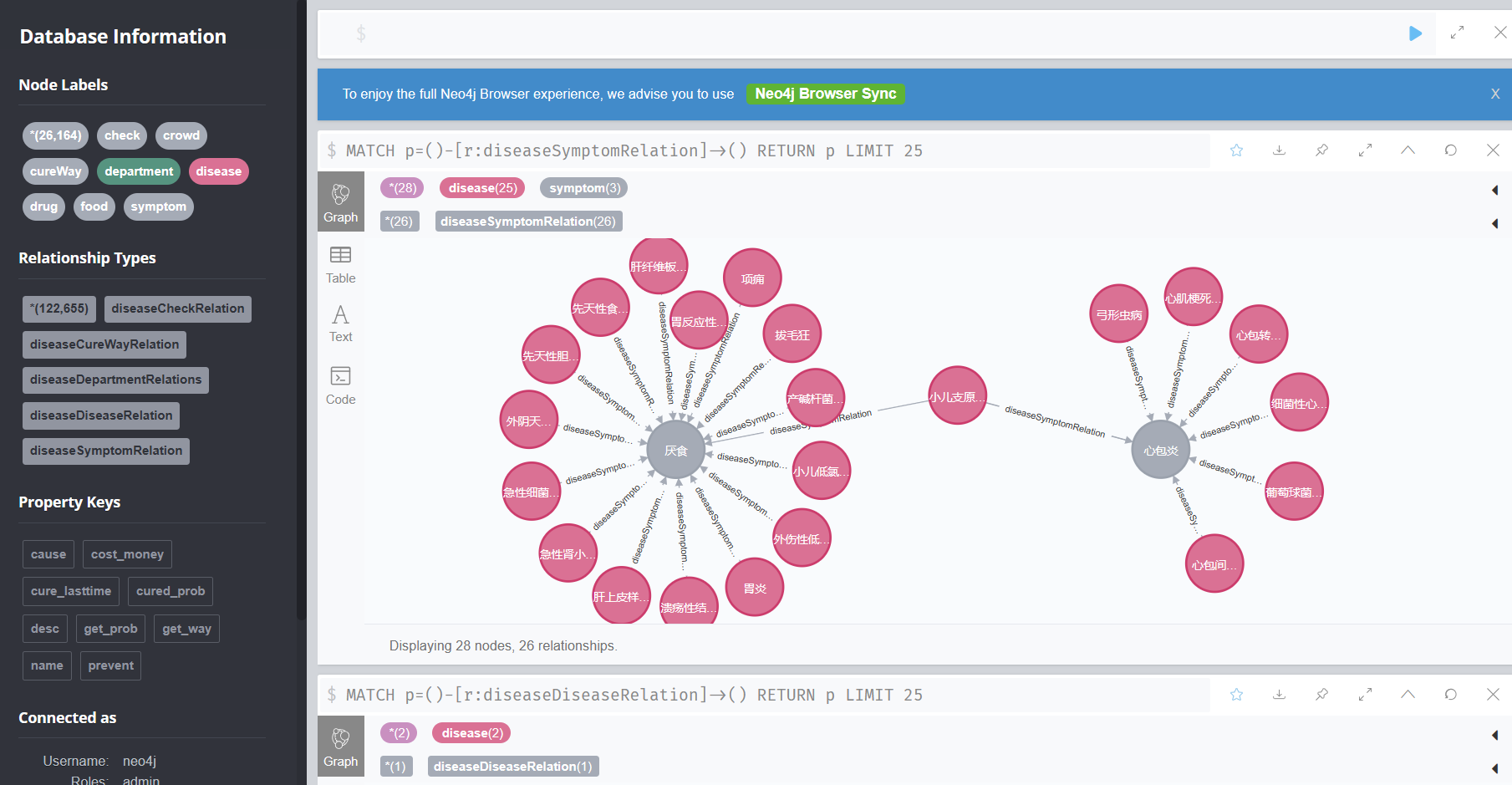
五、数据探索测试
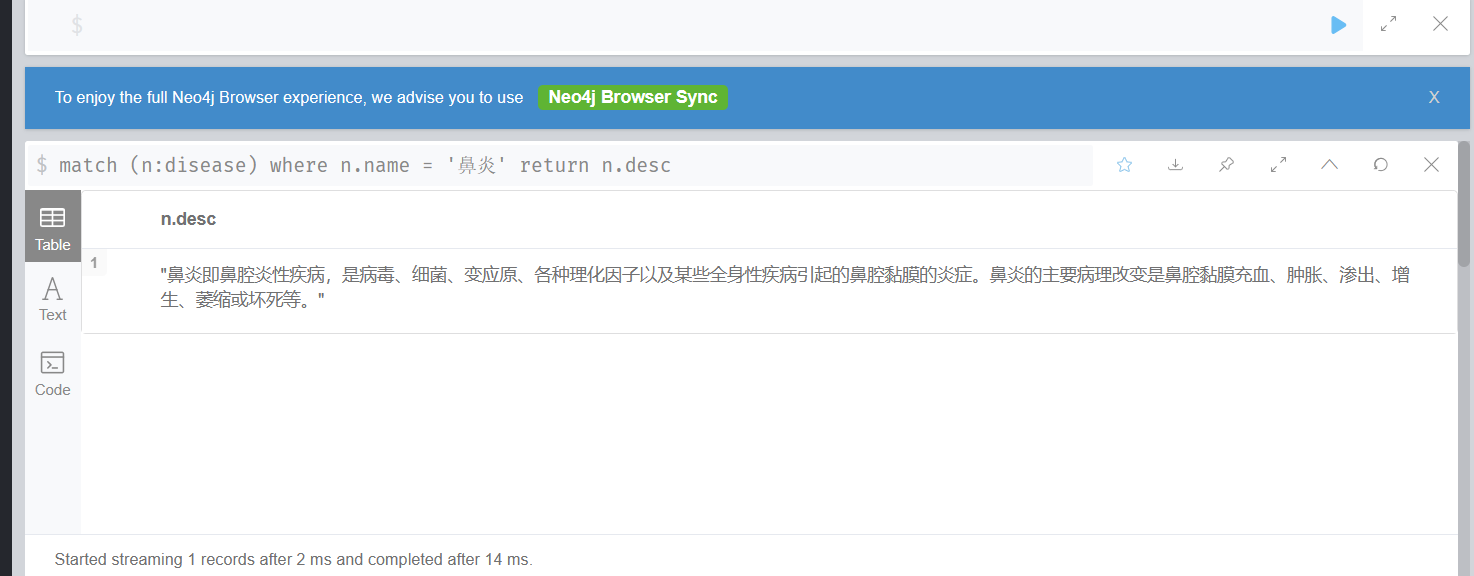
鼻炎的病症描述:
match (n:disease) where n.name = '鼻炎' return n.desc
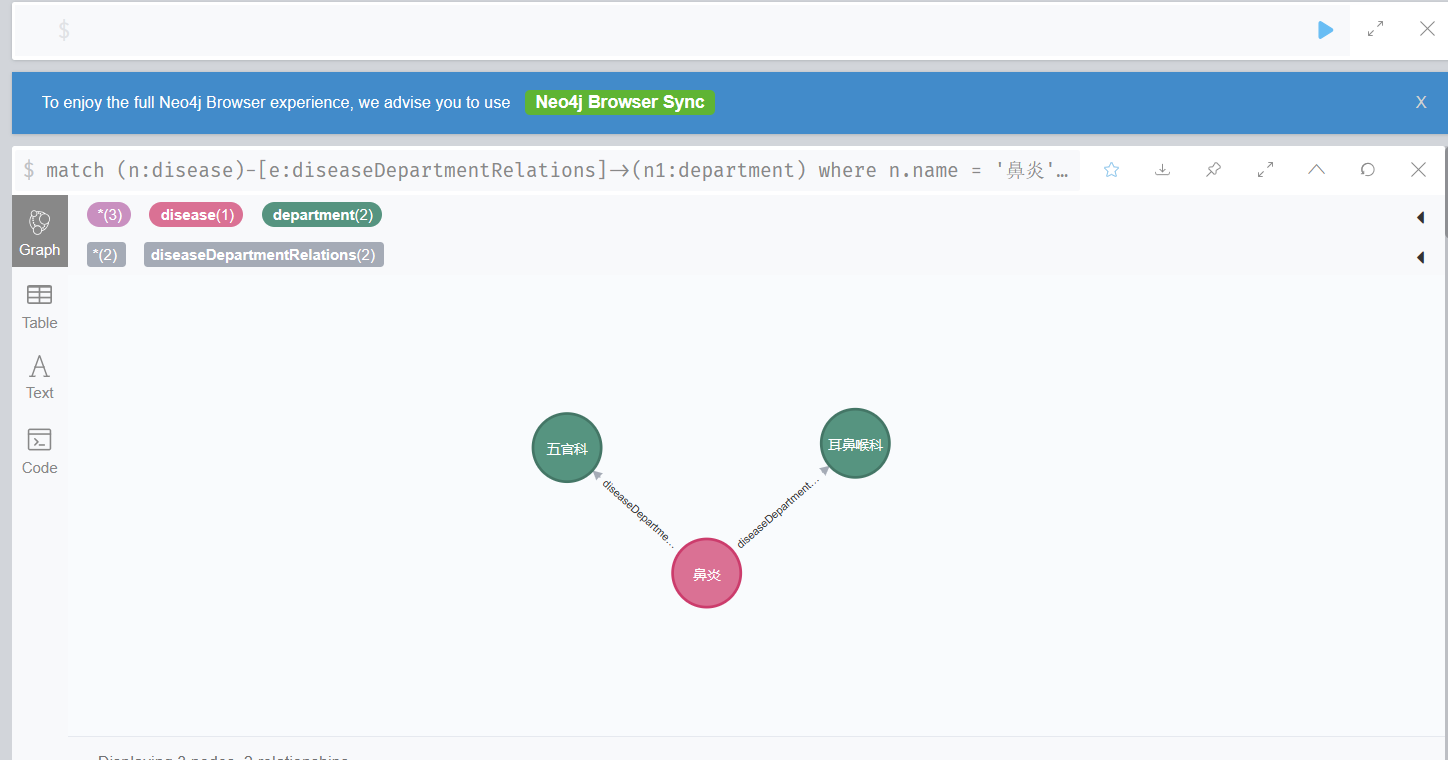
鼻炎所属的科室:
match (n:disease)-[e:diseaseDepartmentRelations]->(n1:department) where n.name = '鼻炎' return n,n1
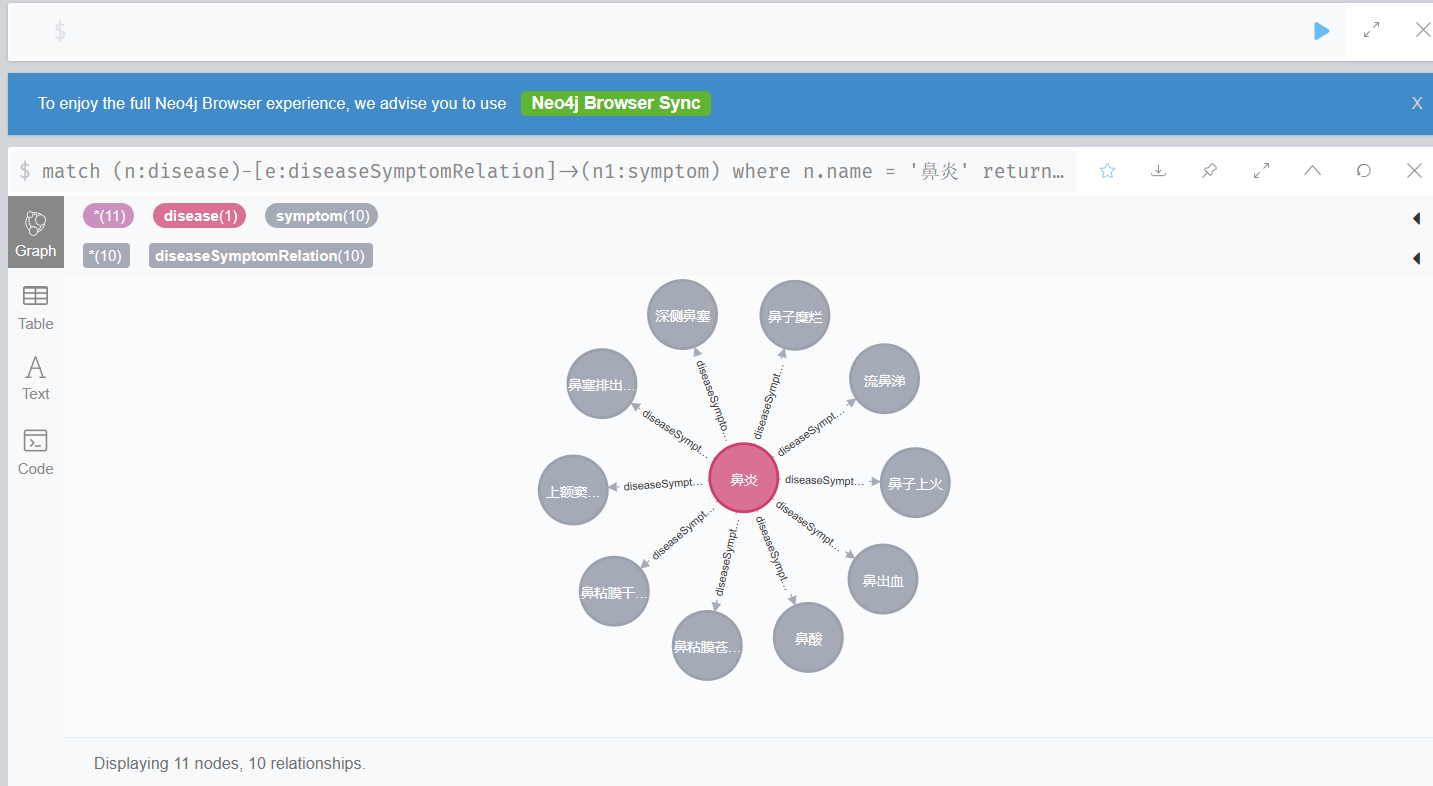
鼻炎的症状:
match (n:disease)-[e:diseaseSymptomRelation]->(n1:symptom) where n.name = '鼻炎' return n,n1
鼻炎的治疗方式:
match (n:disease)-[e:diseaseCureWayRelation]->(n1:cureWay) where n.name = '鼻炎' return n,n1

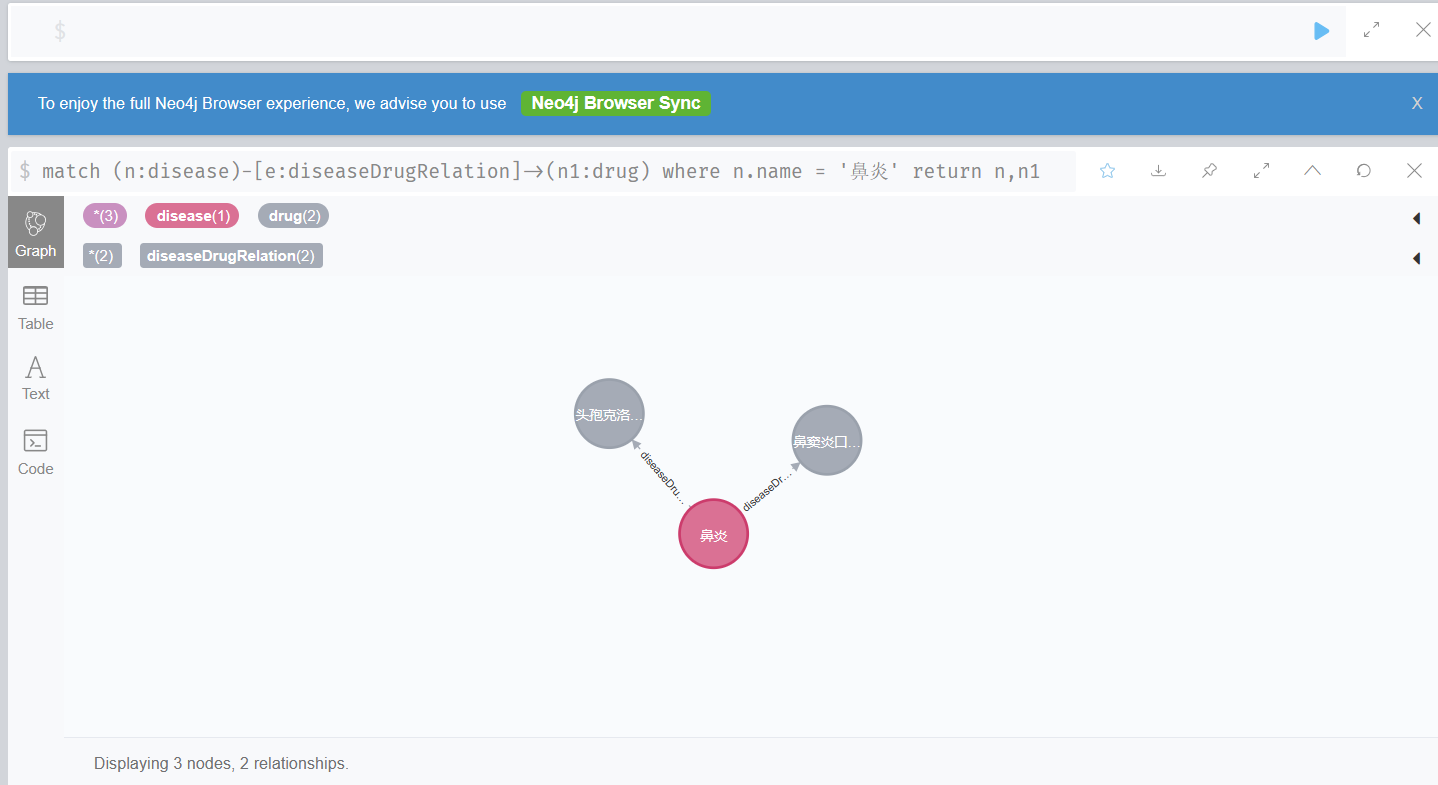
鼻炎应该用什么药:
match (n:disease)-[e:diseaseDrugRelation]->(n1:drug) where n.name = '鼻炎' return n,n1