every blog every motto: You can do more than you think.
https://blog.csdn.net/weixin_39190382?type=blog
0. 前言
密度峰值聚类算法(Density Peak Clustering Algorithm),能够自动发现数据中的密度峰值点,并根据峰值点将数据进行聚类,该算法由Alex Rodriguez和Alessandro Laio于2014年提出。发表science
https://www.science.org/doi/10.1126/science.1242072
一直感觉聚类算法上个世纪应该研究差不多了,没想到这么近(2014)还有不错的算法提出,而且发表在science,有点激动,23333
1. 正文
1.0 思想
- 聚类中心密度应当较大
- 聚类中心应当离比其密度更大的点较远
1.1 概念
- 局部密度 ρ \rho ρ :样本点一定范围内样本的数量,用来描述该点的密度。
- 相对距离 δ \delta δ : 样本点与比他密度更大点之间的距离,一般采用欧式距离。
1.2 实现说明
我们需要计算所有点的局部密度 ρ \rho ρ和相对距离 δ \delta δ,当这两个值都大时,我们将该点定义为密度峰值(类簇中心),然后将剩余点分配给密度比它高的最近数据点所在的类簇。
1.3 密度和距离的计算
1.3.1 局部密度计算
局部密度表示一个样本点一定范围内样本点的数量,反应该点的密集程度
ρ i = ∑ K ( d i s t ( i , j ) ) \rho_i = \sum K(dist(i,j)) ρi=∑K(dist(i,j))
其中K是一个核函数。常见的核函数有:
- 高斯核函数 Gaussian kernel
- 截断核函数 Truncated kernel
- Epanechnikov
- 截断核函数
ρ
i
=
∑
i
≠
j
χ
(
d
i
j
−
d
c
)
\rho_i = \sum_{i \neq j} \chi(d_{ij} - d_c)
ρi=i=j∑χ(dij−dc)
χ
(
x
)
=
{
1
,
x
<
0
0
,
i
x
≥
0
\chi(x) = \begin{cases} 1, & x \ < \ 0\\ 0, &ix \ \geq \ 0 \end{cases}
χ(x)={1,0,x < 0ix ≥ 0
对于每个点,以dc为半径,统计其中的点的数目。数据越多说明密度越大
对于大规模数据,截断函数聚类效果较好,
小规模数据,高斯核函数聚类效果较好。
1.3.2 相对距离计算
注意: 这个概念很多博客都说了,后面的博客就继续抄错了,具体可看原论文,原始表述是min,而很多博客都表述很max。

论文左侧红框:
δ i = min j : ρ j > ρ i ( d i j ) \delta_i = \min_{j:\rho_j > \rho_i} (d_{ij}) δi=j:ρj>ρimin(dij)
一句话概括: 在一个班级里面找成绩比自己好,但是挨着最近的人,计算自己和他的距离
即,找局部密度比当前点 i i i大,但是距离最近的点,计算 i i i到这个点的距离。
论文右侧红框:
For the point with highest density, we conventionally take δ i = m a x i ( d i j ) \delta_i = max_i(d_{ij}) δi=maxi(dij)
翻译:对于密度最大的点,通常认为 δ i = m a x i ( d i j ) \delta_i = max_i(d_{ij}) δi=maxi(dij)
Note that δ i \delta_i δi is much larger than the typical nearest neighbor distance only for points that are local or global maxima in the density.
翻译:注意,只有在密度达到局部最大值或全局最大值时,相对距离 δ i \delta_i δi才比周围的样本(他的邻居)点大很多。
Thus, cluster centers are recognized as points for which the value of δ i \delta_i δi is anomalously large
翻译:因此,聚类中心通常最被认为相对距离较大的点
对于密度最高的点,因为上面的表述需要找比自己密度大的点,但已经是密度最高了,所以自然无法找到比自己密度还大的点。那么对于这个点距离怎么算?
该点计算与距离最远的局部密度最大点的距离。
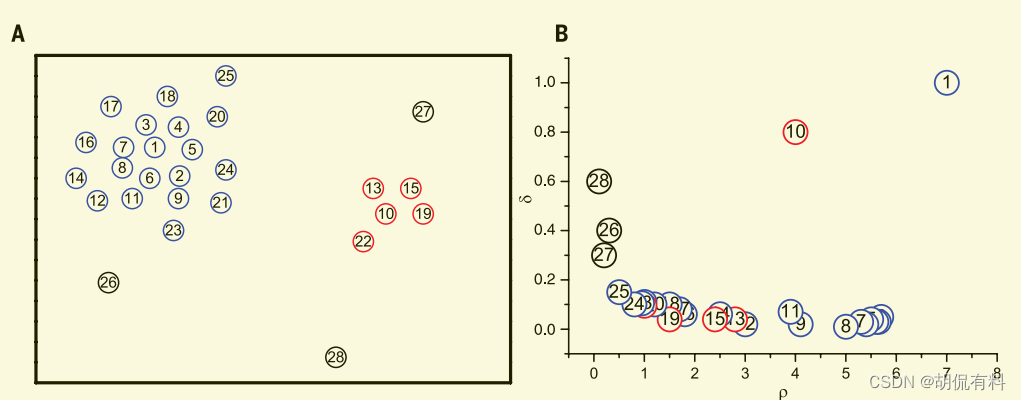
1.3.3 图例说明
右图,
- 横轴:密度
- 纵轴:距离
观察:
- 1和10号点,密度和相对距离都比较大,所以是样本中心
- 26和28号点密度较低,相对距离较大,所以是异常点
- 其他点根据和聚类中心的距离,划分到不同的簇中
对于同一簇中的点,非簇中心点,他的相对距离计算时如何的?
他周围的邻居密度可能比大或者簇中心的密度肯定比他大,那么计算他到邻居或者簇中心的距离。因为在同一簇中,这个距离这比较小的。故存在下右图靠近横轴的一排点,密度不同,但相对距离都比较小。
这对应的上面的翻译2。
1.4 算法步骤
- 计算任意两点之间距离
- 根据截断距离,计算任意点的局部密度 ρ \rho ρ
- 计算任意点的相对距离 δ \delta δ
- 以 ρ \rho ρ为横轴, δ \delta δ为纵轴,画出决策树
- 利用决策树,将 ρ \rho ρ和 δ \delta δ都大的点标记为簇中心, δ \delta δ大 ρ \rho ρ小的点标记为噪声
- 将剩余点进行分配,将每个点分配到它的最近邻且密度比其大的点所在的簇
1.5 案例
import numpy as np
import matplotlib.pyplot as plt
import csv #可以导入csv文件
# 计算数据点两两之间的距离
def getDistanceMatrix(datas):
N, D = np.shape(datas)
dists = np.zeros([N, N])
for i in range(N):
for j in range(N):
vi = datas[i, :]
vj = datas[j, :]
dists[i, j] = np.sqrt(np.dot((vi - vj), (vi - vj)))
return dists
# 找到密度计算的阈值dc
# 要求平均每个点周围距离小于dc的点的数目占总点数的1%-2%
def select_dc(dists):
'''算法1'''
N = np.shape(dists)[0]
tt = np.reshape(dists, N * N)
percent = 2.0
position = int(N * (N - 1) * percent / 100)
dc = np.sort(tt)[position + N]
return dc
# 计算每个点的局部密度
def get_density(dists, dc, method=None):
N = np.shape(dists)[0]
rho = np.zeros(N)
for i in range(N):
if method == None:
rho[i] = np.where(dists[i, :] < dc)[0].shape[0] - 1
else:
rho[i] = np.sum(np.exp(-(dists[i, :] / dc) ** 2)) - 1
return rho
# 计算每个数据点的密度距离
# 即对每个点,找到密度比它大的所有点
# 再在这些点中找到距离其最近的点的距离
def get_deltas(dists, rho):
N = np.shape(dists)[0]
deltas = np.zeros(N)
nearest_neiber = np.zeros(N)
# 将密度从大到小排序
index_rho = np.argsort(-rho)
for i, index in enumerate(index_rho):
# 对于密度最大的点
if i == 0:
continue
# 对于其他的点
# 找到密度比其大的点的序号
index_higher_rho = index_rho[:i]
# 获取这些点距离当前点的距离,并找最小值
deltas[index] = np.min(dists[index, index_higher_rho])
# 保存最近邻点的编号
index_nn = np.argmin(dists[index, index_higher_rho])
nearest_neiber[index] = index_higher_rho[index_nn].astype(int)
deltas[index_rho[0]] = np.max(deltas)
return deltas, nearest_neiber
# 通过阈值选取 rho与delta都大的点
# 作为聚类中心
def find_centers_auto(rho, deltas):
rho_threshold = (np.min(rho) + np.max(rho)) / 2
delta_threshold = (np.min(deltas) + np.max(deltas)) / 2
N = np.shape(rho)[0]
centers = []
for i in range(N):
if rho[i] >= rho_threshold and deltas[i] > delta_threshold:
centers.append(i)
return np.array(centers)
# 选取 rho与delta乘积较大的点作为
# 聚类中心
def find_centers_K(rho, deltas, K):
rho_delta = rho * deltas
centers = np.argsort(-rho_delta)
return centers[:K]
def cluster_PD(rho, centers, nearest_neiber):
K = np.shape(centers)[0]
if K == 0:
print("can not find centers")
return
N = np.shape(rho)[0]
labs = -1 * np.ones(N).astype(int)
# 首先对几个聚类中进行标号
for i, center in enumerate(centers):
labs[center] = i
# 将密度从大到小排序
index_rho = np.argsort(-rho)
for i, index in enumerate(index_rho):
# 从密度大的点进行标号
if labs[index] == -1:
# 如果没有被标记过
# 那么聚类标号与距离其最近且密度比其大
# 的点的标号相同
labs[index] = labs[int(nearest_neiber[index])]
return labs
def draw_decision(rho, deltas, name="0_decision.jpg"):
plt.cla()
for i in range(np.shape(datas)[0]):
plt.scatter(rho[i], deltas[i], s=16., color=(0, 0, 0))
plt.annotate(str(i), xy=(rho[i], deltas[i]), xytext=(rho[i], deltas[i]))
plt.xlabel("rho")
plt.ylabel("deltas")
plt.savefig(name)
plt.show()
def draw_cluster(datas, labs, centers, dic_colors, name="0_cluster.jpg"):
plt.cla()
K = np.shape(centers)[0]
for k in range(K):
sub_index = np.where(labs == k)
sub_datas = datas[sub_index]
# 画数据点
plt.scatter(sub_datas[:, 0], sub_datas[:, 1], s=16., color=dic_colors[k])
# 画聚类中心
plt.scatter(datas[centers[k], 0], datas[centers[k], 1], color="k", marker="+", s=200.)
plt.savefig(name)
plt.show()
if __name__ == "__main__":
dic_colors = {0: (.8, 0, 0), 1: (0, .8, 0),
2: (0, 0, .8), 3: (.8, .8, 0),
4: (.8, 0, .8), 5: (0, .8, .8),
6: (0, 0, 0)}
# file_name='spiral'
# with open('spiral.txt','r') as f:
# lines = f.read().splitlines()
# lines = [line.split("\t")[:-1] for line in lines]
# datas = np.array(lines).astype(np.float32)
file_name='iris'
with open('iris.csv','r') as fc:
reader=csv.reader(fc)
lines1=[]
for line in reader:
lines1.append(line)
lines=lines1[1:]
print("lines:\n", lines)
datas = np.array(lines).astype(np.float32)
print("datas:\n", datas)
# 计算距离矩阵
dists = getDistanceMatrix(datas)
# 计算dc
dc = select_dc(dists)
print("dc", dc)
# 计算局部密度
rho = get_density(dists, dc, method="Gaussion")
# 计算密度距离
deltas, nearest_neiber = get_deltas(dists, rho)
# 绘制密度/距离分布图
draw_decision(rho, deltas, name=file_name + "_decision.jpg")
# 获取聚类中心点
centers = find_centers_K(rho, deltas, 3)
# centers = find_centers_auto(rho,deltas)
print("centers", centers)
labs = cluster_PD(rho, centers, nearest_neiber)
draw_cluster(datas, labs, centers, dic_colors, name=file_name + "_cluster.jpg")
1.6 存在的问题
该部分参考:https://www.cnblogs.com/eisenji/p/16417340.html
1.6.1 多密度峰问题
如果可以在一个簇中找到两个具有较高局部密度和相对距离较大的数据点,这两个点就会被识别成两个簇心。比如下左图被聚成了下右图。优化是,改进该算法中“一个簇只有一个密度峰”的假设。比如,很多改进算法是先以各种算法进行预聚类,再通过各种方法拆分聚类结果,再通过各种方法合并拆分的结果;或者是先拆分后合并。
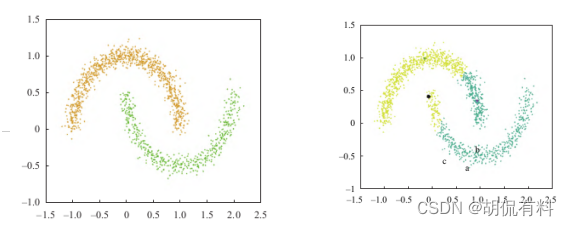
1.6.2 分配策略问题
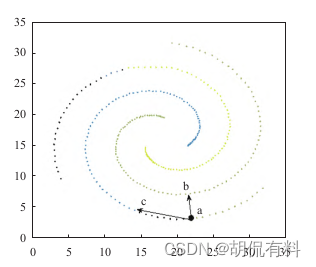
完成簇心的识别后,其余每个点分配到与其最近的高密度点的同一个簇。于是可能会出现下图的错误:a点周围的点的密度都不超过a点。距离a点最近的高密度点是b点,但是a点实际上不应该与b归为一簇,而应该与c点归为同一簇。而分配错了这个点,将导致更多与之相连的点被错误聚类。优化是,边分配边调错;将一步完成分配改为两步或多步;DPC算法只计算了通过点之间关系计算距离和密度,可加入更多的量来协助点的分配。
1.6.3 复杂度问题
时间空间都是n²复杂度,需要很高计算成本,限制了该算法在大规模数据集上的应用。优化是,比如,用网格对象代替数据对象,减少距离计算。或者,对数据进行预划分,同时除去一些小密度的数据对象;或者通过某些方法判定除去更多的不需要参与计算的数据对象,不过需要保证损失聚类精度在可控的范围内。
参考
[1] https://blog.csdn.net/lijunyan5/article/details/116161915
[2] https://blog.csdn.net/m0_74086448/article/details/131635573
[3] https://zhuanlan.zhihu.com/p/406113860
[4] https://www.cnblogs.com/kailugaji/articles/11379198.html#:~:text=%E5%9F%BA%E4%BA%8E%E5%AF%86%E5%BA%A6%E5%B3%B0%E5%80%BC%E7%9A%84%E8%81%9A%E7%B1%BB%E7%AE%97%E6%B3%95%E5%85%A8%E7%A7%B0%E4%B8%BA%E5%9F%BA%E4%BA%8E%E5%BF%AB%E9%80%9F%E6%90%9C%E7%B4%A2%E5%92%8C%E5%8F%91%E7%8E%B0%E5%AF%86%E5%BA%A6%E5%B3%B0%E5%80%BC%E7%9A%84%E8%81%9A%E7%B1%BB%E7%AE%97%E6%B3%95%20%28clustering%20by%20fast%20search%20and,find%20of%20density%20peaks%2C%20DPC%29%E3%80%82%20%E5%AE%83%E6%98%AF2014%E5%B9%B4%E5%9C%A8Science%E4%B8%8A%E6%8F%90%E5%87%BA%E7%9A%84%E8%81%9A%E7%B1%BB%E7%AE%97%E6%B3%95%EF%BC%8C%E8%AF%A5%E7%AE%97%E6%B3%95%E8%83%BD%E5%A4%9F%E8%87%AA%E5%8A%A8%E5%9C%B0%E5%8F%91%E7%8E%B0%E7%B0%87%E4%B8%AD%E5%BF%83%EF%BC%8C%E5%AE%9E%E7%8E%B0%E4%BB%BB%E6%84%8F%E5%BD%A2%E7%8A%B6%E6%95%B0%E6%8D%AE%E7%9A%84%E9%AB%98%E6%95%88%E8%81%9A%E7%B1%BB%E3%80%82
[5] https://blog.csdn.net/qq_37055672/article/details/130000567
[6] https://sci-hub.hkvisa.net/10.1126/science.1242072
[7] https://blog.csdn.net/weixin_45317919/article/details/121472851
[8] https://www.cnblogs.com/eisenji/p/16417340.html