Evolutionary Multi Objective Optimization in Searching for Various Antimicrobial Peptides
小生境共享(Niche Sharing)是生物进化算法中的一个重要概念。在传统的进化算法中,通常会假设每个个体都是独立且不同的,因此可能会导致种群中出现大量相似的个体,难以保持群体多样性。
为了解决这个问题,小生境共享被引入到进化算法中。其基本思想是将种群分割成许多不同的小生境,每个小生境只能容纳一定数量的个体,这样可以避免相似的个体竞争同一资源,从而维持种群的多样性。具体来说,小生境共享包括两个主要的步骤:
-
定义适应度函数:需要定义适应度函数,它不仅考虑个体的表现,还考虑到它所在的生境。通常情况下,适应度函数应该通过惩罚函数的方式,惩罚那些与同一生境中的其他个体过于相似的个体。
-
选择操作:在选择操作中,需要根据适应度函数对种群中的个体进行选择。不同于传统进化算法中的选择操作,小生境共享中的选择操作通常是以生境为单位进行的。也就是说,从每个小生境中选择一个最适应的个体,而不是在整个种群中选择最适应的个体。
通过这种方式,小生境共享可以促进进化算法的探索性,提高搜索的效率。同时,它还能帮助算法避免陷入局部最优解。
抗生素是能够抑制或是杀死病原微生物重要的药物。但是,随着抗生素的广泛使用,病原微生物对传统抗生素的耐药性已经成为越来越严重的问题。抗菌肽是一类极具潜力的抗菌药物。而目前的抗菌肽设计方法难以在海量的多肽序列空间种搜索性能优越的新型抗菌肽。
提出一种基于多目标进化算法的抗菌肽设计方法,可快速高效的获得抗菌活性高且多样性好的抗菌肽。
1.多目标优化问题建模

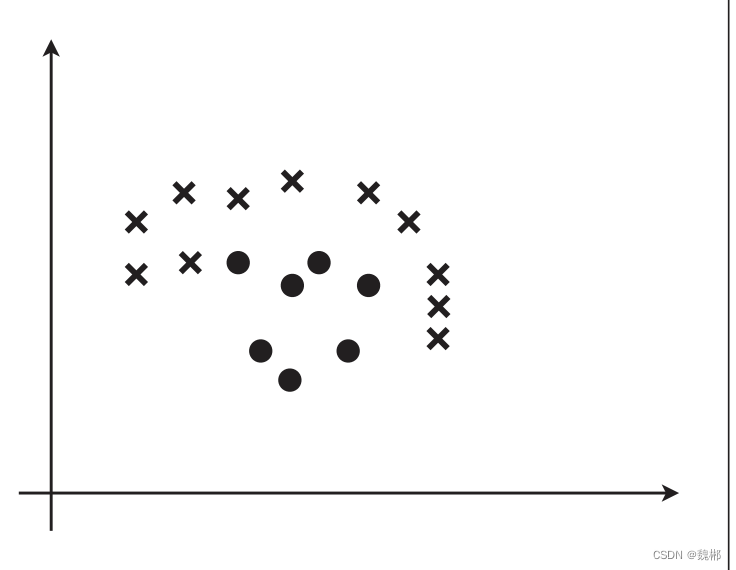
研究团队将抗菌肽设计问题转化为一个多目标优化问题,如图1所示。利用BERT模型预测出多肽的抗菌活性,作为第一个优化目标f1。
同时提出一种新的计算多肽相似性的方法,结合小生境共享计算多样性,作为第二个优化目标f2。
2.多目标优化算法

研究团队针对抗菌肽设计问题,提出一种多目标优化算法,如图2所示。通过交叉变异、环境选择产生新的候选抗菌肽,并将优质的候选抗菌肽存储在精英档案,提高学习效率。为了增强搜索能力,研究团队将局部搜索与进化算法相结合。在搜索后期,使用局部搜索算子对候选抗菌肽进一步开发。
3.实验结果
数据集:由Daniel等人提供,其中包含1778个来自于APD3的已被实验验证的抗菌肽和1778个来自于UniProt的经实验验证的非抗菌肽。
Baselines:LSTM RNN、AMP-LM、AMPGAN v2、LSTMAMP、标准的进化算法(sEA)、小生境进化算法(nEA)
评估指标:Scalable Niching Method (SC)、Pure Diversity (PD)、Dissimilarity to Dataset (DD)、Self-Organizing Map (SOM)
4.总结
研究团队提出了一种新的抗菌肽设计方法(AMPEMO)。AMPEMO将抗菌肽设计公式化为一个双目标优化问题。
第一个目标是关于深度学习模型预测的抗菌活性(适应度),第二个目标是通过小生境共享估计多样性(多样性)。
并且提出了一种多目标进化算法来解决这个问题。该算法使用了一个基于分解的框架和一个精英档案(保留优质的种群为下一个孩子),结合局部搜索策略(变异强度可学习)以提高搜索效率。最后,研究团队将AMPEMO与标准的进化算法、小生境进化算法和四种目前最先进的基于深度学习的方法进行比较。实验结果表明,AMPEMO在设计优质抗菌肽方面优于现有比较方法。
笔者有话说:
进化算法现在已经比较完善,但是在“如何交叉”、“变异”上面还有一些问题,怎么才能交叉变异的有效是一个值得探索的问题。
IEEE CIM | DrugAI团队提出基于多目标进化算法的抗菌肽设计方法