我们直接从PACS系统里拷贝下来的图像,很多情况下是乱序的,随机命名的。如下图
从这个文件夹名字,我们只知道患者的 ID 信息,不知道这个图像是什么模态(CT/MRI/Xray),也不知道扫的是哪个部位,平扫还是增强,层厚是多少等等信息。这些信息都只有用专业DICOM查看软件才能知道
因此,推荐在做一切预处理前,先把这些文件 sort 一下,根据自己需要的信息重命名文件夹。
这里我们只需要借助一个开源代码,dicomsort.py,就可以迅速 sort 啦,而且速度非常快。
下载 dicomsort
文件下载地址
https://github.com/pieper/dicomsort
从上述链接中,下载dicomsort.py这个文件,只下载这个文件即可
确保依赖项已安装
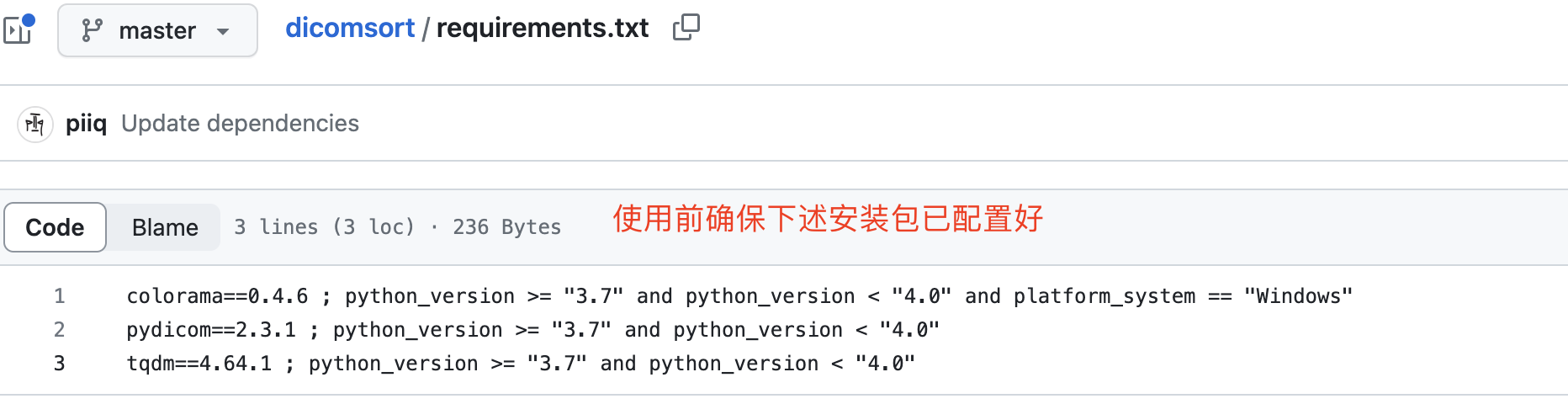
如图,使用这个dicomsort.py文件前,确保上述依赖项已安装,比如最重要的包pydicom。
如果没有安装,则使用pip install pydicom
sort 文件

如图,假设在test case文件夹下,有若干个CT图像需要sort,那么把这些文件都统一存在一个文件夹中,我们这里是对CT1202302110080里面的文件进行sort. 假设有若干个CT1202302110080,那么再创建一个文件夹,如’images_raw’,然后把所有CT号命名的患者图像都存进去。
我们再新建一个 ‘sorted‘文件夹,用于存放 sort 好后的文件。并且,把下载好的dicomsort.py放在这里。
然后在test case下打开终端,输入下列命令进行sort
python dicomsort.py input_folder/ outputfolder/%PatientID_%PatientName/%Modality/%StudyDate_%StudyTime_%StudyDescription_%StudyID/%SeriesNumber_%SeriesDescription/%InstanceNumber.dcm
这里的outputfolder后面可以自定义文件夹的长度和名字。如一级子文件夹是由ID(此案例中是CT号)和患者名字命名,二级子文件夹是由图像模态命名(CT/MR等),依次类推。
注意:这里outputfolder的命名很长,每次都要敲这行命令也记不住,可以把这行命令放在dicomsort_script.txt文件里,每次只需要粘贴复制就方便很多
演示:

结果展示:
原始文件夹

sort后的效果
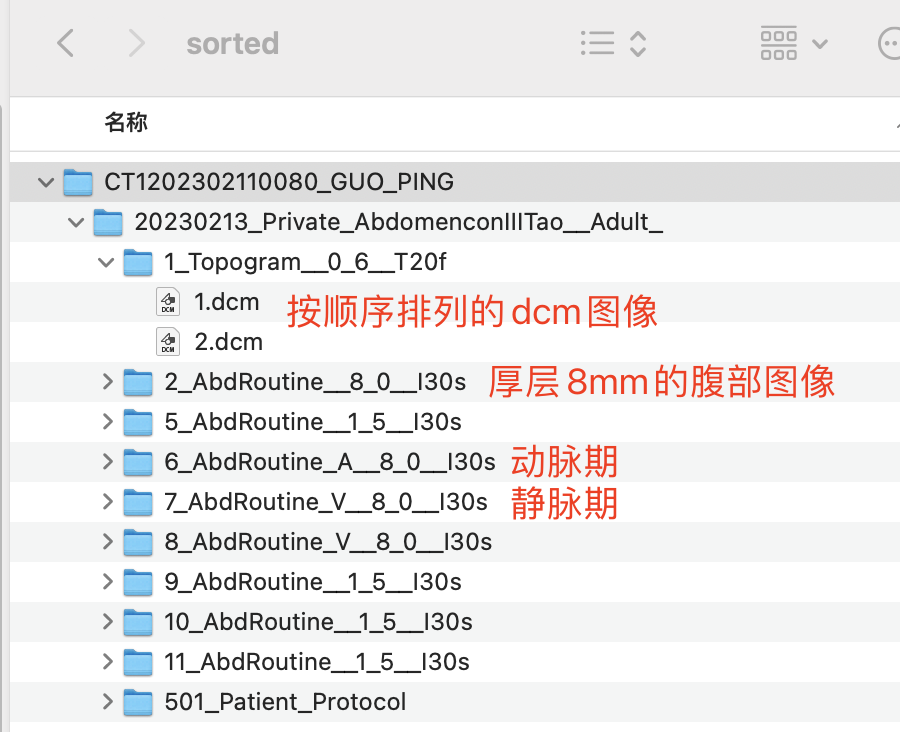
是不是一目了然啦。
当然,你对命名有自己的想法,或者输出有更高的要求,python dicomsort.py --help获取更多信息
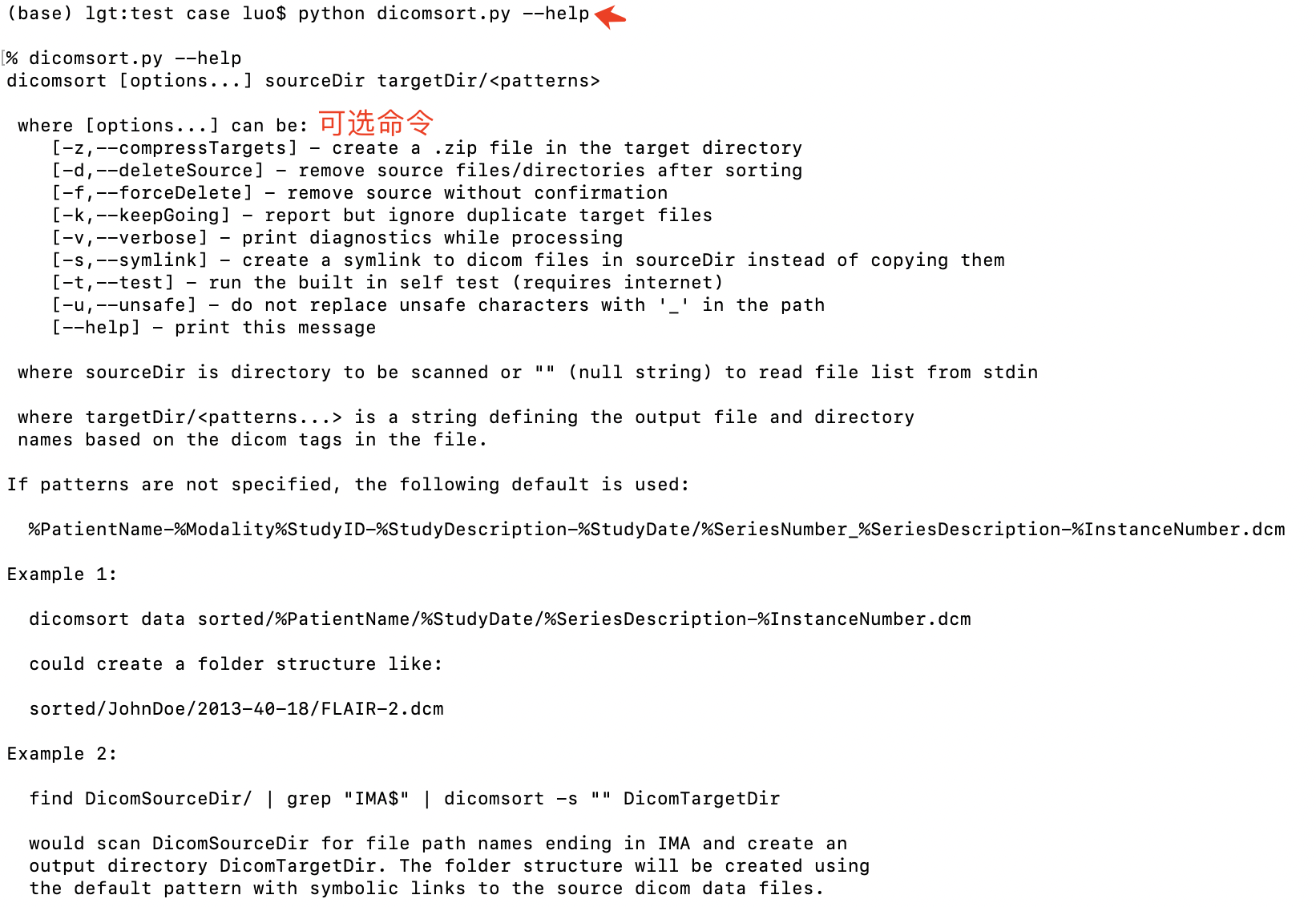
有时间的朋友可以研究一下