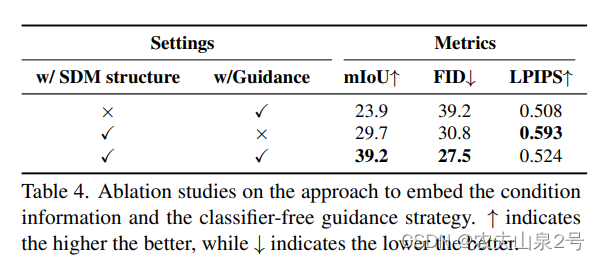
BrainStat工具箱在茗创科技往期推文【点此阅读→资源分享 | 利用机器学习进行高级MRI分析】中作过简单介绍。近日,NeuroImage杂志发布了题为BrainStat: a toolbox for brain-wide statistics and multimodal feature associations的预印版文章。这篇文章详细阐述了BrainStat工具箱包含的模块,并提供了在Python和MATLAB中执行该工具箱的教程和示例代码。
导读
神经成像数据集的分析和解释已成为一项多学科的工作,不仅依赖于统计方法,而且越来越多地依赖于与其他脑源性特征(如基因表达、组织数据、功能和认知结构)的关联。本研究介绍了BrainStat工具箱——用于体积和基于表面的脑成像数据集的单变量和多变量线性模型工具箱,以及与基因表达和组织空间图有关结果的多域特征关联,基于任务态fMRI的元分析,以及在几种常见表面模板上的静息态fMRI序列。将统计数据和特征的关联组合到一个工具箱中,不仅简化了分析过程,而且促进了跨模态研究。该工具箱在Python和MATLAB中实现,这是神经成像和神经信息学领域广泛使用的两种编程语言。BrainStat是公开可用的,并辅以可扩展的文档。
前言
神经成像能够对个体以及群体的形态、微观结构、功能和连接进行全脑测量。通过不断增强的图像处理技术,这些数据可以纳入到一个标准化的参考框架中,包括立体定位体素空间(如常用的MNI152空间)、基于表面的空间(如fsaverage)、MNI152-CIVET表面或灰度坐标,以及分割方案。将神经成像数据配准到一个公共空间,可以应用统计分析,包括在每个测量单元上执行并行统计检验的大量单变量广义线性和混合效应模型。通常,此类分析需要使用多种工具和程序来执行,从而降低了工作流程的可重复性,并增加了人为错误的风险。基于此,本研究提出了BrainStat,这是一个统一的工具箱,可以在一个内聚的,透明的和开源的框架中实现这些分析。
神经成像研究的高级分析工作流程越来越依赖于先前获得的跨多种(非)成像模式的数据集的可用性。当映射到与神经成像测量相同的参考系时,这些数据集可用于研究结果的情境化,并有助于结果的解释和验证。研究结果可以在大脑功能结构的既定序列中进行情境化,例如基于静息态fMRI或功能梯度的内在功能社区,两者都允许根据既定的大脑功能结构来解释研究结果。情境化的另一种常用法是使用Neurosynth、NiMARE或BrainMap进行自动元分析。这些工具提供了对先前发表的数千项fMRI研究进行即席元分析的能力。将统计地图与认知术语相关的大脑激活图数据库相关联,即所谓的元分析解码,它提供了一种定量方法来推断与空间统计模式相关的合理认知过程。最后,转录组学和组织学的事后数据集映射到一个公共神经成像空间,使神经成像结果与基因表达和微观结构模式相关联。这些发现可以提供有关大脑中分子和细胞特性的信息,这些信息在空间上与观察到的统计图相协变化。通过结合这些特征关联技术,可以推断神经成像结果的功能、组织学和遗传相关性。BrainStat提供了一个集成的解码工具来执行这些多模态特征关联。
本研究工具箱可以在Python和MATLAB中并行实现,这是神经成像研究社区中的两种常见编程语言。因此,BrainStat的一个关键设计选择是最大限度地实现同质化,这增强了工具的可访问性,并帮助用户在没有先验编程专业知识的情况下学习一种或两种编程语言。BrainStat依赖于一个简单的面向对象框架来简化分析工作流程。该工具箱可在https://github.com/MICA-MNI/BrainStat上公开获取,相应文档说明可在https://brainstat.readthedocs.io/上获得。本研究将工具箱分为两个主要模块:统计和情境化(图1)。接下来将介绍如何执行图1所示的分析。
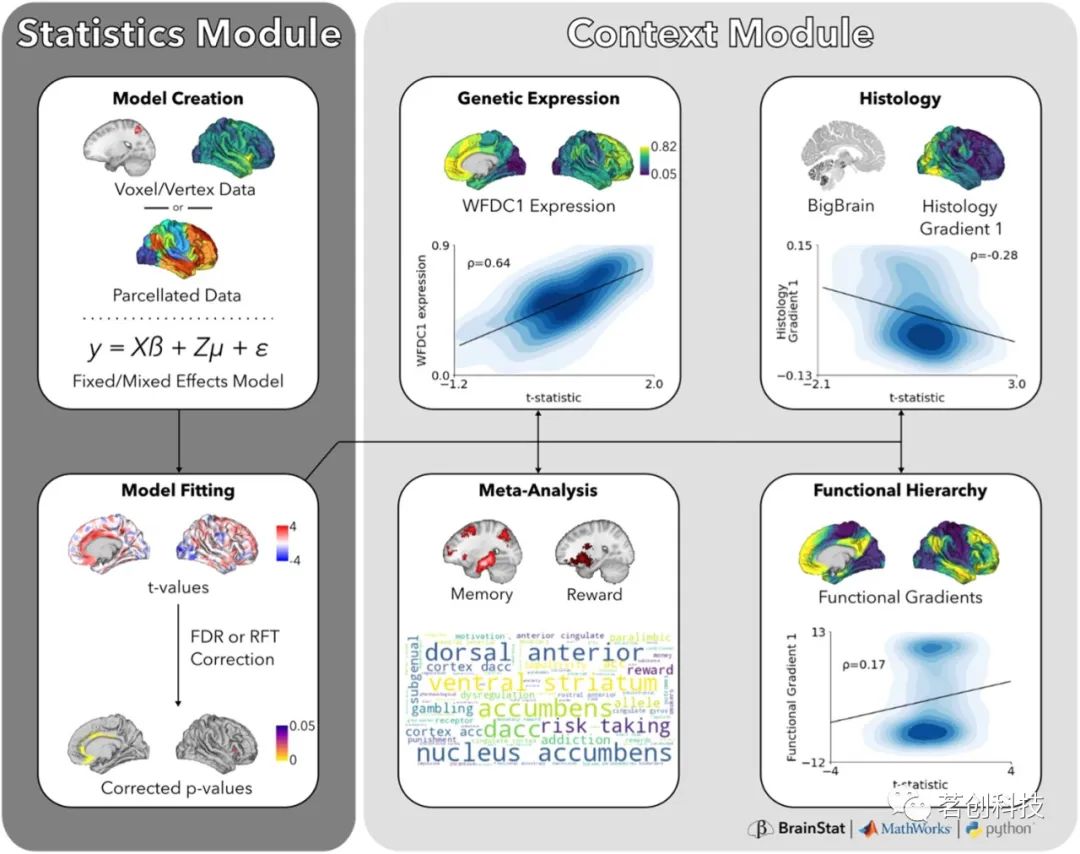
图1.BrainStat工作流程。工作流分为用于线性固定和混合效应模型的统计模块(深灰色)和用于将结果与外部数据集结合起来的情境化模块(浅灰色)。要使用统计模块,用户必须提供体素、顶点或分割数据,必须指定固定效应或混合效应模型,并且必须指定模型对比度。指定这些参数后,BrainStat将计算模型的t值和校正后的p值。然后可以在情境化模块中使用t值图或任何其他脑图,以将统计结果嵌入任务态fMRI元分析的标记,并建立功能层次结构、遗传表达和组织学标记。
统计模块
统计模块建立在SurfStat之上,SurfStat是一个经典的MATLAB包,可用于执行固定效应和混合效应线性模型。为了在BrainStat中创建和拟合此类模型,用户提供了subject-by-region-by-variate响应矩阵以及使用直观的模型公式框架创建的预测模型。这种方法允许将固定/随机效应直接定义为主要感兴趣的变量或控制协变量,以便于进行横断面和纵向分析。对于混合效应建模,BrainStat使用G端规范,这样它就可以将多个随机效应作为独立的效应,目前拟合是通过限制性最大似然估计完成的。
为了比较感兴趣的变量(如健康/疾病、年龄)的影响,必须指定一个对比。BrainStat可以处理单变量或多变量响应数据,并提供了两种广泛使用的多重比较校正分析选项,即错误发现率和随机场理论。错误发现率控制数据中逐点(即顶点、体素、分割)假阳性的比例,而随机场理论校正报告假阳性结果的概率(无论是在峰值水平还是聚类水平)。
为了说明统计模块,研究者下载了微结构连接组学(MICA-MICs)数据集的70名参与者的皮层厚度和人口统计数据,其中12名参与者被扫描了两次(图2A)。研究者以此创建了一个线性模型,以年龄和性别及其交互作用为固定效应,以参与者作为随机效应(图2B)。这些都是使用FixedEffect和MixedEffect(因其可能包含随机效应和固定效应而得名)设置的。接下来,将对比定义为年龄,即t值为正值表示皮层厚度随着年龄的增长而减小。该模型采用单侧检验对皮层厚度数据进行拟合。图2C绘制了从随机场论得出的t值、聚类和峰值p值,以及进行错误发现率校正后的顶点p值。研究者发现,在MICA-MICs数据集中,基于随机场理论的团簇水平上存在年龄对皮层厚度的影响,但在这些簇内没有明显的顶点峰值,而在顶点水平上具有边缘显著性。这表明年龄对皮层厚度的影响覆盖广泛的区域。尽管该示例使用了自由的聚类定义阈值(p<0.01),但通常建议使用更严格的阈值(p<0.001),特别是在使用空间平滑程度较低的数据时。

图2.Python代码示例:使用BrainStat拟合年龄对皮层厚度影响的固定效应一般线性模型。
情境化模块
情境化模块可以计算具有多模态神经特征统计图的二元相关性。从v0.3.6开始,情境化模块可以链接到:①任务态fMRI元分析,②源自静息态fMRI的功能序列,③遗传表达,以及④组织学/细胞结构(图1)。元分析子模块测试与特定术语相关的脑图谱和任务态fMRI元分析之间的关联。静息态模块将神经成像结果与功能梯度相关联,这是一种表示功能连接组的低维方法。转录组学子模块从艾伦人脑图谱中提取基因表达。最后,组织学子模块从BigBrain(人脑细胞结构的3D重建)获取人脑3D图像。这些子模块都支持公共表面模板,并在可行的情况下支持自定义分割。总的来说,它们为微观和宏观大脑组织方面的统计结果分析铺平了道路。
元分析解码
BrainStat的元分析解码子模块使用来自Neurosynth和NiMARE的数据,根据其认知关联来解码统计映射(通过对先前基于任务态fMRI结果的元分析得出)。简而言之,为许多(认知)术语创建了元分析激活图,这些图可以与给定的统计映射相关联,以确定与统计映射关系最强的术语。该方法允许识别与以前发表的基于任务态的功能神经成像研究中广泛使用的认知术语的间接关联,而不依赖于在同一队列中获得的认知任务。事实上,元分析解码已被用于评估神经成像发现的认知关联。
对于元分析Neurosynth数据库中的每个术语,本研究计算了哪些研究使用该术语的频率至少为千分之一(NiMARE中的默认参数)。接下来,使用NiMARE中实现的多级核密度卡方分析计算这些标签的元分析映射。对于任何用户提供的基于表面的统计映射,将映射从表面插值到体积空间。最后,对于数据库中的每个元分析映射,计算元分析映射和统计映射之间的体素积矩相关。图3显示了用元分析术语检索前面计算的t统计映射相关性的示例。

图3.元分析解码。
静息态序列
静息时大脑的功能结构被描述为一组连续的维度,称为梯度。这些梯度突出了区域之间的渐变,并可用于通过评估与其他标记的点关系来将发现嵌入到人脑的功能架构中。先前的研究使用功能梯度来评估大脑功能结构与高级认知、海马亚区连接、β淀粉样蛋白表达和衰老、微结构组织、系统发育变化和疾病状态改变的关系。
BrainStat中包含的功能梯度源自S1200版本fsaverage5的重采样平均功能连接矩阵(以降低计算复杂度)。随后计算连接组梯度。BrainSpace的参数如下:余弦相似性核、扩散图嵌入、alpha=0.5、稀疏度=0.9和diffusion_time=0。用于计算第一个函数梯度和t统计映射之间相关性的示例代码如图4所示。这两张图之间的斯皮尔曼相关性较低(ρ=0.17)。然而,为了检验这种相关的显著性,需要校正数据中的空间自相关。BrainSpace是BrainStat的一个依赖项,包括旋转测试[1] 、Moran谱随机化和变异函数匹配三种校正。

图4.与功能梯度的关联。
遗传表达
艾伦人脑图谱(Allen Human Brain Atlas)是一个微阵列基因表达数据的数据库,其中包含超过2万个基因,这些基因来自6名成年捐赠者的组织样本。该资源可用于推导神经成像数据与分子因子之间的关联,从而深入了解产生解剖和连接组标志物的机制。例如,这些数据可用于研究遗传因素与功能连接、解剖连接之间的关系,以及疾病中连接性的变化。BrainStat的遗传解码模块利用abagen工具箱来计算给定分区的遗传表达数据。abagen的默认参数遵循以下步骤。首先,它使用alleninf包(https://github.com/chrisgorgo/alleninf)提供的坐标获取并更新所有6个捐赠者组织样本的MNI152坐标。接下来,它对探头执行基于强度的滤波,以删除不超过背景噪声的探头。随后,对于索引相同基因的探针,它选择在捐赠者之间具有最高微分稳定性的探针。然后将组织样本与分区方案中的区域相匹配。跨基因的每个样本的表达值以及跨样本的每个基因的表达值使用sigmoid函数进行归一化。最后,计算每个捐赠者的每个区域内的样本均值,然后在捐赠者之间取平均值。有关使用非默认参数的步骤详细信息,请参阅abagen文档(https://abagen.readthedocs.io/)。在Python中,BrainStat直接调用abagen,因此所有参数都可以修改。在MATLAB中,如果abagen不可用,本研究工具箱包含了用abagen预先计算的遗传表达矩阵,并为许多常见的分割方案提供默认参数。图5显示了一个示例,该示例获取先前定义的功能图谱的遗传表达,并将输出与t统计映射相关联。然后,从该模块得出的表达可用于进一步的分析,例如通过推导遗传表达的主成分并将其与先前得出的统计映射进行比较。
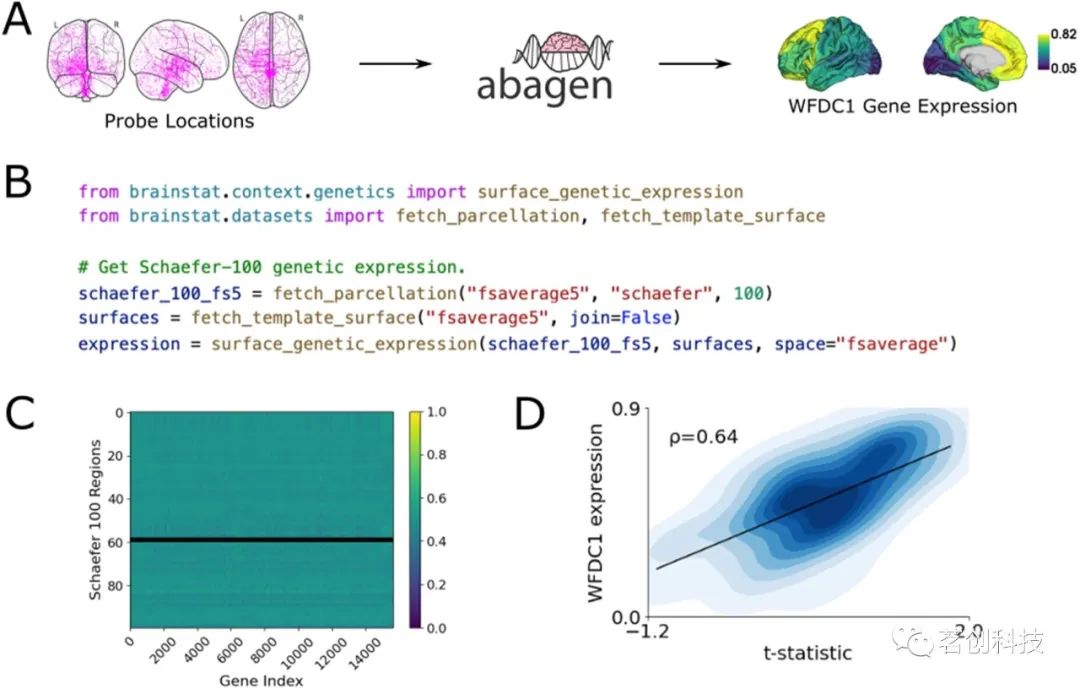
图5.与遗传表达的关联。
组织学
BigBrain图谱是对人脑切片和细胞体染色的三维重建。它的数字化各向同性分辨率为20微米,是第一个公开可用的全脑3D组织学数据集。因此,它非常适合将神经成像标志物与组织学特性相关联。例如,用于交叉验证MRI衍生的微观结构研究,根据组织学特性定义感兴趣的区域,或将连接组标记与微观结构相关联。组织学子模块旨在简化神经成像结果与BigBrain数据集的集成。该子模块使用来自BigBrain图谱的表面样本,分布在皮层地幔的50个不同深度。这些剖面的协方差,也称为微结构剖面协方差,是通过对平均强度剖面进行偏相关校正来计算的。使用默认参数的BrainSpace从微结构剖面协方差计算细胞结构变化的主轴。本研究发现第一个特征向量与ρ=-0.28的t统计映射之间存在相关性(图6)。
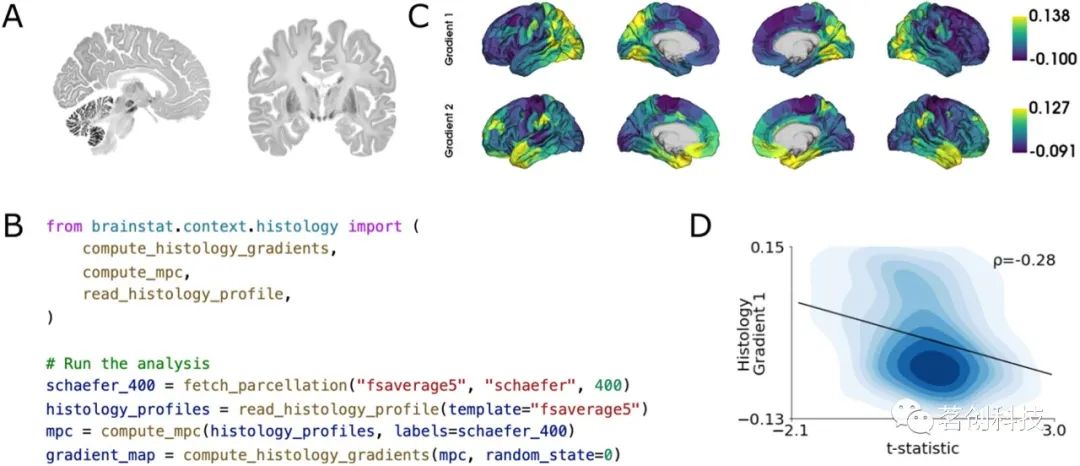
图6.与组织学标志物的关联。
讨论
脑成像数据的分析需要单变量和多变量统计推断的工具,并且越来越多地利用来自多种资源的数据以促进重要结果的解释和情境化。尽管神经影像社区已经提供了许多工具来执行单独的分析步骤,但目前还没有将统计推断和情境化方法统一起来的软件包。先前的一些工具允许对神经成像数据进行统计分析,包括Matlab中的SPM(https://www.fil.ion.ucl.ac.uk/spm/)、Matlab中的SurfStat(https://www.math.mcgill.ca/keith/surfstat/),以及Python中的nilearn(https://nilearn.github.io/)等。此外,也有一些工具允许将研究结果情境化,包括neuromaps(https://github.com/netneurolab/neuromaps)和ENIGMA工具箱(https://enigma-toolbox.readthedocs.io/)。作为唯一一款结合了统计分析和情境化的工具,并且可以在Python和Matlab中实现,BrainStat旨在提供一个完全开放访问的工具,进一步促进和巩固分析工作流程。希望这种并行实现有助于该领域分析人员和研究人员的教学和培训。值得注意的是,工具箱的模块化设置允许彼此独立地运行统计和情境化分析。此外,BrainStat还补充了一个完整且易学的在线文档,为新手和专家用户提供了一个集成分析脑成像数据的入口。
线性模型是基于神经成像推断的核心技术。许多常见的单变量和多变量统计分析,包括t检验、F检验、多元线性回归和(M)AN(C)OVA,可以被认为是一般线性模型的特殊情况。因此,一般线性模型的使用在整个神经成像研究中很普遍,同时也很重要。BrainStat的统计模块旨在为神经成像数据提供灵活的多元线性建模框架。本文的重点是展示BrainStat提供的可能性,并概述了工具箱关键功能的可访问教程。虽然现在有许多软件包可用于实现不同的分析,但这些通常是独立发布的,将这些集成起来需要专业知识,并且通常需要精通特定的编程语言。BrainStat将这些工具集成到一个统一的多语言框架中,从而提高了它们的可访问性并简化了神经成像研究的分析过程。希望通过降低这些技术的进入门槛,减少人为错误的机会,从而加速神经成像社区的跨模态研究。
目前,BrainStat实现了针对微观尺度(即转录组学和组织学)和功能特征(例如,fMRI元分析和静息态网络)的情境化工作流。这允许用户能够在基础和临床神经成像研究中采用越来越流行的分析方法。值得注意的是,一般情况下,情境关联分析的应用并不一定意味着对大脑组织的微观和宏观属性之间以及结构和功能之间关联的方向性有任何假设。事实上,人类大脑中的微观-宏观关联以及结构-功能关系仍然是一个高度活跃的研究主题,通常表明不同领域之间复杂的双向关系。
理论和实证研究都表明了可复制性在科学中的重要性。开放获取数据集和软件的需求增加可以通过允许其他人使用相同的数据和程序重做实验来提高可重复性,并减少分析中的人为错误。BrainStat对这一过程是有帮助的。通过将统计处理和多域特征关联统一到跨两种编程语言的单个包中,生成的代码将更加自动化。此外,BrainStat可能会增加缺乏建立此类集成管道专业知识的研究人员对所有这些方法的可访问性。鼓励研究人员和用户为不断增强BrainStat工具箱的功能和范围做出贡献。鼓励用户将需要解决的问题发布到GitHub问题页面(https://github.com/MICA-MNI/BrainStat/issues)。同样,通过GitHub pull requests(https://github.com/MICA-MNI/BrainStat/pulls)支持集成来自世界各地用户的新分析方法,并且可以成为未来版本的一部分。
数据和代码可用性
BrainStat可通过PyPi(https://pypi.org/project/brainstat/)、FileExchange(https://www.mathworks.com/matlabcentral/fileexchange/89827-brainstat)和GitHub(https://github.com/MICA-MNI/BrainStat)免费获得。文档可在 https://brainstat.readthedocs.io/上获得。BrainStat在Windows、macOS和Linux上支持Python 3.7-3.9和MATLAB R2019b+。但是,建议用户参考文档中的安装指南以了解最新要求。本文中使用的所有数据都可以通过BrainStat数据加载器访问。
本文中提供的示例可在Github存储库(Matlab:https://github.com/MICA-MNI/BrainStat/blob/master/extra/nimg_figures_matlab_code.mlx;Python:https://github.com/MICA-MNI/BrainStat/blob/master/extra/nimg_figures_python_code.ipynb)中获取。此外,关于线性模型的教程(Matlab:https://brainstat.readthedocs.io/en/master/matlab/tutorials/tutorial_1.html;Python:https://brainstat.readthedocs.io/en/master/python/generated_tutorials/plot_tutorial_01_basics.html)和情境解码(Matlab:https://brainstat.readthedocs.io/en/master/matlab/tutorials/tutorial_2.html;Python:https://brainstat.readthedocs.io/en/master/python/generated_tutorials/plot_tutorial_02_context.html)也可以在GitHub存储库中找到。
原文:Sara Lariviere, S¸ eyma Bayrak, Reinder Vos de Wael, Oualid Benkarim, Peer Herholz, Raul Rodriguez-Cruces, Casey Paquola, Seok-Jun Hong, Bratislav Misic, Alan C. Evans, Sofie L. Valk, Boris C. Bernhardt, BrainStat: a toolbox for brain-wide statistics and multimodal feature associations, NeuroImage (2022), doi: https://doi.org/10.1016/j.neuroimage.2022.119807
小伙伴们点星标关注茗创科技,将第一时间收到精彩内容推送哦~