1.任务描述
本关任务:利用遗传算法求解 TSP 问题。
2.相关知识
为了完成本关任务,你需要掌握:1. 遗传算法;2. TSP问题。
遗传算法
一个后继状态由两个父状态决定,以k个随机产生的状态开始(population),一个状态表示成一个字符串。
定义一个健康度量函数用来评价状态的好坏程度,通过选择,交叉,突变的操作产生下一轮状态。
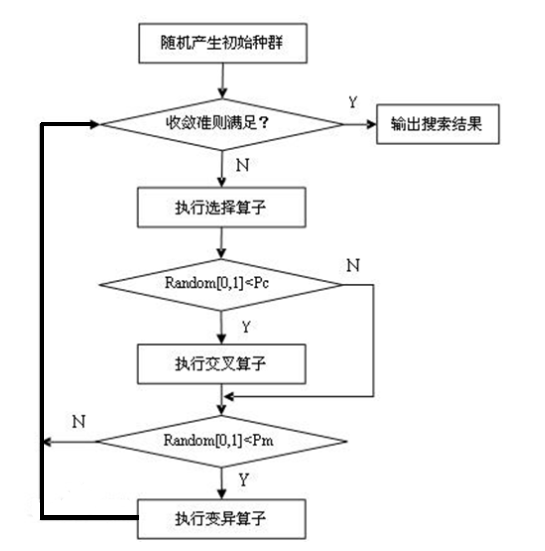
TSP问题
旅行商问题,即 TSP 问题(Traveling Salesman Problem)又译为旅行推销员问题、货郎担问题,是数学领域中著名问题之一。
假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路径的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。
3.编程要求
在右侧编辑器中补充void select()和void cross()函数,解决 TSP 问题,求所有路径中的最小路径路程。
4.测试说明
平台会对你编写的代码进行测试:
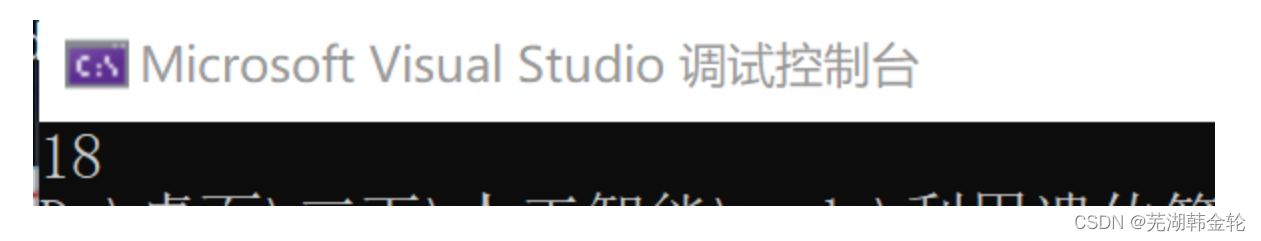
预期输出:18
5.实验过程
下面是关于非补充部分的代码解释:
1.宏定义一些常量,用来简化代码,提高效率
#define cityNum 10 //城市数量
#define popSize 10 //种群大小
#define croRate 0.85 //交叉概率
#define mutRate 0.1 //变异概率
#define MAX 999 //最大距离
2.定义“染色体”结构体,一个染色体用于标识一个个体
struct Chrom
{
int cityArr[cityNum]; //城市编号
char name; //染色体名称
float adapt; //个体适应度
int dis; //个体距离
};
3.定义种群和新种群以及临时变量
struct Chrom genes[popSize]; //种群
struct Chrom genesNew[popSize]; //新种群
struct Chrom temp;
4.定义十个城市名以及其之间的距离,用邻接矩阵表示
char names[cityNum] = { 'A','B','C','D','E','F','G','H','I','J' }; //城市名
int distance[cityNum][cityNum] = { { 0, 1, 2, 3, 4, 5, 6, 7, 8, 9 },
{ 1, 0, 1, 2, 3, 4, 5, 6, 7, 8 },
{ 2, 1, 0, 1, 2, 3, 4, 5, 6, 7 },
{ 3, 2, 1, 0, 1, 2, 3, 4, 5, 6 },
{ 4, 3, 2, 1, 0, 1, 2, 3, 4, 5 },
{ 5, 4, 3, 2, 1, 0, 1, 2, 3, 4 },
{ 6, 5, 4, 3, 2, 1, 0, 1, 2, 3 },
{ 7, 6, 5, 4, 3, 2, 1, 0, 1, 2 },
{ 8, 7, 6, 5, 4, 3, 2, 1, 0, 1 },
{ 9, 8, 7, 6, 5, 4, 3, 2, 1, 0 }
}; //距离邻接矩阵
5.void initGroup()函数:用于初始化群体,生成一组随机的染色体
void initGroup()
{
int i, j, k;
int t = 0;
int flag = 0;
srand(time(NULL)); //用于初始化随机数种子,time(NULL)返回当前系统时间
for (i = 0; i < popSize; i++) //每次生成一个染色体
{
temp.name = names[i]; //定义temp结构体,记录染色体的名称、适应度和距离
temp.adapt = 0.0f;
temp.dis = 0;
for (j = 0; j < cityNum;)
{
t = rand() % cityNum; //每次在城市列表中随机选取一个城市编号
flag = 1;
for (k = 0; k < j; k++)
{
if (genes[i].cityArr[k] == t) //判断选取的城市是否已经被选中
{
flag = 0;
break;
}
}
if (flag) //选取的城市没有被选中
{
temp.cityArr[j] = t; //存入数组中
genes[i] = temp; //生成一个完整的染色体
j++; //j自增1
}
}
}
}
6.void popFitness()函数:用于计算种群中每个个体的适应度值
void popFitness() //计算种群中每个个体的适应度值
{
int i, n1, n2;
for (i = 0; i < popSize; i++) //循环遍历种群中的所有个体
{
genes[i].dis = 0; //初始化当前个体的路径长度为0
for (int j = 1; j < cityNum; j++) //循环遍历当前个体中所有的城市
{
n1 = genes[i].cityArr[j - 1]; //获取前一个城市的编号
n2 = genes[i].cityArr[j]; //获取当前城市的编号
genes[i].dis += distance[n1][n2]; //将前一个城市到当前城市的距离累加到路径长度中
}
//将最后一个城市到第一个城市的距离加到路径长度中,得到该个体的总路径长度
genes[i].dis += distance[genes[i].cityArr[0]][genes[i].cityArr[cityNum - 1]];
//计算当前个体的适应度值,并保存到genes数组中
genes[i].adapt = (float)1 / genes[i].dis;
}
}
7.int chooseBest()函数:从种群中选择适应度最高的个体
int chooseBest()
{
int choose = 0; //适应度最高的个体的索引
float best = 0.0f; //当前适应度最高的值
best = genes[0].adapt; //初始化当前适应度最高的值
for (int i = 0; i < popSize; i++)
{
if (genes[i].adapt < best) //如果当前个体的适应度值比当前适应度最高的值还小
{
best = genes[i].adapt; //更新当前适应度最高的值
choose = i; //更新适应度最高的个体的索引
}
}
return choose; //返回适应度最高的个体的索引
}
下面需要对代码进行补充
1.补充select()函数
选择操作是遗传算法中的一个重要步骤,它的作用是从当前种群中选出一些适应度较高的个体,作为下一代个体的双亲,从而保留优良的基因
常用的实现方法有: 适应度比例法,随机遍历抽样法,局部选择法,锦标赛选择法和 序选择法(轮盘赌方法)
这里我们使用轮盘赌方法:
- 计算每个个体的适应度值和总适应度值
- 计算每个个体被选中的概率
- 生成一个0到1之间的随机数
- 从第一个个体开始累加选择概率,直到大于或等于随机数为止
- 返回当前累加到的个体作为选中结果
下面是具体代码实现:
void select()
{
float biggestSum = 0.0f; //定义适应度总和
float adapt_pro[popSize];
float pick = 0.0f;
int i;
for (i = 0; i < popSize; i++) //计算适应度总和
{
biggestSum += genes[i].adapt;
}
for (i = 0; i < popSize; i++) //计算每个个体的适应度占总适应度的比例
{
adapt_pro[i] = genes[i].adapt / biggestSum;
}
for (i = 0; i < popSize; i++)
{
pick = (float)rand() / RAND_MAX; //随机选择一个个体
/********** Begin **********/
float sum = 0.0f;
for (int j = 0; j < popSize; j++)
{
sum += adapt_pro[j]; //累加适应度比例
if (sum > pick) //当累加值超过pick时,选择对应的个体
{
genesNew[i] = genes[j];
break;
}
}
/********** End **********/
}
for (i = 0; i < popSize; i++) //将新生成的种群genesNew复制到原来的genes数组中,以更新种群
{
genes[i] = genesNew[i];
}
}
首先求出适应度总和,然后计算每个个体的适应度占总适应度的比例。遍历所有种群,每次遍历中随机生成一个0到1之间的随机数,用于模拟轮盘赌法中的轮盘转动,决定选择哪个个体。当某个个体的累加适应度比例超过这个随机数即可选择这个个体,因为它被选择的概率更高,适应度更高,基因更优良。接着将新种群复制回原种群数组中。
2.补充cross()函数
交叉操作是遗传算法中产生新个体的主要操作过程,它模拟了自然选择和遗传中发生的复制、交叉和变异等现象,以某一概率相互交换某两个个体之间的部分染色体
corss()函数的主要思想是,从第一个第二个个体开始两两进行交叉,首先随机生成一个0-1之间的浮点数,如果大于交叉概率(已经宏定义),则不进行交叉操作。
其中move变量是用来控制交叉操作的次数和对象的,它表示当前要进行交叉操作的两个个体在种群中的位置。例如,如果move=0,那么就表示第一个个体和第二个个体要进行交叉操作。move变量每次增加2,直到达到种群大小popSize-1为止这样可以保证每两个相邻的个体都有一次机会进行交叉操作,并且不会重复或遗漏。
随机生成两个交叉点。这里使用这篇文章的例子
两个父代分别为:
- 12|3456|789
- 46|2738|159
其交叉点位置pos1和pos2分别为2和6。接着交换两个交叉点之间的基因片段,生成的两个子代分别为:
- 12|2738|789
- 46|3456|159
很明显两个子代都有基因冲突,比如第一个子代中2,7,8基因都有两个,会产生冲突,所以要补充函数用来解决冲突。一个办法是交换两个冲突子代中的冲突基因。
首先记录下冲突基因的位置,保存到conflict数组中。注意只看非交叉片段和交叉片段的冲突。如12|2738|789中12和2738中的2冲突,记录下12中2的下标位置。同理789和2738中的7和8冲突,记录789中7和8的下标位置。
因此conflict1[3]={1,6,7},conflict2[3]={0,1,7}
接着将两个冲突子代的1和0,6和1,7和7的下标的对应基因进行交换,即可解决冲突,新生成的子代为:
- 14|2738|659
- 27|3456|189
代码如下:
void cross()
{
float pick;
int choice1, choice2; //交叉的两个父代
int pos1, pos2; //记录交叉点位置
int temp;
int conflict1[popSize]; //记录父代1的冲突基因位置
int conflict2[popSize]; //记录父代2的冲突基因位置
int num1; //num用来记录冲突基因个数,且两个父代冲突基因数相同
int num2;
int index1, index2; //交换基因时使用
int move = 0; //控制交叉操作的次数和对象
while (move < popSize - 1)
{
pick = (float)rand() / RAND_MAX;
// 如果随机数大于交叉概率,则不进行交叉操作,直接跳过
if (pick > croRate)
{
move += 2;
continue;
}
choice1 = move;
choice2 = move + 1;
// 随机选择两个交叉点
pos1 = rand() % popSize;
pos2 = rand() % popSize;
while (pos1 > popSize - 2 || pos1 < 1)
{
pos1 = rand() % popSize;
}
while (pos2 > popSize - 2 || pos2 < 1)
{
pos2 = rand() % popSize;
}
//保证pos1比pos2小
if (pos1 > pos2)
{
temp = pos1;
pos1 = pos2;
pos2 = temp;
}
//交换pos1到pos2之间的基因片段
for (int j = pos1; j <= pos2; j++)
{
temp = genes[choice1].cityArr[j];
genes[choice1].cityArr[j] = genes[choice2].cityArr[j];
genes[choice2].cityArr[j] = temp;
}
//处理基因冲突问题
num1 = 0;
num2 = 0;
if (pos1 > 0 && pos2 < popSize - 1)
{
/********** Begin **********/
for (int j = 0; j < pos1; j++)//记录第一个交叉点之前的片段和交叉片段之间发生冲突的基因位置
{
for (int k = pos1; k <= pos2; k++)
{
if (genes[choice1].cityArr[j] == genes[choice1].cityArr[k])
{
conflict1[num1++] = j;
}
if (genes[choice2].cityArr[j] == genes[choice2].cityArr[k])
{
conflict2[num2++] = j;
}
}
}
/********** End **********/
for (int j = pos2 + 1; j < popSize; j++)//记录第二个交叉点之后的片段和交叉片段之间发生冲突的基因位置
{
for (int k = pos1; k <= pos2; k++)
{
/********** Begin **********/
if (genes[choice1].cityArr[j] == genes[choice1].cityArr[k])
{
conflict1[num1++] = j;
}
if (genes[choice2].cityArr[j] == genes[choice2].cityArr[k])
{
conflict2[num2++] = j;
}
/********** End **********/
}
}
}
if ((num1 == num2) && num1 > 0)//将两个冲突子代的冲突基因进行交换
{
for (int j = 0; j < num1; j++)
{
index1 = conflict1[j];
index2 = conflict2[j];
temp = genes[choice1].cityArr[index1];
genes[choice1].cityArr[index1] = genes[choice2].cityArr[index2];
genes[choice2].cityArr[index2] = temp;
}
}
move += 2;
}
}
8.void mutation()函数
变异函数的作用是在一定概率下随机交换某个物种的两个城市顺序,以增加种群的多样性
void mutation()
{
double pick; //随机数
int pos1, pos2, temp; //交换位置和临时变量
for (int i = 0; i < popSize; i++) //遍历每个种群
{
pick = (float)rand() / RAND_MAX; // 生成一个0到1之间的随机数
if (pick > mutRate) // 如果随机数大于变异率,则跳过该物种
{
continue;
}
pos1 = rand() % popSize; // 随机生成第一个交换位置
pos2 = rand() % popSize; // 随机生成第二个交换位置
while (pos1 > popSize - 1) // 如果第一个位置超出范围,则重新生成
{
pos1 = rand() % popSize;
}
while (pos2 > popSize - 1) // 如果第二个位置超出范围,则重新生成
{
pos2 = rand() % popSize;
}
int a = genes[i].dis; // 记录原来的总距离
temp = genes[i].cityArr[pos1]; // 交换两个城市的顺序
genes[i].cityArr[pos1] = genes[i].cityArr[pos2];
genes[i].cityArr[pos2] = temp;
popFitness(); // 计算新的适应度函数值
if (genes[i].dis > a) // 如果新的总距离大于原来的,则恢复原来的顺序(贪心策略)
{
temp = genes[i].cityArr[pos1];
genes[i].cityArr[pos1] = genes[i].cityArr[pos2];
genes[i].cityArr[pos2] = temp;
}
}
}
6.完整代码
#include "stdio.h"
#include "stdlib.h"
#include "time.h"
#define cityNum 10 //城市数量
#define popSize 10 //种群大小
#define croRate 0.85 //交叉概率
#define mutRate 0.1 //变异概率
#define MAX 999 //最大距离
//定义染色体的结构
struct Chrom
{
int cityArr[cityNum]; //城市编号
char name; //染色体名称
float adapt; //个体适应度
int dis; //个体距离
};
struct Chrom genes[popSize]; //种群
struct Chrom genesNew[popSize]; //新种群
struct Chrom temp;
char names[cityNum] = { 'A','B','C','D','E','F','G','H','I','J' }; //城市名
int distance[cityNum][cityNum] = { { 0, 1, 2, 3, 4, 5, 6, 7, 8, 9 },
{ 1, 0, 1, 2, 3, 4, 5, 6, 7, 8 },
{ 2, 1, 0, 1, 2, 3, 4, 5, 6, 7 },
{ 3, 2, 1, 0, 1, 2, 3, 4, 5, 6 },
{ 4, 3, 2, 1, 0, 1, 2, 3, 4, 5 },
{ 5, 4, 3, 2, 1, 0, 1, 2, 3, 4 },
{ 6, 5, 4, 3, 2, 1, 0, 1, 2, 3 },
{ 7, 6, 5, 4, 3, 2, 1, 0, 1, 2 },
{ 8, 7, 6, 5, 4, 3, 2, 1, 0, 1 },
{ 9, 8, 7, 6, 5, 4, 3, 2, 1, 0 }
}; //距离邻接矩阵
//初始化群体,生成一组随机的染色体
void initGroup()
{
int i, j, k;
int t = 0;
int flag = 0;
srand(time(NULL)); //用于初始化随机数种子,time(NULL)返回当前系统时间
for (i = 0; i < popSize; i++) //每次生成一个染色体
{
temp.name = names[i]; //定义temp结构体,记录染色体的名称、适应度和距离
temp.adapt = 0.0f;
temp.dis = 0;
for (j = 0; j < cityNum;)
{
t = rand() % cityNum; //每次在城市列表中随机选取一个城市编号
flag = 1;
for (k = 0; k < j; k++)
{
if (genes[i].cityArr[k] == t) //判断选取的城市是否已经被选中
{
flag = 0;
break;
}
}
if (flag) //选取的城市没有被选中
{
temp.cityArr[j] = t; //存入数组中
genes[i] = temp; //生成一个完整的染色体
j++; //j自增1
}
}
}
}
void popFitness() //计算种群中每个个体的适应度值
{
int i, n1, n2;
for (i = 0; i < popSize; i++) //循环遍历种群中的所有个体
{
genes[i].dis = 0; //初始化当前个体的路径长度为0
for (int j = 1; j < cityNum; j++) //循环遍历当前个体中所有的城市
{
n1 = genes[i].cityArr[j - 1]; //获取前一个城市的编号
n2 = genes[i].cityArr[j]; //获取当前城市的编号
genes[i].dis += distance[n1][n2]; //将前一个城市到当前城市的距离累加到路径长度中
}
//将最后一个城市到第一个城市的距离加到路径长度中,得到该个体的总路径长度
genes[i].dis += distance[genes[i].cityArr[0]][genes[i].cityArr[cityNum - 1]];
//计算当前个体的适应度值,并保存到genes数组中
genes[i].adapt = (float)1 / genes[i].dis;
}
}
//从种群中选择适应度最高的个体
int chooseBest()
{
int choose = 0; //适应度最高的个体的索引
float best = 0.0f; //当前适应度最高的值
best = genes[0].adapt; //初始化当前适应度最高的值
for (int i = 0; i < popSize; i++)
{
if (genes[i].adapt < best) //如果当前个体的适应度值比当前适应度最高的值还小
{
best = genes[i].adapt; //更新当前适应度最高的值
choose = i; //更新适应度最高的个体的索引
}
}
return choose; //返回适应度最高的个体的索引
}
void select()
{
float biggestSum = 0.0f; //定义适应度总和
float adapt_pro[popSize];
float pick = 0.0f;
int i;
for (i = 0; i < popSize; i++) //计算适应度总和
{
biggestSum += genes[i].adapt;
}
for (i = 0; i < popSize; i++) //计算每个个体的适应度占总适应度的比例
{
adapt_pro[i] = genes[i].adapt / biggestSum;
}
for (i = 0; i < popSize; i++)
{
pick = (float)rand() / RAND_MAX; //随机选择一个个体
/********** Begin **********/
float sum = 0.0f;
for (int j = 0; j < popSize; j++)
{
sum += adapt_pro[j]; //累加适应度比例
if (sum > pick) //当累加值超过pick时,选择对应的个体
{
genesNew[i] = genes[j];
break;
}
}
/********** End **********/
}
for (i = 0; i < popSize; i++) //将新生成的种群genesNew复制到原来的genes数组中,以更新种群
{
genes[i] = genesNew[i];
}
}
void cross()
{
float pick;
int choice1, choice2; //交叉的两个父代
int pos1, pos2; //记录交叉点位置
int temp;
int conflict1[popSize]; //记录父代1的冲突基因位置
int conflict2[popSize]; //记录父代2的冲突基因位置
int num1; //num用来记录冲突基因个数,且两个父代冲突基因数相同
int num2;
int index1, index2; //交换基因时使用
int move = 0; //控制交叉操作的次数和对象
while (move < popSize - 1)
{
pick = (float)rand() / RAND_MAX;
// 如果随机数大于交叉概率,则不进行交叉操作,直接跳过
if (pick > croRate)
{
move += 2;
continue;
}
choice1 = move;
choice2 = move + 1;
// 随机选择两个交叉点
pos1 = rand() % popSize;
pos2 = rand() % popSize;
while (pos1 > popSize - 2 || pos1 < 1)
{
pos1 = rand() % popSize;
}
while (pos2 > popSize - 2 || pos2 < 1)
{
pos2 = rand() % popSize;
}
//保证pos1比pos2小
if (pos1 > pos2)
{
temp = pos1;
pos1 = pos2;
pos2 = temp;
}
//交换pos1到pos2之间的基因片段
for (int j = pos1; j <= pos2; j++)
{
temp = genes[choice1].cityArr[j];
genes[choice1].cityArr[j] = genes[choice2].cityArr[j];
genes[choice2].cityArr[j] = temp;
}
//处理基因冲突问题
num1 = 0;
num2 = 0;
if (pos1 > 0 && pos2 < popSize - 1)
{
/********** Begin **********/
for (int j = 0; j < pos1; j++)//记录第一个交叉点之前的片段和交叉片段之间发生冲突的基因位置
{
for (int k = pos1; k <= pos2; k++)
{
if (genes[choice1].cityArr[j] == genes[choice1].cityArr[k])
{
conflict1[num1++] = j;
}
if (genes[choice2].cityArr[j] == genes[choice2].cityArr[k])
{
conflict2[num2++] = j;
}
}
}
/********** End **********/
for (int j = pos2 + 1; j < popSize; j++)//记录第二个交叉点之后的片段和交叉片段之间发生冲突的基因位置
{
for (int k = pos1; k <= pos2; k++)
{
/********** Begin **********/
if (genes[choice1].cityArr[j] == genes[choice1].cityArr[k])
{
conflict1[num1++] = j;
}
if (genes[choice2].cityArr[j] == genes[choice2].cityArr[k])
{
conflict2[num2++] = j;
}
/********** End **********/
}
}
}
if ((num1 == num2) && num1 > 0)//将两个冲突子代的冲突基因进行交换
{
for (int j = 0; j < num1; j++)
{
index1 = conflict1[j];
index2 = conflict2[j];
temp = genes[choice1].cityArr[index1];
genes[choice1].cityArr[index1] = genes[choice2].cityArr[index2];
genes[choice2].cityArr[index2] = temp;
}
}
move += 2;
}
}
void mutation()
{
double pick; //随机数
int pos1, pos2, temp; //交换位置和临时变量
for (int i = 0; i < popSize; i++) //遍历每个种群
{
pick = (float)rand() / RAND_MAX; // 生成一个0到1之间的随机数
if (pick > mutRate) // 如果随机数大于变异率,则跳过该物种
{
continue;
}
pos1 = rand() % popSize; // 随机生成第一个交换位置
pos2 = rand() % popSize; // 随机生成第二个交换位置
while (pos1 > popSize - 1) // 如果第一个位置超出范围,则重新生成
{
pos1 = rand() % popSize;
}
while (pos2 > popSize - 1) // 如果第二个位置超出范围,则重新生成
{
pos2 = rand() % popSize;
}
int a = genes[i].dis; // 记录原来的总距离
temp = genes[i].cityArr[pos1]; // 交换两个城市的顺序
genes[i].cityArr[pos1] = genes[i].cityArr[pos2];
genes[i].cityArr[pos2] = temp;
popFitness(); // 计算新的适应度函数值
if (genes[i].dis > a) // 如果新的总距离大于原来的,则恢复原来的顺序(贪心策略)
{
temp = genes[i].cityArr[pos1];
genes[i].cityArr[pos1] = genes[i].cityArr[pos2];
genes[i].cityArr[pos2] = temp;
}
}
}
int main()
{
char c = 0;
//printf("\n\t\t******************************** ÒÅ´«Ëã·¨Çó½âTSP(ÂÃÐÐÉÌ)ÎÊÌâ *********************************\n");
initGroup(); //³õʼ»¯
popFitness(); //¸üÐÂÊý¾Ý
//Êä³öÅäÖÃÐÅÏ¢
/*printf("\n\t\t»ùÒò³¤¶È:%d",cityNum);
printf("\tÖÖȺ´óС:%d",popSize);
printf("\t½»²æ¸ÅÂÊ:%.2f",croRate);
printf("\t±äÒì¸ÅÂÊ:%.2f",mutRate);
printf("\t½ø»¯´úÊý:%d",MAX);
printf("\tÔ¤Éè×îÓŽ⣺18");
printf("\n\n\t\t**********************************************************************************************\n");*/
int i, j;
//Êä³ö¾àÀë¾ØÕó
/*printf("\n\t\t--------- ³ÇÊоàÀë¾ØÕó ---------\n");
printf("\t\t");
int i,j;
for(i = 0; i < cityNum; i ++)
{
for(j = 0;j < cityNum; j ++)
{
printf(" %d",distance[i][j]);
}
printf("\n\t\t");
}
printf("--------------------------------");*/
//Êä³ö·¾¶ÐÅÏ¢
/*printf("\n\t\t-------- ³õʼÖÖȺ»ùÒò¿â --------\n");
printf("\t\t ");
for(i = 0; i < cityNum; i ++)
{
printf(" %c",genes[i].name);
}
printf("\n\t\t");
for(i = 0;i < cityNum; i ++)
{
printf("%c",genes[i].name);
for(j = 0; j < cityNum; j ++)
{
printf(" %d",genes[i].cityArr[j]);
}
printf("\n\t\t");
}
printf("--------------------------------\n");*/
//printf("\n\t\tÑ°Çó×îÓŽâÖУº");
//ͨ¹ý²»¶Ï½ø»¯£¬Ö±µ½´ïµ½¶¨ÒåµÄ½ø»¯´úÊý
for (i = 0; i < MAX; i++)
{
select();
cross();
mutation();
popFitness();//¸üÐÂÊý¾Ý
int temp = (int)MAX / 20;
/*if( i % temp == 0)
{
//printf("¨‚");
Sleep(200);
}*/
}
//printf("Íê³É");
//printf("\n\n\t\t×îÓÅ·¾¶£º");
/*for(i = 0; i < cityNum ; i++)
{
printf("%d-->",genes[chooseBest()].cityArr[i]);
}
printf("%d",genes[chooseBest()].cityArr[0]);*/
printf("%d", genes[chooseBest()].dis);
//printf("\n\n\t\tÊÇ·ñÔÙÊÔÒ»´Î?(Y/y) ÊÇ/(N/n) ·ñ£º");
fflush(stdin);
/* c = getchar();
fflush(stdin);
if(c=='N'||c=='n')
{
break;
}*/
//printf("\n\t\t");
return 0;
}
运行结果: