为什么选择PCA?
- 当有许多输入属性时,很难将数据可视化。在机器学习领域有一个非常著名的术语“维度诅咒”。
- 基本上,它指的是数据集中的属性数量越多,对机器学习模型的准确性和训练时间产生不利影响。
- 主成分分析(PCA)是解决这个问题的一种方法,用于更好的数据可视化和提高准确性。
PCA是如何工作的?
- PCA是在应用任何ML算法之前执行的无监督预处理任务。PCA基于“正交线性变换”,这是一种将数据集的属性投影到新坐标系上的数学技术。描述最大方差的属性称为第一主成分,并放置在第一个坐标处。
- 类似地,在描述方差方面处于第二位的属性被称为第二主成分,依此类推。简而言之,完整的数据集可以用主成分来表示。通常,超过90%的方差可以由两个或三个主成分解释。
- 因此,主成分分析或PCA通过选择捕获关于数据集的最大信息的最重要属性来将数据从高维空间转换到低维空间。
Python实现
- 要在Scikit learn中实现PCA,在应用PCA之前必须标准化/规范化数据。
- PCA是从sklearn. decomposition导入的。我们需要选择所需数量的主成分。
- 通常,n_components被选择为2以获得更好的可视化效果,但这取决于数据。
- 通过fit和transform方法,传递属性。
- 主成分的值可以使用components 检查,而每个主成分解释的方差可以使用explained_variance_ratio计算。
1.导入所有库
# import all libraries
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
2.加载数据
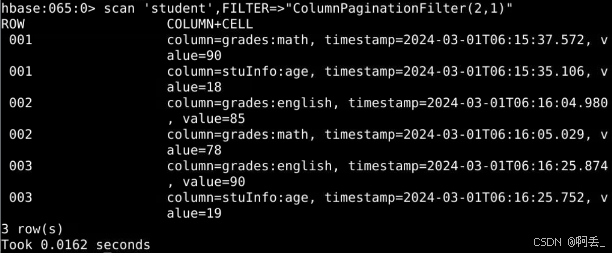
从sklearn. datasets加载breast_cancer数据集。很明显,数据集有569个数据项,具有30个输入属性。有两种输出类别-良性和恶性。由于有30个输入要素,因此无法将此数据可视化。
#import the breast _cancer dataset
from sklearn.datasets import load_breast_cancer
data=load_breast_cancer()
data.keys()
# Check the output classes
print(data['target_names'])
# Check the input attributes
print(data['feature_names'])
输出

3.应用PCA
- 在PCA之前标准化数据集。
- 从sklearn.decomposition导入PCA。
- 选择主成分的数量。
这里我们选择3。执行完这段代码后,我们知道x的维数是(569,3),而实际数据的维数是(569,30)。因此,很明显,利用PCA,维度的数量从30减少到3。如果我们选择n_components=2,则维度将减少到2。
# construct a dataframe using pandas
df1=pd.DataFrame(data['data'],columns=data['feature_names'])
# Scale data before applying PCA
scaling=StandardScaler()
# Use fit and transform method
scaling.fit(df1)
Scaled_data=scaling.transform(df1)
# Set the n_components=3
principal=PCA(n_components=3)
principal.fit(Scaled_data)
x=principal.transform(Scaled_data)
# Check the dimensions of data after PCA
print(x.shape)
输出
(569,3)
4.检查特征
principal.components_提供了一个数组,其中行数表示主成分的数量,而列数等于实际数据中的特征数量。 我们可以很容易地看到,当n_components被选择为3时,有三行。但是,与实际数据一样,每行有30列。
# Check the values of eigen vectors
# prodeced by principal components
principal.components_

5.绘制主成分图(可视化)
绘制主成分图以更好地显示数据。 虽然我们取了n_components =3,但这里我们分别使用前2个主成分和3个主成分绘制了一个二维图和三维图。对于三个主成分,我们需要绘制一个3D图。颜色显示原始数据集的2个输出类-良性和恶性。很明显,主成分在两个输出类之间显示出明显的分离。
plt.figure(figsize=(10,10))
plt.scatter(x[:,0],x[:,1],c=data['target'],cmap='plasma')
plt.xlabel('pc1')
plt.ylabel('pc2')

对于三个主成分,我们需要绘制一个3D图。x[:,0]表示第一主成分。类似地,X[:,1]和X[:,2]表示第二主成分和第三主成分。
# import relevant libraries for 3d graph
from mpl_toolkits.mplot3d import Axes3D
fig = plt.figure(figsize=(10,10))
# choose projection 3d for creating a 3d graph
axis = fig.add_subplot(111, projection='3d')
# x[:,0]is pc1,x[:,1] is pc2 while x[:,2] is pc3
axis.scatter(x[:,0],x[:,1],x[:,2], c=data['target'],cmap='plasma')
axis.set_xlabel("PC1", fontsize=10)
axis.set_ylabel("PC2", fontsize=10)
axis.set_zlabel("PC3", fontsize=10)

6.计算方差比
Explained_variance_ratio提供了主成分解释了多少变化的概念。
# check how much variance is explained by each principal component
print(principal.explained_variance_ratio_)
输出
array([0.44272026, 0.18971182, 0.09393163])