小罗碎碎念
群里今天有人提问Histolab相关的问题,作者在交流的过程中谈到了Slideflow,恰好我没有看过这篇文章,所以便有了这期推文。

| 作者角色 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | James M. Dolezal | Section of Hematology/Oncology, Department of Medicine, University of Chicago Medical Center | 芝加哥大学医学中心血液学/肿瘤学部 |
| 通讯作者 | Alexander T. Pearson | Section of Hematology/Oncology, Department of Medicine, University of Chicago Medical Center | 芝加哥大学医学中心血液学/肿瘤学部 |
文献速览
这篇文章介绍了Slideflow,一个用于数字病理学的深度学习工具库,它支持多种深度学习方法,提供全切片图像处理和实时可视化功能。
Slideflow支持多种深度学习方法,并提供了一个快速的全切片界面来部署训练好的模型。这个工具库包括用于全切片图像数据处理、高效染色归一化和增强、弱监督全切片分类、不确定性量化、特征生成、特征空间分析和可解释性的独特工具。Slideflow的全切片图像处理经过高度优化,能够在2.5秒内完成40倍放大的全切片平铺提取。
文章强调了Slideflow的灵活性和用户友好性,它提供了一个端到端的工具集,用于构建、测试、解释和部署深度学习模型,适用于各种研究目标。Slideflow支持Tensorflow和PyTorch框架,并且可以在多种硬件设备上实时可视化切片、预测、热图和特征空间特征。
此外,文章还讨论了Slideflow在实际数据集上的应用案例,包括用于预测头颈部鳞状细胞癌中人类乳头瘤病毒(HPV)状态的基准数据集。研究结果表明,使用Slideflow训练的模型在测试集上表现出了较高的准确性和敏感性。
最后,文章讨论了Slideflow的潜在优势和一些限制,并提出了未来开发路线图,包括多组学数据集成、细胞分割和分类能力的扩展、以及提高图形用户界面的无代码模型构建支持等。
代码&数据
- Slideflow GitHub 仓库:
- 链接:https://github.com/jamesdolezal/slideflow
- 作用:Slideflow的源代码库,用于获取Slideflow工具库的代码,进行安装、使用和进一步的开发。
- Docker Hub:
- 链接:https://hub.docker.com/r/jamesdolezal/slideflow
- 作用:提供了预构建的Docker容器,方便用户在隔离环境中部署和管理Slideflow的依赖。
- The Cancer Genome Atlas (TCGA):
- 链接:https://portal.gdc.cancer.gov/projects/TCGA-HNSC
- 作用:提供了头颈部鳞状细胞癌项目的全切片图像数据集,用于训练和测试Slideflow工具库中的深度学习模型。
一、背景
患者组织与肿瘤标本的病理切片在数字格式下的捕获与存储日益增多,其用途广泛。
深度学习模型在分析数字病理切片图像方面开创了新维度,使作者能够提取具有临床意义的信息[1]。这些模型不仅加速并提升了病理临床工作流程,而且能检测到人眼难以察觉的细微形态学特征,从而提高诊断的效率和准确性[2, 3]。
此外,深度学习模型允许直接从数字病理图像进行基因组亚型分类[4-7],并在患者风险分层[8]、预后[9]和治疗选择[10]方面显示出潜力。数字病理学还可能通过虚拟染色增加在资源匮乏环境中对苏木精-伊红(H&E)和免疫组化染色的可获取性,这些工具能够提高临床工作流程的效率并提供先进的诊断,这在训练有素的病理学家数量有限或完全缺失的情况下可能是无法实现的[11]。
尽管深度学习在数字病理学中的应用具有广泛前景,但开发复杂性和计算资源的获取仍是广泛采用的障碍。目前,对于作为分析平台的功能性开源软件有着日益增长的需求。
有效的软件设计对于数字病理学中的深度学习研究工具包至关重要。
模型性能随着提供训练数据的增多而提高[12],但数据存储要求可能会对数据集大小和实验范围施加实际限制。统一且高效的数据存储可以减少数据冗余并优化训练模型所需的计算资源。计算效率高的软件可以减少数据处理和存储需求,同时缩短训练时间,使不同设备能力的团队能够训练自己的模型或为新的研究目标访问预训练模型。
此外,旨在支持临床决策的深度学习模型必须是透明和可解释的[13]。解释模型如何得出决策以及与预测相关的确定性水平可以帮助建立临床医生的信任,这有助于促进这些工具在临床实践中的更广泛采用。无缝整合可解释性和不确定性量化的软件在推动这些深度学习工具的潜在临床应用方面具有显著优势。
随着计算工具在生物科学中变得越来越重要,确保它们对具有不同计算背景的研究人员可获取性是至关重要的。
这可以通过全面的文档、直观的代码设计和积极的项目开发来实现。在创建临床应用的分析工具时,还需考虑目标终端用户可能不具备广泛的计算专业知识。
图形用户界面(GUI)可以为这类用户提供一个易于访问的入口点,从而促进深度学习工具在临床环境中的部署。
在更广泛的机器学习文献中,有许多软件库可协助开发快速且健壮的深度学习应用,降低研究入门障碍,简化开发工作流程,并提高迭代和创新的速度。
然而,计算病理学研究有许多独特的流程,使得直接利用通用深度学习库具有挑战性,包括全切片图像处理、染色标准化和特定的病理学算法,如组织分割、细胞识别和多重实例学习。目前需要易于访问的软件库,提供常用的计算病理学工作流程,作为研究和开发的基础。
目前,大多数计算病理学研究论文要么分享针对特定研究应用定制的代码,这些代码不能立即泛化到其他数据集或研究问题,要么根本不分享代码[14]。
有几套现有软件库试图解决这个问题,为病理人工智能研究提供一个可泛化的工具包。其中一些库已多年未更新[15, 16],而其他库则继续积极开发并获得社区的支持和利用。
TIAToolbox是一个基于PyTorch的稳健的计算病理学库,提供全切片图像处理和染色标准化、基于瓦片的分类以及组织和核分割的工具[17]。TIAToolbox还提供了一个清晰且设计良好的代码库以及详细且用户友好的文档。尽管CLAM不是积极开发中的项目,但它是一个流行的基于PyTorch的深度学习仓库,包括全切片图像处理、使用在ImageNet上预训练的ResNet50模型进行特征提取以及基于注意力的多重实例学习[18]。
PathML是一个基于PyTorch的深度学习库,提供各种切片格式的全切片图像处理、用于核检测和基于弱监督的瓦片分类的模型架构以及详细的在线文档[19]。
DeepPath是一个基于Tensorflow的库,提供SVS切片的全切片图像处理和支持训练基于Inception-v3的弱监督瓦片分类模型[20]。
Histolab是一个针对SVS切片的全切片图像预处理库,旨在协助下游深度学习任务,因此不包括模型开发工具[21]。
最后,MONAI是一个主要用于放射学图像的基于PyTorch的深度学习框架,但其病理工作组正在寻求扩大对病理应用的支持[22]。这些库中没有一个提供可供有限或无编程经验的人使用的交互式用户界面,以灵活部署模型到全切片图像。
深度学习病理研究在过去几年中取得了巨大进步。
最先进的方法现在包括自监督学习、多重实例学习、生成对抗网络和不确定性量化。
为了最大化对研究者的相关性,为计算病理学研究者设计的库应提供高度灵活且高效的数据处理流程,支持新算法的快速实现,以及详细说明库如何随着新方法的涌现而扩展的文档。
设计一个灵活的软件平台,能够随着不断发展的领域增长和扩展,是一项具有挑战性的任务,但将为研究者带来众多好处,如减少软件开发时间、更容易访问最先进的算法,以及提高可重复性。
作者因此致力于开发一个从头开始构建的深度学习框架,注重灵活性、可扩展性和易用性,同时强调模型开发和交互式全切片图像模型部署。
认识到当前计算病理学工具的限制和未满足的需求,作者推出了Slidefow。
Slidefow是一个全面的Python包,旨在弥合这些差距,提供一套用户友好的工具,用于构建、测试、解释和部署深度学习模型,以应用于组织病理学。
Slidefow旨在克服计算效率、可访问性和解释性方面的挑战,同时赋能研究者和临床医生,充分利用不断发展的深度学习方法在数字病理学中的潜力。
二、技术概述
Slidefow是一个Python包,提供了一套深度学习工具库,这些工具库使用PyTorch和Tensorflow后端实现。它被开发用于为科学研究构建和部署深度学习组织病理学应用,提供端到端工具包,包括高效的数据处理、模型训练、评估、不确定性量化、可解释性以及在图形用户界面(GUI)中模型部署。
Slidefow包含一个全切片用户界面,Slidefow Studio,用于实时生成全切片图像的预测和热力图。Slidefow易于部署,其分发版本可在Python包索引(PyPI)上获得,预构建的Docker容器可在Docker Hub上获取。
2-1:全切片图像处理
Slidefow支持九种切片扫描仪供应商(见表1),并包括两个切片读取后端——cuCIM[23],这是一个高效的、基于GPU加速的TIFF和SVS切片读取框架,以及VIPS[24],这是一个基于OpenSlide的框架,增加了对其他切片格式的支持。

处理全切片图像(WSI)以进行下游深度学习应用的第一步是切片级遮罩和过滤,这一过程确定切片的哪些区域是相关的,哪些区域应该被忽略。可以使用Slidefow Studio提供的全切片可视化工具或使用QuPath[25]或Aperio ImageScope[26]等替代程序手动标注感兴趣区域(ROIs)。可以使用Otsu的阈值分割[27]进行组织检测,该过程遮罩背景区域。可以使用高斯模糊过滤[28]来遮罩笔迹和失焦区域,其参数可通过经验测试确定,并可供用户自定义。
此外,Slidefow包含一个用于自定义切片级遮罩的API,在线文档[30]中展示了一个使用此API应用DeepFocus[29]模型检测失焦区域的示例。
在切片级遮罩和过滤之后,WSIs被分割成更小的子区域。
图1展示了全切片图像平铺提取和图像处理的数据流程。

这个过程可以分为三个主要部分:
a. 初始切片处理和网格准备
- 切片注释:全切片图像可以被注释为感兴趣的区域(ROI),这样在后续分析中只包括切片的相关区域。
- 可选的切片过滤步骤:在这个阶段可以应用高斯模糊过滤和大津阈值(Otsu’s thresholding)等可选步骤,以去除背景或突出感兴趣的区域。
- 网格划分:将剩余的切片区域划分成网格,为平铺提取做准备。
b. 平铺提取过程中的数据流
- 平铺级别过滤:如果需要进行平铺级别的过滤,如白空或灰空过滤,可以使用给定位置的最高金字塔层级的低倍图像进行背景过滤。
- 图像提取:如果平铺通过过滤,提取全倍率图像,可选择调整大小以匹配目标微米尺寸,进行染色归一化,并转换为PNG图像、JPEG图像或Numpy数组。
- 数据存储:提取的平铺可以作为单独的文件保存到磁盘,或者缓冲到TFRecords中,以加快数据集的读取速度。
c. 从TFRecords读取时的数据流
- TFRecords缓冲:平铺图像以JPEG或PNG压缩格式缓冲在TFRecords中,并存储切片和位置元数据。
- 数据迭代:在数据集迭代过程中,数据被解码并转换为张量。
- 增强处理:可以应用增强处理,包括随机翻转/旋转、随机JPEG压缩和随机高斯模糊。
- 染色归一化:如果在平铺提取期间没有执行染色归一化,可以在实时迭代TFRecord数据集时执行此步骤。
- 图像标准化和标签转换:图像随后被标准化,切片名称使用提供的CSV注释文件或Pandas DataFrame转换为真实结果标签。
这个流程图是理解Slideflow工具库如何处理和准备全切片图像数据的关键,它涵盖了从原始图像到可用于深度学习模型训练的标准化数据的整个处理链。通过这种方式,研究人员可以有效地从大量的病理学切片中提取有用的信息,并利用深度学习模型进行分析和诊断。
图像数据可以使用JPEG(有损)或PNG(无损)压缩进行编码。
图像瓦片以网格形式提取,可以选择重叠或抖动进行数据增强。在瓦片提取期间,图像瓦片可以经历额外的过滤步骤,使用灰度空间或空白空间过滤,可能识别出Otsu阈值分割未能成功移除的高背景内容瓦片。
灰度空间过滤通过将图像转换为HSV色彩空间,将饱和度低于给定阈值的像素分类为灰色,并在灰色像素的比例超过预设阈值时,将瓦片作为背景丢弃。
空白空间过滤通过计算每个像素的亮度(红色、绿色和蓝色通道的平均值),将亮度高于给定阈值的像素分类为白色,并在白色像素的比例超过预设阈值时,将瓦片作为背景丢弃。默认过滤阈值通过先前的经验测试确定,并可由用户自定义。瓦片级背景过滤在较低的放大倍数下进行,以提升计算效率。
瓦片提取的有效光学放大倍数可以通过指定金字塔层级或以微米为单位的瓦片宽度来确定(见图2)。
图2展示了基于放大倍数和平铺尺寸(微米)的两种不同的平铺提取策略,并比较了它们在不同情况下的应用效果。

a. 在20倍有效光学放大倍数下的比较
- 切片1:内部金字塔图像存储有2.5倍、10倍、20倍和40倍的放大图像。
- 切片2:图像仅存储有2.5倍、10倍和40倍的放大图像,缺少20倍的层级。
- 基于放大倍数的提取策略:提取与目标光学放大倍数相匹配的图像金字塔层级的平铺。在这个例子中,由于切片2缺少20倍的层级,因此无法在20倍的放大倍数下提取平铺。
- 基于微米尺寸的提取策略:对于切片2,可以在40倍层级提取图像平铺,并调整尺寸以实现20倍的有效光学放大倍数。这种方法可以确保即使在缺少特定放大倍数层的情况下,也能提取出所需尺寸的平铺。
b. 在10倍有效光学放大倍数下的比较
- 切片1和切片3:两者都有10倍的层级,但由于扫描器的差异,它们的有效光学放大倍数略有不同,分别为10倍和10.5倍。
- 基于放大倍数的提取策略:从切片1和切片3提取的图像平铺会有不同的有效光学放大倍数,因为它们原始层级的放大倍数不同。
- 基于微米尺寸的提取策略:从切片3中提取10.5倍层级的图像平铺,并调整尺寸以匹配切片1的10倍有效光学放大倍数。这种策略确保所有提取的图像平铺具有相同的有效光学放大倍数,无论原始扫描层的放大倍数如何。
结论
- 基于放大倍数的提取:依赖于图像金字塔中特定放大倍数的可用性,如果缺少特定层级,则无法提取,且不同切片的相同放大倍数层可能代表不同的实际放大倍数。
- 基于微米尺寸的提取:提供了更大的灵活性和一致性,允许从最高的可用层级提取图像平铺,并调整尺寸以匹配目标放大倍数,确保不同切片之间的一致性。
如果使用金字塔层级,图像瓦片将在指定的像素大小下提取。并非所有切片都有相同的光学放大倍数作为金字塔层,因此这种方法可能需要跳过一些切片。此外,这种方法将下游分析可探索的放大倍数限制为每个切片中可用的放大倍数。
另一方面,如果指定了以微米为单位的瓦片宽度,图像瓦片将从最近的更高放大倍数的金字塔层提取并缩小到目标微米宽度,允许研究人员精确指定有效光学放大倍数。因此,基于微米的瓦片提取更为灵活。
使用cuCIM时,图像使用双线性插值进行缩放,使用VIPS时,图像使用Lanczos插值[32]进行缩放。瓦片提取经过重度优化并使用多进程加速,使得在40倍放大倍数下全切片瓦片提取仅需2.5秒/张。快速的瓦片提取使研究人员能够快速实验不同的放大倍数。
在提取瓦片后,将自动生成包含WSIs与瓦片遮罩叠加的图像和每个WSI选定的瓦片的视觉总结报告,使研究人员能够快速评估遮罩、过滤和提取图像的质量。
这一步骤可以识别潜在的数据集问题,如失焦切片、背景或伪影移除不理想或ROI质量低下。Slideflow Studio是一个包含在Slideflow中的交互式全切片图形用户界面(GUI),可用于实时预览切片遮罩、背景过滤和染色标准化设置。
交互式可视化有助于快速确定最佳切片处理参数,如果需要进一步调整这些设置。
2-2:组织和细胞分割
Slideflow提供了多种工具,用于在全切片图像上进行组织和细胞的分割。
这些工具包括基于深度学习的分割模型,可以在Slideflow Studio中进行训练和部署。Slideflow Studio允许用户交互式地调整分割参数,实时查看结果,并导出分割掩码以供进一步分析。
2-3:模型训练与评估
Slideflow支持使用PyTorch和Tensorflow框架进行深度学习模型的训练和评估。
它提供了数据加载器、预处理管道和训练循环的抽象,简化了模型开发过程。用户可以通过配置文件自定义训练参数,包括优化器、学习率和损失函数。
Slideflow还提供了模型评估工具,包括交叉验证和混淆矩阵分析,以及不确定性量化和模型解释性的方法。
2-4:不确定性量化与解释性
Slideflow集成了不确定性量化和模型解释性的功能,这对于临床决策支持尤为重要。
不确定性量化可以帮助识别模型预测中的不确定性,而模型解释性技术如梯度加权类激活映射(Grad-CAM)[33]可以提供视觉证据,说明模型是如何达到其预测的。
这些功能在Slideflow Studio中均可访问,使得研究人员和临床医生能够更好地理解模型的预测行为。
2-5:模型部署
Slideflow提供了一个易于使用的界面,用于将训练好的模型部署到全切片图像上。
通过Slideflow Studio,用户可以加载模型,并在全切片图像上生成预测和热力图。
此外,Slideflow支持将模型部署为Web服务,使得模型预测可以远程访问,便于在多地点和多设备上使用。
2-6:染色标准化和增强
数字苏木精-伊红(H&E)染色标准化用于帮助减少切片之间染色颜色和强度差异所导致的批次效应,可以在瓦片提取期间或模型训练实时进行。
可用的染色标准化方法包括Reinhard[33]、Macenko[34]和Vahadane[35],以及Reinhard的遮罩变体(仅在非白色区域应用标准化)和Reinhard和Macenko标准化器的快速变体,其中亮度标准化步骤被禁用。
Reinhard和Macenko标准化器具有原生Numpy/OpenCV、Tensorflow和PyTorch实现,以提高计算效率并支持GPU加速。染色标准化器包括几个默认参考拟合,可以可选地拟合到用户定义的图像。
实时染色增强可以在使用Reinhard或Macenko标准化器时在训练期间执行。
Slideflow包括一种新颖的染色增强方法,通过动态随机化染色标准化目标,使用预设的标准差围绕标准化拟合值。由于随机化的染色矩阵目标围绕标准化值中心,因此结果是标准化和增强的组合。
这种方法与Tellez等人描述的染色增强方法不同,他们在解卷积染色矩阵空间中进行染色增强,而没有进行标准化[36]。
此外,Slideflow还包括一个选项,在Macenko染色标准化过程中使用与相应WSI相关的上下文信息。使用上下文标准化时,利用一张WSI的染色图案来指导一个图像瓦片的标准化。给定图像瓦片的解卷积H&E通道使用从上下文图像计算的最大H&E浓度进行标准化,而不是从要转换的图像计算这些最大浓度。
当使用此选项时,上下文图像是一个WSI的缩略图,其中去除了背景和ROI外的区域,以防止笔迹和其他伪像干扰染色解卷积。上下文标准化不建议在伪像去除不理想或ROI不可用时使用。
2-7:训练弱监督、基于瓦片的模型
Slidefow包括多种工具,用于训练WSI的深度学习分类、回归和事件时间模型。
弱监督、基于瓦片的模型使用深度学习图像分类器为切片中的所有瓦片生成预测,并通过对瓦片预测进行平均来计算最终的切片级预测(见图3)。

图3概述了基于平铺的深度学习模型的方法,包括训练和评估两个阶段:
训练阶段 (a部分)
-
平铺提取:
- 在进行任何必要的切片处理(如注释、过滤等)之后,从全切片图像中提取平铺。
- 提取的平铺被缓冲到TFRecords中,这是一种高效的数据存储和读取格式,便于后续处理。
-
数据读取与增强:
- 在训练过程中,从TFRecords中读取平铺。
- 对平铺进行数据增强,如随机翻转、旋转等,以提高模型的泛化能力。
- 进行染色归一化处理,以消除不同切片之间的染色差异。
- 对平铺进行标准化处理,使其符合模型输入的要求。
-
弱监督学习:
- 使用弱监督方法训练模型,即每个平铺的真实标签由其对应切片的真实标签确定。
- 这种方法允许在只有切片级别标签的情况下训练模型,而不需要平铺级别的详细标注。
评估阶段 (b部分)
-
直接提取与处理:
- 在评估阶段,直接从切片中提取平铺,无需将其缓冲到TFRecords中。
- 对提取的平铺进行必要的处理,如染色归一化。
-
生成预测:
- 对每个切片的每个平铺生成预测。
- 这些预测可以反映模型对每个平铺的分类或回归结果。
-
计算切片级别预测:
- 将所有平铺的预测结果平均,得到最终的切片级别预测。
- 这种方法假设切片中的所有平铺都对最终预测有所贡献。
-
可视化:
- 将平铺级别的预测结果可视化为热图,这有助于直观地展示模型在切片上的预测结果。
- 热图可以突出显示模型预测高置信度或高重要性的区域。
尽管这种方法在高度异质性肿瘤或显著形态学特征稀疏的情况下可能有限,但它已被证明是多种生物应用的有效方法[6, 37-39]。除了平均外,Slidefow还支持使用各种聚合函数计算切片级和患者级预测,以及任意用户定义的函数。
Slidefow包括数据集组织工具,以轻松支持用于生物标志物开发的标准化训练、验证、测试范式。
首先指定一个保留的测试集,可以通过手动或使用几种数据集分割工具来完成。剩余数据集可以采用几种方法进行分割,包括固定分割、自助法、k折交叉验证和站点保留k折交叉验证[40]。
模型架构、损失函数和训练超参数随后被配置;包括一些预配置的模型架构的列表,见补充文件1:表1。

Slidefow还支持使用Tensorflow或PyTorch的定制模型和损失函数。
模型可以训练以产生单一分类、多分类、连续或事件时间结果。还可以使用额外的任意输入,如临床变量,通过在后卷积层进行多模态融合,训练多模态模型。
超参数可以使用网格搜索或贝叶斯超参数优化进行调整,后者使用SMAC3[41]包。超参数搜索空间可以轻松定制,并提供了一些预配置的搜索空间。
在训练期间,图像可以进行增强以拓宽训练领域并促进泛化(见图1C)。图像可以进行随机角度旋转和随机水平或垂直翻转,随机JPEG压缩,随机高斯模糊和染色增强。当使用Tensorflow时,图像标准化为均值为0、标准差为1,而使用PyTorch时,图像标准化为范围为0-1。
默认情况下,使用小批量平衡确保每个批次中所有切片的平等代表和所有类别的平等代表(见补充文件1:图2)。
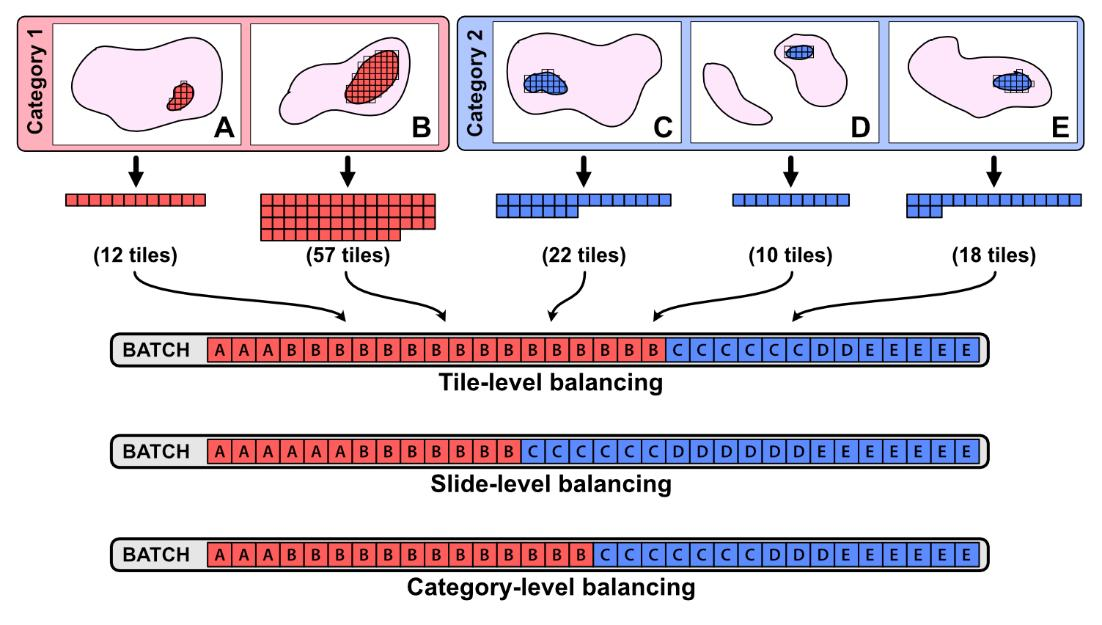
在训练过程中,会过采样切片和类别以实现此平衡。小批量平衡可以自定义或禁用。
在训练期间,一个epoch被定义为训练数据集中的图像瓦片总数除以批量大小。
用户可以指定在epoch中间进行验证检查的间隔。早期停止可以配置为当损失或准确率的指数移动平均值平稳时触发,或者可以训练预定义数量的epoch。
为了计算效率,训练可以分布在多个GPU上进行。训练进度可以使用Tensorboard[42]在本地或使用Neptune.ai[43]进行远程监控。

已提供了一些预训练的分类模型,这些模型是在甲状腺癌(BRAF-RAS基因表达评分)、乳腺癌(雌激素受体[ER]阳性与ER阴性)和肺癌(腺癌与鳞状细胞癌)上训练的,并已发布在Hugging Face[44-46]。
综上所述,Slidefow是一个全面且高效的Python包,专为数字病理学研究设计。它提供了从数据预处理到模型部署的端到端工具链,支持高效的数据处理、模型训练、评估、不确定性量化、可解释性,以及图形用户界面中的模型部署。
Slidefow通过提供多种工具来处理全切片图像,包括组织检测、切片级遮罩、背景过滤、染色标准化和增强,以及模型训练和部署,简化了深度学习在组织病理学中的应用。
此外,Slidefow还包括数据集组织工具,支持标准化的训练、验证、测试范式,并提供了预训练的分类模型,可以加速新研究项目的启动。Slidefow的灵活性和易用性使其成为研究人员和临床医生的强大工具,有助于推动深度学习技术在数字病理学领域的进一步发展和应用。
2-8:弱监督瓦片基模型的评估
在训练后,性能指标将从验证数据集中自动计算,包括准确性、接收者操作特征曲线下面积(AUROC)和平均精度(AP)。
切片级预测是通过将一个切片的所有瓦片的一元预测进行平均计算得出的,而患者级预测则是通过对给定患者所有切片的瓦片级预测进行平均计算得出的。瓦片级、切片级和患者级预测在训练和评估过程中都被保存,如果需要,研究人员可以计算自定义指标。保存的模型还可以应用于保留的测试集,计算测试集指标,或者应用于单个切片进行推断。
预测热图是通过在切片上叠加瓦片级预测生成的,也可以为单个切片或多个切片生成热图。热图计算通过并行化的瓦片提取和多进程加速。热图可以渲染并导出为图像,或者在Slidefow Studio的全切片图形用户界面中以实时导航方式交互式查看。
2-9:不确定性量化
在医学AI应用中,可靠的不确定性量化对于临床决策制定和潜在的患者安全至关重要[47, 48]。
Slidefow支持使用蒙特卡罗丢弃范式[49]进行不确定性估计。采用这种方法,模型在训练和推断过程中都包含丢弃层。在推断过程中,单个图像在网络中进行多次前向传播,结果分布代表最终预测(均值)和不确定性(标准差)。
瓦片级不确定性可以转换为切片级不确定性,并用于后续的置信度阈值处理,使用作者之前描述的不确定性阈值算法[31]。
简而言之,从一组验证预测中确定最佳不确定性阈值,低于该阈值的预测与高于该阈值的预测相比,更可能是正确的。然后丢弃不确定性高于该阈值的预测。这个阈值首先在瓦片级别确定,然后在切片级别确定。
使用嵌套交叉验证确定给定训练数据集的阈值,以防止数据泄露。这种不确定性估计和置信度阈值处理方法提高了高置信度预测的准确性,并防止了域迁移。
2-10:图像特征和特征空间分析
将图像转换为特征向量提供了一种进行特征空间分析和更先进的、基于整个切片的分类模型(如多重实例学习(MIL))的途径。
Slidefow提供了几种处理图像瓦片为数值向量的树特征生成方法:预训练的网络、微调的分类器以及自监督学习(见图4)。
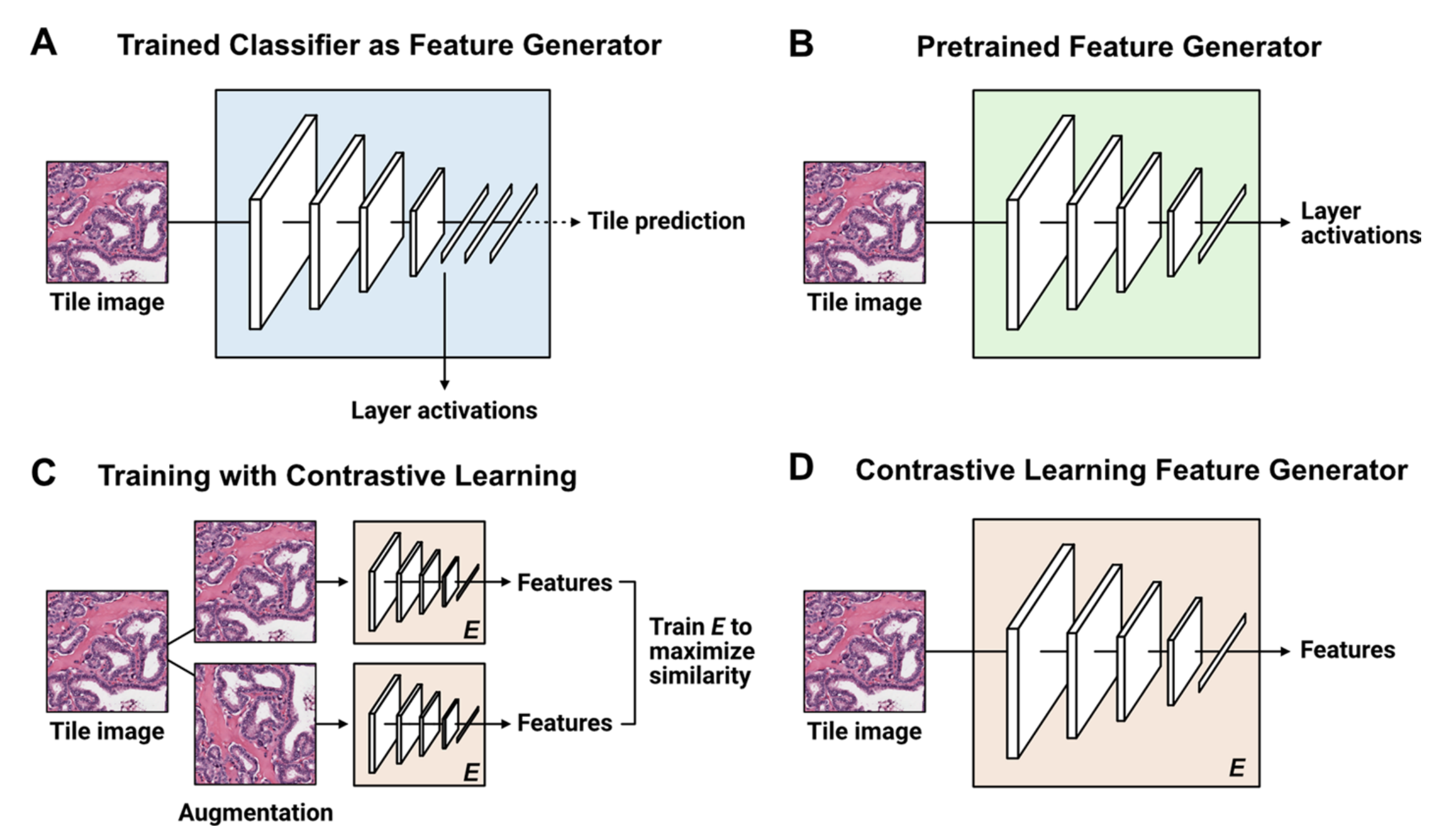
a. 使用训练好的分类器作为特征生成器
- 方法:通过指定神经网络中的某一层,利用这层的激活值来生成特征。
- 应用:这种方法可以将已经训练好的分类器重新用于特征提取,无需从头开始训练模型,适用于快速实现和部署。
b. 使用预训练模型作为特征生成器
- 通用预训练模型:如在ImageNet上预训练的模型,这些模型已经在大规模数据集上学习了丰富的特征表示。
- 病理学特定预训练模型:如CTransPath,这些模型针对病理学图像进行了优化,能更好地捕捉病理相关的特征。
- 优势:利用预训练模型可以减少训练时间和资源消耗,同时利用模型在相关任务上预训练的知识。
c. 自监督对比学习(SimCLR)
- 方法:使用自监督学习方法训练特征生成器,不需要类别标签。
- 原理:通过设计一种损失函数,使得同一图像的不同变换版本在特征空间中更接近,而不同图像的特征则更远离。
- 应用:这种方法特别适用于缺乏标注数据的情况,可以从未标注的病理学图像中学习有效的特征表示。
d. 从自监督学习训练的模型中计算特征
- 步骤:
- 使用自监督学习方法(如SimCLR)训练模型。
- 使用训练好的模型提取图像特征。
- 优势:自监督学习训练的模型能够捕捉到图像的内在结构和模式,生成的特征对于多种下游任务都具有较好的泛化能力。
对于所有方法,特征可以计算单个图像瓦片、单个切片或从TFRecords读取的图像瓦片。
对于将图像转换为特征向量,可以使用多种预训练的网络。Slidefow包括一个API,用于计算在ImageNet上预训练的模型的任意神经网络层的激活,通过指定架构名称和层名称。如果指定了多个层,每个层的激活都将被计算并连接。
还有一个API用于轻松使用预训练的、特定于病理学的CTransPath[50]和RetCCL[51]网络来生成特征。Slidefow微调的分类器也可以用于特征生成,计算任意神经网络层的激活。此外,还有一个API用于轻松训练自监督学习模型SimCLR,通过对比预训练提供另一种特征生成途径。
一旦为图像瓦片数据集计算了特征,Slidefow提供了多种工具,用于灵活地进行维数减少和特征空间可视化,使用UMAP[52]。
UMAP图可以快速生成,并标记结果、临床变量和可能的混杂因素,如站点。相应的图像瓦片可以叠加到UMAP图中以帮助解释,作者称之为马赛克图。马赛克图是通过将给定的UMAP投影分割成网格(默认50×50),然后为网格空间中的任何点绘制相应的图像瓦片生成的。在网格空间中选择瓦片可以是随机的(默认),也可以选择距离该网格空间质心最近的瓦片。
马赛克图可以与标记的UMAP一起查看,提供关于形态学图像特征、感兴趣的结果和其它相关临床因素之间关系的见解。这个过程还可以帮助识别潜在的混杂和偏差来源。马赛克图可以导出为高分辨率图形,也可以在Slideflow Studio中以交互式方式查看。
2-11:弱监督多实例学习
基于瓦片的整张切片分类模型可能不适用于特征稀疏或异质、或无法获得病理学家标注的感兴趣区域的切片数据集。
基于注意力的多实例学习(MIL)模型提供了一种弱监督整张切片分类的理论上仍然稳健的方法,即使在相关特征稀疏或感兴趣区域不可用的情况下也是如此[53]。这些模型通过图像瓦片特征向量的包来生成预测,模型将学会通过注意力加权来忽略不具信息性的图像瓦片。这些模型的性能在很大程度上取决于从图像瓦片中计算的特征的质量。
Slideflow可以训练三种类型的MIL模型,包括:
- 传统的MIL[54]
- 基于注意力的MIL[55]
- 约束注意力的聚类MIL(CLAM)[18]
提供了一个API,用于快速将图像瓦片转换为特征向量,并从这些生成的特征训练MIL模型。
训练使用FastAI框架[56]执行,包括余弦退火学习率调度或一周期学习率调度的选项。对于CLAM,提供了使用原始发布的训练循环而不是FastAI训练器的选项。训练好的MIL模型可以用于生成WSI的注意热图,突出显示加权高的区域的切片。与基于瓦片分类模型的预测热图类似,注意热图可以生成并导出为高分辨率图像。
2-12:生成对抗网络
越来越多的证据表明,生成对抗网络(GANs)在病理学应用中具有潜在的实用性。
GANs可以生成逼真的合成组织病理学图像,这些图像已被用于训练增强、染色和颜色标准化、图像增强和解释性。
Slideflow包括一个API,用于使用StyleGAN2[58]和StyleGAN3[59],以及可选的从TFRecords中保存的图像瓦片进行类条件训练。模型架构、训练范式和配置选项都与它们的原始实现相等。
该API提供了一种方法,用于从已存储在TFRecords中的预处理图像训练这些GANs,而不需要额外的数据处理或不同的格式,并且还提供了一个接口,用于使用训练好的分类器计算生成图像的预测。
对于类条件GANs,提供了一些工具,用于使用特征空间嵌入插值从中间类生成图像。要生成两个类之间的图像,作者计算每个类的相关类嵌入,然后进行线性插值以实现中间嵌入。
这些中间嵌入可以用于类或层混合应用[60]。从训练好的GANs生成的图像可以导出为原始PNG或JPG图像,保存为TFRecord格式,或使用Slidefow Studio实时可视化。
树预训练条件GANs,在甲状腺癌(基于BRAF样与RAS样基因表达)、乳腺癌(基于雌激素受体[ER]阳性与ER阴性)和肺癌(基于腺癌与鳞状细胞癌)上训练,已在Hugging Face[61-63]上发布。
2-13:模型解释性
可解释的人工智能(XAI)方法是医学成像研究中的一个日益重要的组成部分。
这些技术可以提供有关模型学到的图像特征的见解,支持模型预测的生物学合理性,并提高模型信任度[13]。Slidefow包括四种模型解释性方法:
- 热图
- 马赛克图
- 基于梯度的像素归因(显著性图)
- 条件生成对抗网络(cGANs)
热图,包括预测热图和注意热图,为快速评估与最终预测相关的WSI区域提供了一个途径。上述描述的热图可以计算单个切片或多个切片的数据集,并且可以导出为高分辨率图像或以交互方式查看。
马赛克图提供了一种在任意神经网络层进行特征空间探索的工具。通过病理学家的解释,它们可以突出显示结果变量与形态学特征之间的关联,并揭示不同类别之间图像特征的空间关系。
基于梯度的像素归因方法突出显示了图像中与给定的神经网络模型预测相关的像素。这些归因热图,或显著性图,可以通过多种提供的方法计算,包括Grad-CAM[64]、标准梯度[65]、综合梯度(及其变体)[66]和XRAI[67]。显著性图可以以原始热图的形式显示,也可以作为关联图像的覆盖层。包括了一些实用函数,用于快速比较不同的显著性图方法。显著性图也可以在Slidefow Studio中以实时交互方式查看WSI。
cGANs为训练好的神经网络模型提供了一种数据级别的解释性方法,使用作者最近描述的范式[60]。cGANs生成的合成组织病理学图像可以展示与结果类别相关的形态学特征,并提供关于无法用显著性图定位的大规模组织病理学特征的重要性,例如结构、基质、胶体和染色模式的不同。
2-14:组织和细胞分割
Slidefow还提供用于构建和使用组织和细胞分割模型的工具。
可以使用各种架构(包括U-Net、DeepLabV3、FPN等)从标记的区域兴趣中训练二进制和多类组织分割模型,这些架构由Iakubovskii[68]实现。
在训练期间,使用用户定义的微米/像素放大倍数的全切片缩略图和配对的区域兴趣遮罩进行随机裁剪、翻转和角度旋转的增强。一旦训练完成,组织分割模型可以部署用于生成区域兴趣或用于切片级遮罩和质量控制,通过程序化接口或Slidefow Studio。
一个预训练的肿瘤识别组织分割模型,在Te Cancer Genome Atlas[69]上的8,122张切片和配对的病理学家注释的区域兴趣上训练,已在Hugging Face[70]上提供。
Slidefow使用Cellpose[71]进行整张切片的细胞识别和分割。
可以使用预训练或用户训练的模型生成细胞分割掩码和中心点,通过程序化接口或Slidefow Studio。
细胞直径以微米配置,而不是像素,以提高跨不同放大倍数切片的一般化能力。细胞分割以瓦片方式应用于整张切片图像,分割掩码拼接在一起形成整张切片掩码。
掩码和中心点可以用于指导瓦片提取,使每个提取的瓦片代表一个检测到的细胞/核,或导出为NumPy格式以供下游使用和进一步分析。
2-15:全切片可视化与Slidefow Studio
Slidefow包括一个可视化工具,Slidefow Studio,用于交互式查看WSI、焦点预测、热图、显著性、不确定性和马赛克图。
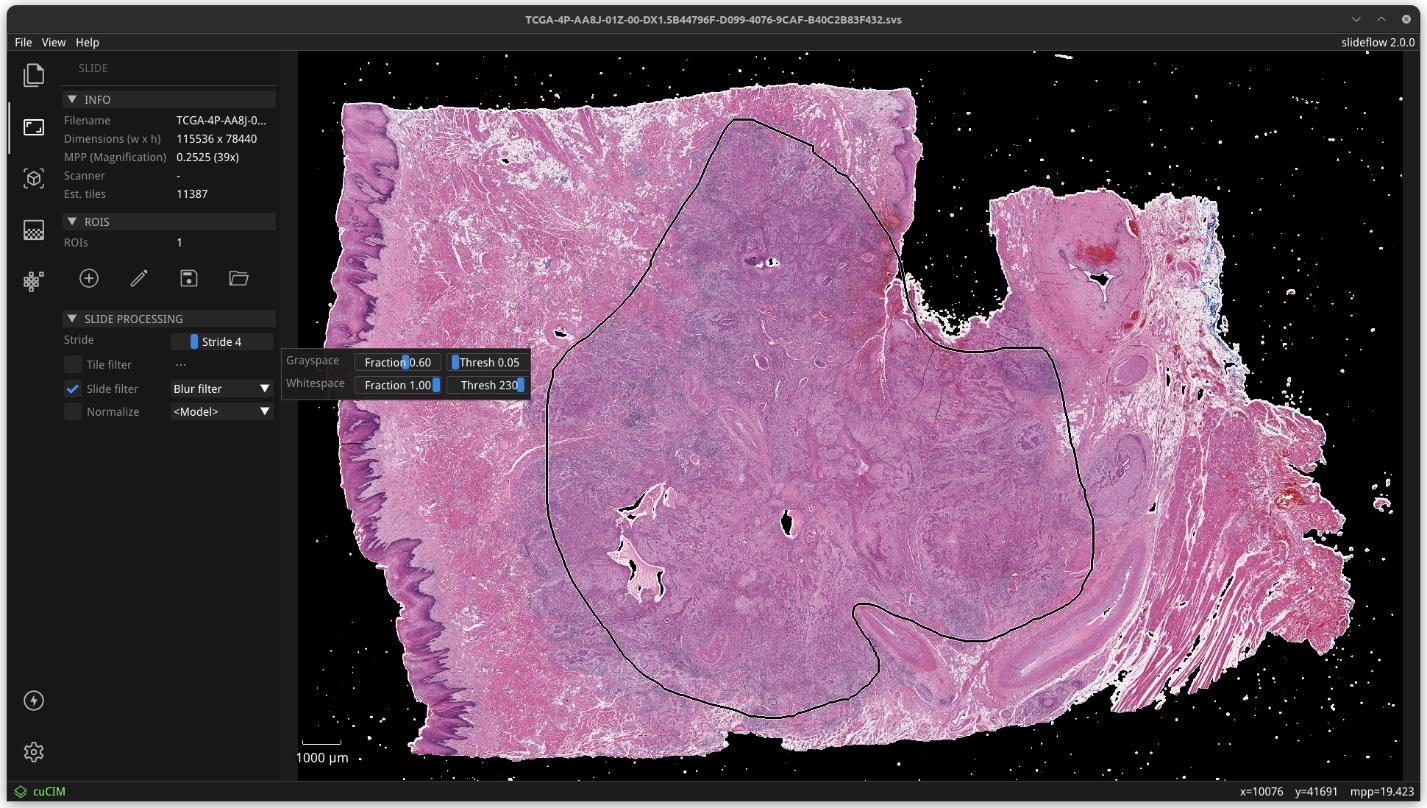
使用cuCIM或VIPS读取幻灯成像数据,并通过多进程加速,使用OpenGL渲染。Slide处理设置,如Otsu的阈值分割、灰度空间过滤、染色标准化等,可以实时预览以帮助快速确定最佳幻灯处理参数(见补充文件1:图4)。ROI注释可以加载、编辑和添加,使用套索选择工具。
一旦加载了模型,在幻灯上右键点击将显示一个预览窗口,显示该位置提取的图像瓦片,以及染色标准化前后的图像(如果适用)(见图5)。

通过基于梯度的像素归因生成的显著性图也可以以热图的形式显示,或者作为提取的图像瓦片上的覆盖层。瓦片级预测和不确定性显示在控制面板中。一旦加载了模型,就可以计算、自定义和显示全切片预测热图。
在性能设置中可以启用“低内存”模式,以减少内存消耗,但计算热图的速度较慢。在计算热图后,最终切片级预测(如果适用)将显示在控制面板中。
马赛克图也可以在Slidefow Studio中以交互方式查看(见补充文件1:图5)。
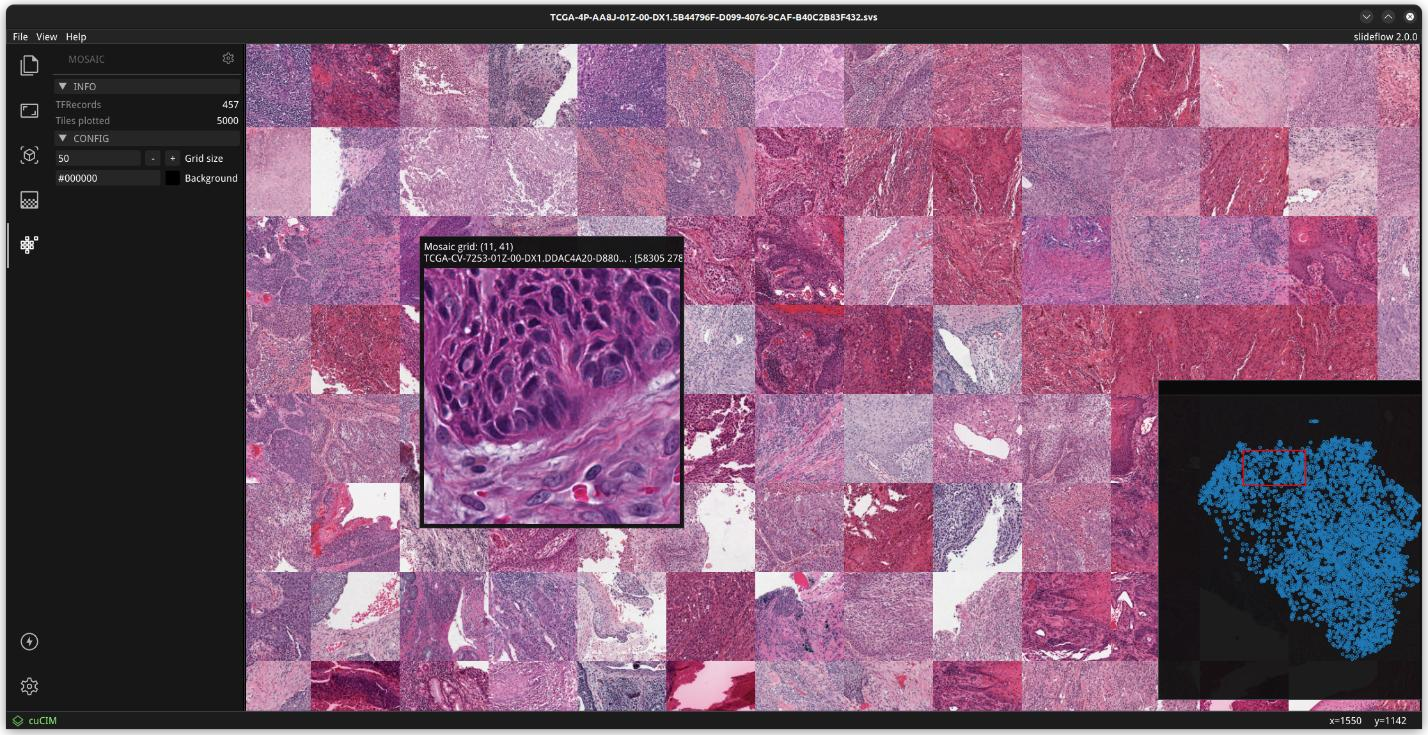
与静态图像相比,这个界面允许通过交互式缩放更仔细地检查高分辨率的图像瓦片,并允许动态修改马赛克网格大小。右下角的一个弹出窗口显示了相应的UMAP图,其中红色框指示当前在视图中显示的图的部分。当鼠标悬停在图像瓦片上时,将显示来自该瓦片位置的幻灯对应部分的更大部分,揭示了周围的形态学上下文。
综上所述,Slidefow为医学成像研究提供了一套全面的工具,包括模型解释性、组织和细胞分割以及全切片可视化。这些工具通过提供模型预测的透明度和解释性、精确的分割和定位以及直观的全切片图像查看,有助于研究人员和临床医生更好地理解数据,并做出更准确的诊断和治疗决策。
Slidefow Studio作为一个交互式的全切片图形用户界面,使得这些复杂的过程更加直观和易于操作,从而提高了研究的效率和可访问性。
2-16:程序化接口
Slidefow可以通过Python包索引(PyPI)进行安装,预构建的Docker容器可用于更轻松地管理依赖关系。
一旦安装,使用Slidefow的第一步是创建一个项目,以促进原始数据、处理数据、保存的模型和实验结果的组织。
项目是通过slidefow.Project类创建和加载的。通过与项目关联的dataset配置JSON文件定义全切片图像、病理学家ROI注释和处理后的TFRecords的位置。可以提供基于CSV文件或Pandas DataFrame的地面实况患者和切片级别的诊断和临床结果。
为了测试和功能探索,提供了几个预配置的项目示例,包括肺癌、乳腺癌和甲状腺癌;当使用时,全切片图像和相关临床注释将自动从公共仓库下载。
大多数功能,如训练分类模型、自监督学习、特征生成或GAN训练,都需要在给定放大倍数和像素分辨率下从全切片图像中提取的处理图像瓦片。
数据组织和处理由slidefow.Dataset类监督,可以通过使用Project.dataset()方法并指定所需的放大倍数和目标像素大小来生成其实例。与项目关联的临床注释会自动传递给数据集,并可用于数据集过滤。可以使用Dataset.split()轻松地将数据集分割为训练、测试和验证子集。
全切片图像处理和瓦片提取是通过Dataset.extract_tiles()方法执行的,生成的TFRecords会根据项目数据集配置自动组织和存储。一旦提取瓦片,可以使用Dataset.tensorfow()和Dataset.torch()方法分别轻松创建一个tensorfow.data.Dataset或torch.utils.data.DataLoader,该方法动态读取和处理来自TFRecords的成像数据。
分类模型使用slidefow.ModelParams类进行配置,该类定义了模型架构和训练超参数。
要训练一个模型,使用Project.train(),并提供一个ModelParams对象、一个训练和验证数据集以及临床注释中包含地面实况标签的列名。保留的测试集可以使用Project.evaluate()进行评估,并为切片生成预测可以使用Project.predict()。
在线文档包括关于训练的更多细节,包括逐步教程、使用自定义模型和损失函数的说明,以及带有ROI标签的强监督模型的训练。
文档还包含有关项目配置、数据集管理、多实例学习、特征空间分析、生成对抗网络和分割的程序化接口的进一步描述[30]。
2-17:支持的硬件和软件环境
Slidefow是一个需要Python 3.7或更高版本的Python包,并需要Tensorflow或PyTorch。
一些功能,如GANs和组织分割,需要PyTorch。模型训练需要一个基于Linux的操作系统,如Ubuntu或RHEL/CentOS,并在专用GPU的帮助下大大加速。可以使用交互式界面在各种操作系统和硬件环境中部署训练好的模型。
此用户界面已在基于Linux的系统、Windows、macOS(Intel和Apple芯片)以及Raspberry Pi和Jetson Nano等ARM设备上进行了测试。建议使用专用GPU进行模型部署,但不是必需的。
三、结果
Slidefow提供的各种工具已被用于许多不同的研究目标,包括基因表达预测[6]、预后[76]、不确定性量化[31]、偏差和批次效应的识别[40]、药物响应预测[77]以及生成合成组织病理学用于模型解释性[60]。
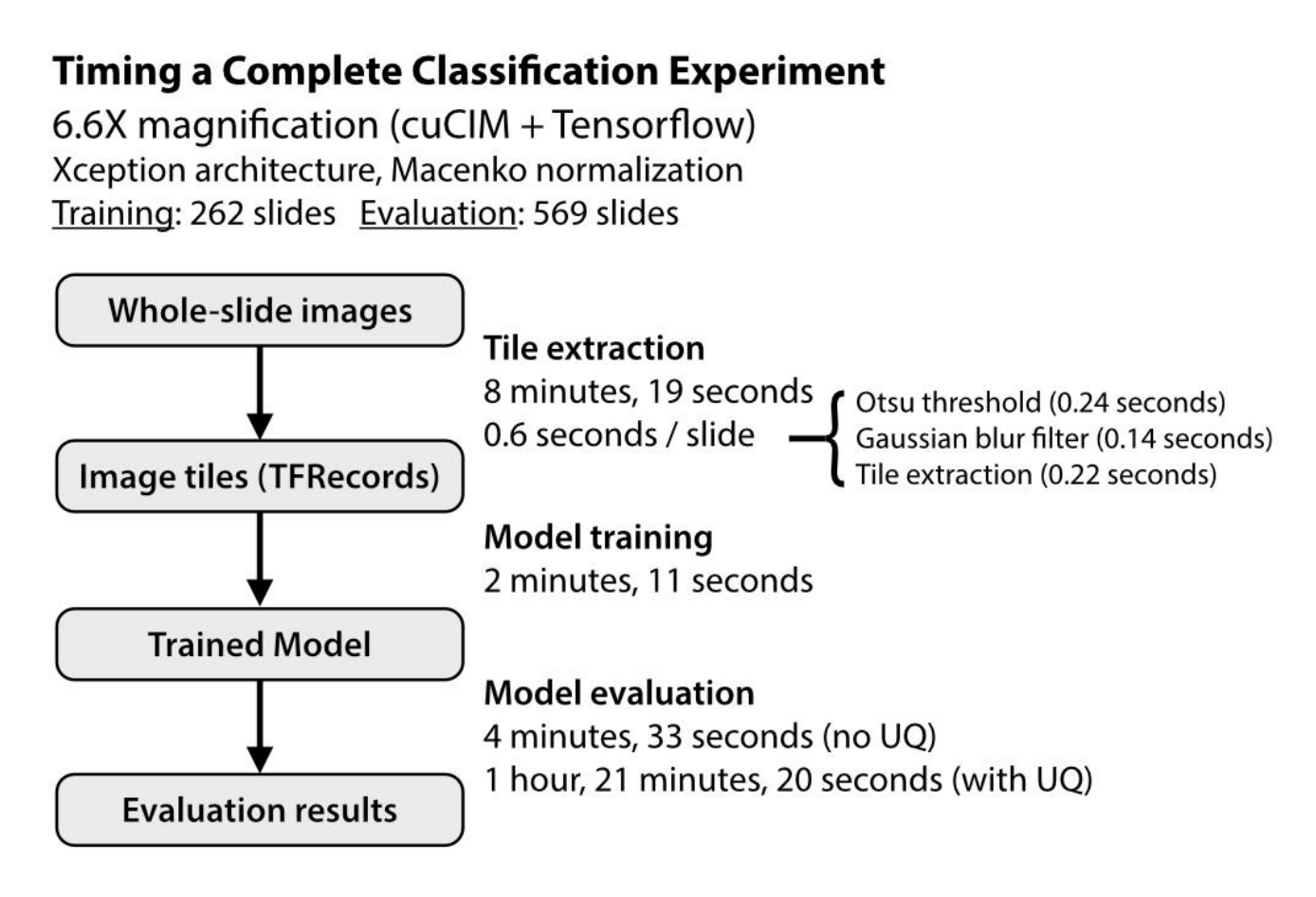
为了展示这些工具在实际数据集上的应用,作者将展示在头颈鳞状细胞癌的人乳头瘤病毒(HPV)状态预测基准数据集上的结果。训练/验证数据集包含来自芝加哥大学的262名患者(151名HPV阴性,111名HPV阳性),而保留的外部数据集包含来自癌症基因组图谱(TCGA)的26个网站的459名患者(407名HPV阴性,52名HPV阳性)。
所有患者都有一张相关的全切片图像。
3-1:切片处理
病理学家为所有切片标注了围绕肿瘤区域的ROI,除了整个样本被确定全部为肿瘤的情况。
在这个数据集上探索了Otsu阈值分割和Gaussian模糊过滤。在某些情况下,Otsu的阈值分割突出了笔迹作为前景组织,因此除了ROI外,还使用这两种背景过滤方法对所有切片进行了处理。
图像瓦片在299像素和7微米大小的范围内提取,范围从76微米(40倍放大)到1208微米(2.5倍)。使用cuCIM后端,瓦片提取速度在614到2091瓦片/秒之间(0.38–25切片/秒)(图6A和B)。
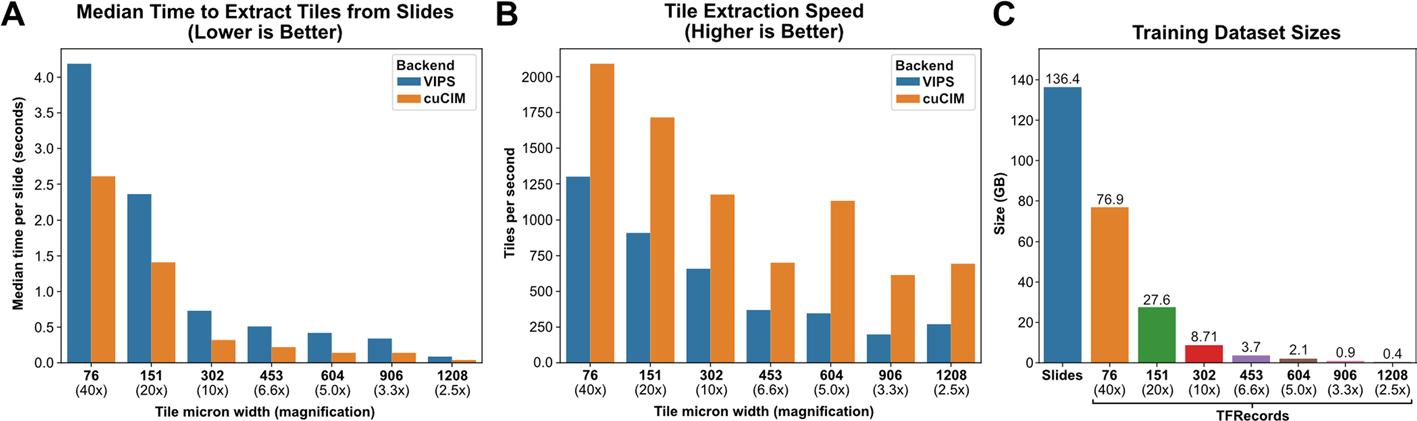
为了比较,还使用VIPS进行了瓦片提取,瓦片提取速度在198到1300瓦片/秒之间(0.24–11.5切片/秒)。Otsu的阈值分割每张切片增加了0.30±0.12秒,Gaussian模糊过滤每张切片增加了0.50±0.93秒。使用cuCIM进行瓦片提取比使用VIPS快1.6–3.3倍(图6A,B)。
相关瓦片提取PDF报告的示例页面见补充文件1:图6和7。

TFRecord缓冲允许Tensorflow以每秒11,453张图像的速度迭代数据集,PyTorch以每秒6,784张图像的速度迭代数据集。缓冲数据集大小见图6C。
对基于瓦片的背景过滤进行了比较实验,但没有用于下游分析(图7)。
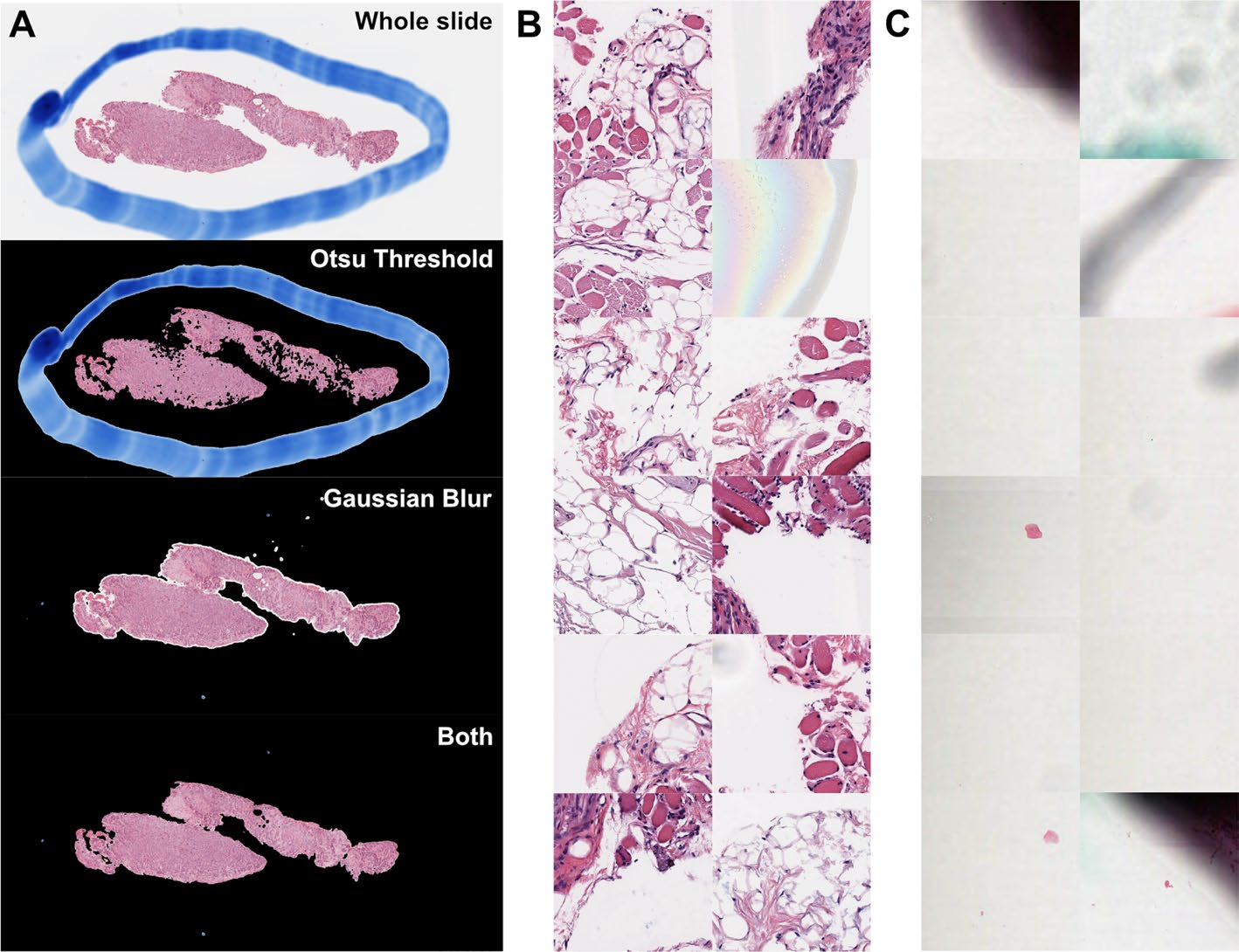
Otsu的阈值分割比灰度空间过滤识别出6.4%±8.2%更多的背景瓦片。Otsu的阈值分割移除但灰度空间过滤未移除的图像瓦片通常包括边缘瓦片或背景内容异质性的图像,如脂肪或基质(图7B)。
在绝大多数情况下(85.2%的切片),灰度空间过滤和空白空间过滤识别的背景差异<5%。在12.6%的情况下,灰度空间过滤移除了空白空间过滤移除的10.2%的背景。在2.2%的情况下,空白空间过滤未能移除任何背景瓦片。
比较了三种染色标准化方法及其变体,如图8所示。
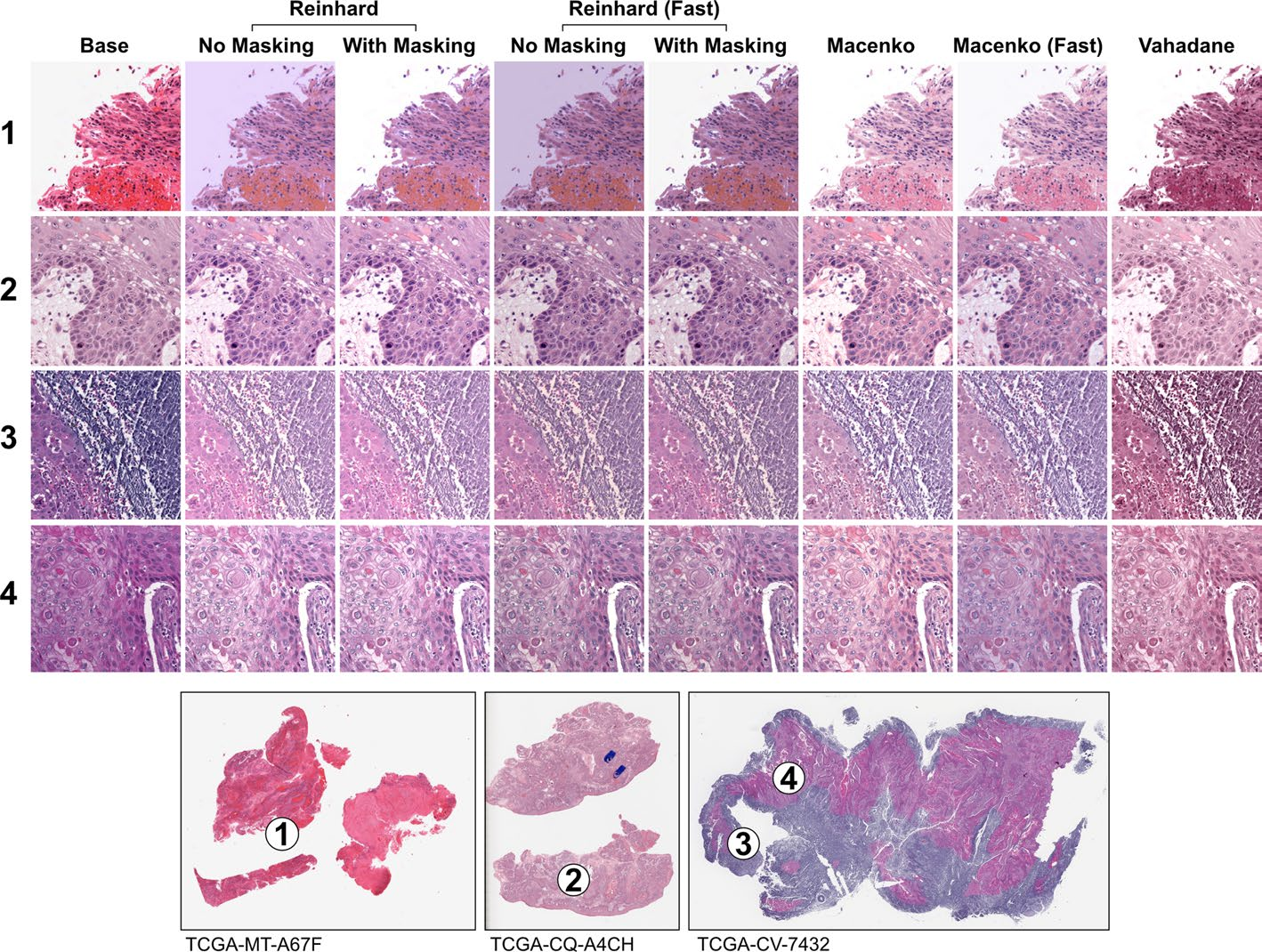
Reinhard方法的快速变体与标准Reinhard变体产生了类似的结果,但在感知亮度上略有差异。在大多数情况下,遮罩和不遮罩的Reinhard变体产生了相似的图像,但在包含高背景内容的图像瓦片中,不遮罩的变体会产生粉色调的背景。
与标准Macenko标准化相比,上下文感知的Macenko标准化通常产生了具有更高感知对比度的图像,但在明亮区域偶尔会洗掉细微细节,例如含有脂肪的区域(补充文件1:图8)。

Macenko染色增强产生了逼真、无伪像、具有多样染色色调的图像(补充文件1:图9)。

计算效率在Tensorflow和PyTorch之间进行了比较,结果见图9。

去除亮度标准化步骤后,使用Tensorflow的Reinhard标准化器的峰值染色标准化速度提高了272%,Macenko标准化器的速度提高了51%。使用PyTorch时,Reinhard和Macenko标准化器的速度分别提高了26%和24%。
当在GPU上处理时,Reinhard标准化器表现出优越的计算性能,而当在CPU上处理时,Macenko标准化器更快。Vahadane算法的实用性受到处理时间长的限制,无法用于实时标准化。
基于定性和计算效率的组合,选择了标准Macenko染色标准化策略进行后续分析。实时染色增强在模型训练期间使用。
3-2:弱监督瓦片基分类
在机构数据集的262张切片上,使用三折交叉验证和Tensorflow后端,训练了弱监督的二元分类模型以预测HPV状态。
首先,在2.5倍至40倍之间的七个放大倍数水平上,使用Xception架构和一组超参数对第一个交叉折叠进行了训练(图10)。

最佳性能出现在6.6X(瓦片微米宽度为453),因此这个放大倍数水平被用于后续分析。
使用贝叶斯超参数优化和“浅层”搜索空间配置,在第一个交叉折叠上调整超参数,最多进行50次迭代,并为每个超参数组合训练5个重复模型。每个模型的平均训练时间为2分钟11秒。使用最佳表现的超参数组合训练了10个模型,并与使用起始超参数训练的10个模型进行了比较。
AUROC没有通过优化超参数得到改善(0.808 vs. 0.803, P=0.11),因此初始超参数用于后续分析(补充文件1:表2)。

三个交叉折叠的AUROC分别为0.80、0.84和0.78,AP分别为0.79、0.83和0.77(图11)。

使用基于丢弃的不确定性量化方法确定置信度阈值。然后,使用之前确定的最佳超参数和Tensorflow后端在完整数据集上对模型进行了最终训练。
当在TCGA的459张切片组成的保留测试集上进行验证时,该模型产生了AUROC为0.87和AP为0.80的结果。使用不确定性量化和平滑度阈值处理,89.5%的切片具有高置信度预测,高置信度预测的AUROC为0.88。在交叉验证确定的预设预测和不确定性阈值下,最终模型在具有高置信度预测的测试集上的准确率为90.2%,灵敏度为77.0%,特异性为92.0%。
在最终模型在保留测试集上进行训练和评估后,进行了一次后验分析,以评估染色增强对模型性能的影响。在完整的芝加哥大学训练数据集上,使用和不使用染色增强分别训练了10个重复模型,然后在TCGA的外部测试集上进行评估。这种后验分析表明,染色增强在测试集上导致AUROC略微但统计学上显著提高,从0.871±0.008增加到0.882±0.014(P=0.021,单侧t检验)。
3-3:多实例学习
为了训练多实例学习(MIL)模型,首先从全切片图像中以6.6X的放大倍数(453微米宽度)提取图像瓦片,并使用Macenko标准化。病理学家注释的ROI未用于MIL实验。
然后使用预训练模型CTransPath[50]计算所有提取的图像瓦片的图像特征。MIL模型使用CLAM单分支架构[18]构建,并使用一组默认超参数进行训练(补充文件1:表3)。

模型总共训练了20个周期。使用树折交叉验证,MIL模型的AUROC分别为0.77、0.78和0.79。在芝加哥大学完整数据集上不进行验证,然后将最终模型测试在TCGA测试集上。在这个保留的测试集上,最终模型的AUROC为0.81,AP为0.77。
3-4:特征空间分析
使用包含的特征生成接口,为弱监督瓦片基模型生成了训练数据集和测试数据集的特征,并通过UMAP降维方法可视化了特征空间。
TCGA和芝加哥大学特征空间的UMAP图显示了训练和测试数据中HPV阴性与HPV阳性图像的良好类别分离(图12A和B)。

芝加哥大学特征空间的马赛克图突出了与HPV状态相关的已知生物相关图像特征(图12E):
- 区域1,富含HPV阳性图像,显示了紧密排列的细胞,细胞质较少,周围有炎症。
- 区域3,主要由HPV阴性图像组成,显示了大量的角化以及具有增加细胞质的异形细胞。这两个区域的发现与已知的组织病理学关联一致[78, 79]。
- 区域2,一个包含HPV阳性和HPV阴性图像的中间区域,显示了角化、炎症和细胞质含量不同的细胞。这一特征空间分析支持了模型预测的生物学合理性。
使用包含的特征生成接口生成了用于训练多实例学习模型的CTransPath特征,并进行了可视化。使用CTransPath特征在芝加哥大学训练数据集上分离HPV阴性和HPV阳性图像瓦片不如使用瓦片基模型的特征清晰(图12C)。
在TCGA的测试数据集上,使用这种可视化方法无法清晰地区分HPV阳性和HPV阴性图像瓦片(图12D)。特征空间中HPV阳性和HPV阴性图像之间的较差分离可能部分解释了瓦片基模型和MIL模型性能差异的原因。
3-5:可解释性
从最终瓦片基模型生成了预测热图,平均每张切片的生成时间为22.1±4.2秒。预测和不确定性的示例热图如图13所示。

一般来说,低不确定性区域和强烈预测HPV阴性状态的区域显示出高角化和具有异形核的细胞。低不确定性区域和强烈预测HPV阳性状态的区域通常显示出紧密排列的细胞,细胞核单调,周围有炎症细胞浸润。这两种观察结果与HPV状态已知的历史病理学关联一致[78, 79]。
使用vanilla梯度、三种综合梯度的变体和XRAI生成了一个正确预测HPV阴性的图像瓦片的一个示例的显著性图(图15A)。
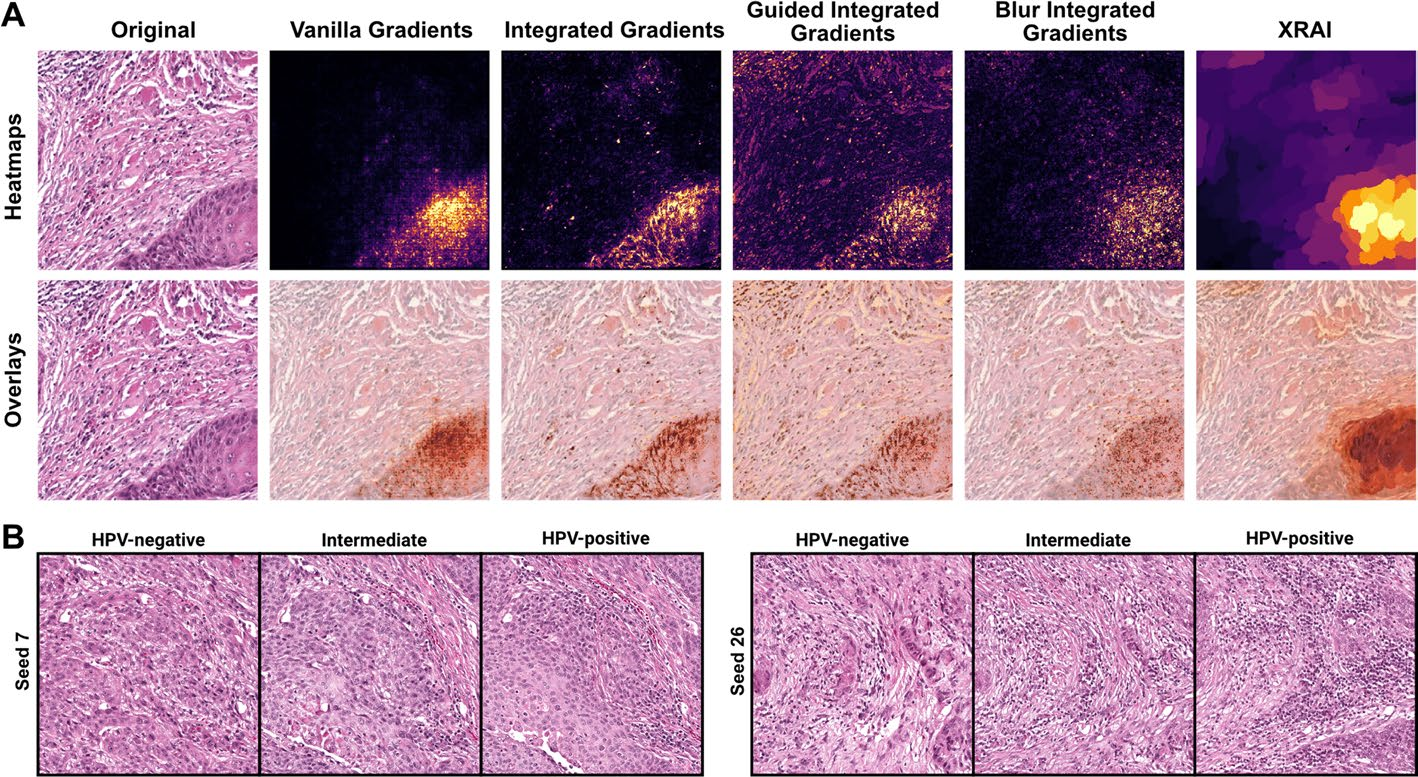
对于这个例子,所有显著性图都突出了图像瓦片中角化较重的部分,这是一个已知与HPV阴性相关的组织病理学因素。最后,在机构数据集上训练了一个条件生成对抗网络(cGAN),根据HPV状态进行条件化,通过合成组织病理学为训练好的分类器提供生成解释[60]。
使用了StyleGAN2架构,使用原始实现中的默认超参数[58]。训练在四个A100 GPU上进行了38小时,并在生成1500万张图像后停止,因为进一步训练出现了发散。通过这种方法生成的可视化突出了角化和核异形性的差异,这在合成HPV阴性图像中增加,而炎症细胞浸润在合成HPV阳性图像中增加(图15B)。这些差异与HPV状态已知的历史病理学关联一致,进一步支持了学习的图像特征的生物学合理性[78, 79]。
3-6:硬件部署
上述实验和基准测试是在一个基于Linux的工作站上进行的,该工作站配备了AMD Treadripper 3960X CPU和NVIDIA A100 40 GB GPU。
该硬件上训练和评估分类模型所需的总时间在补充文件1:图11中提供了详细信息。
为了评估将全切片图形界面作为WSI分析的可部署工具的可行性,Slidefow Studio被部署并测试在各种Linux工作站、Windows 10桌面(配有专用GPU)、Intel MacBook Pro、M2 MacBook Pro、Raspberry Pi 4(4 GB)和Jetson Orin Nano上。
所有设备都以可用的性能运行Slidefow Studio,并成功地为WSI生成了预测。对于模型训练,GPU加速仅在具有专用GPU的系统上可用。
四、结论
Slidefow是一个灵活的、端到端的深度学习工具包,用于数字病理学。
它具备计算高效的整张切片图像处理、与Tensorflow和PyTorch兼容的数据存储、高效的GPU加速染色标准化以及一个图形用户界面(GUI),用于支持训练好的深度学习模型的部署。
Slidefow包含了多种数字病理学的深度学习方法,支持广泛的研究目标,无需切换软件环境或重新处理数据。该软件文档齐全,旨在支持具有不同编程经验的研究人员在数字组织病理学中开发新的深度学习应用程序。



















