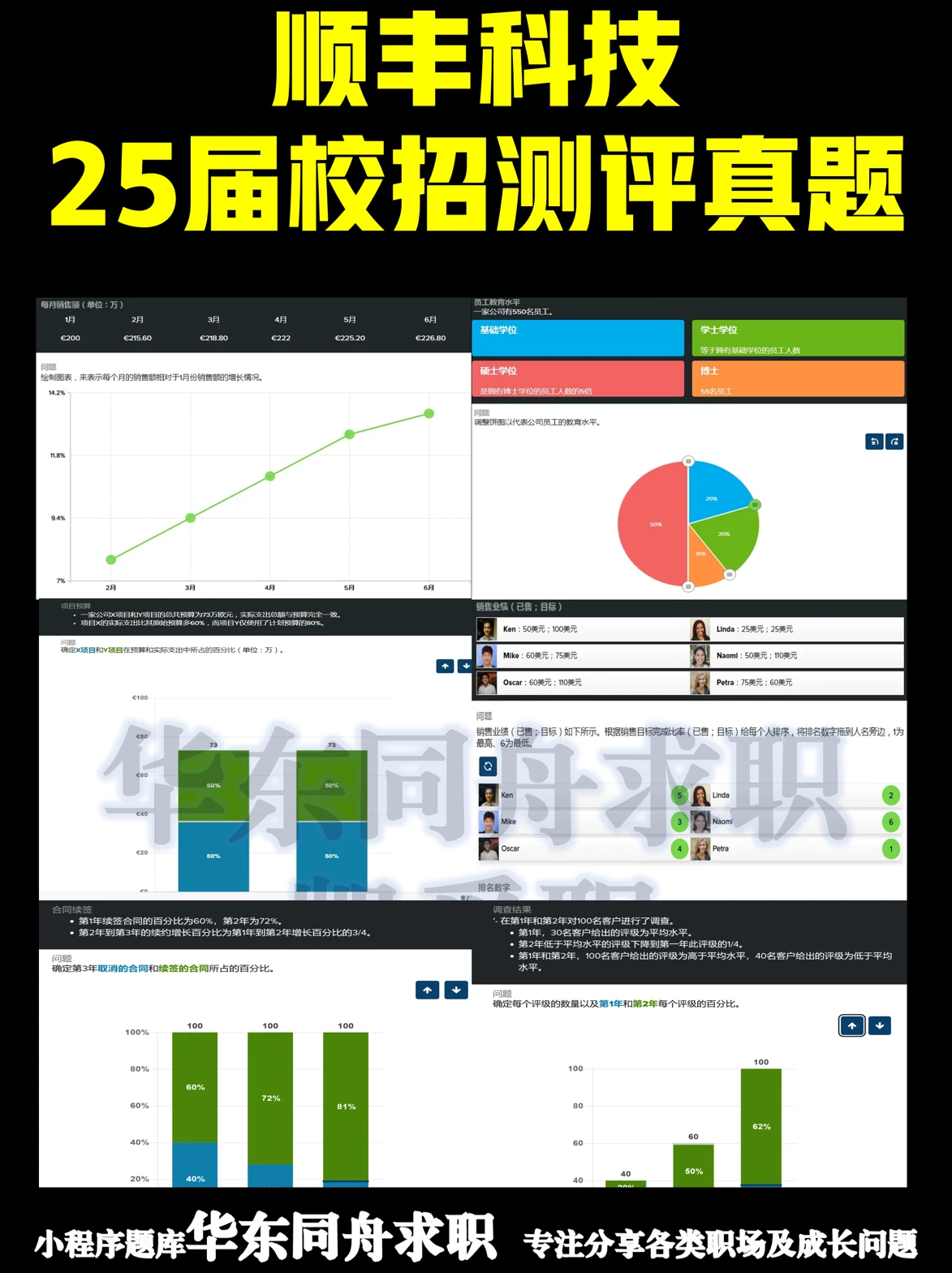
PCA(主成分分析)的优缺点:
优点:降低数据的复杂性,识别最重要的多个特征;
缺点:不一定需要,且可能损失有用信息;
适用数据类型:数值型数据。
移动坐标轴
如下图,如果要求我们画出一条直线,这条线要尽可能覆盖这些点。

在PCA中,我们对数据的坐标进行旋转,该旋转的过程取决于数据的本身。第一条坐标轴旋转到覆盖数据的最大方差位置。数据的最大方差给出了数据的最重要信息。
在选择了覆盖数据最大差异化的坐标轴之后,我们选择第二条坐标轴。假如该坐标轴与第一条坐标轴垂直,它就是覆盖数据次大差异性的坐标轴。这里更严谨的说法是正交。利用PCA,我们将数据坐标轴旋转至数据角度上的那些最重要方向。

我们已经实现了坐标轴的旋转,接下来是降维。坐标轴的旋转并没有减少数据的维度。考虑上图的数据,其中包含3个不同的类别。要区分这3个类别,可以使用决策树。决策树是基于一个特征来做决策的。我们会发现,在x轴上可以找到一些值,这些值能够很好的将这3个类别分开。这样,我们就可能得到一些规则,比如当(x<4)时,数据属于类别0.如果使用SVM这样稍微复杂一些的分类器,我们就会得到更好地分类面和分类规则,比如当(w0*x+w1*y+b)>0时,数据也属于类别0.SVM可能比决策树得到更好的分类间隔,但是分类超平面时就很难解释。
通过PCA进行降维处理,我们就可以同时获得SVM和决策树的优点:一方面,得到了和决策树一样简单的分类器,同时分类间隔和SVM一样好。考虑到上图中的下半部分,其中的数据来自于上半部分并经PCA转换之后绘制而成的。如果仅使用原始数据,那么这里的间隔会比决策树的间隔更大。另外,由于只需要考虑一维信息,因此数据就可以通过比SVM简单得多的、很容易采用的规则进行区分。
还是上图的数据中,我们只需要一维信息即可,因为另一维信息只是对分类缺乏贡献的噪声数据。在二维平面下,这一点看上去微不足道,但是如果在高维空间下则意义重大。
通过数据集的协方差矩阵及其特征值的分析,我们可以求得这些主成分的值。一旦得到了协方差矩阵的特征向量,我们就可以保存最大的N个值。这些特征向量也给出了N个最重要特征的真实结构。我们可以通过将数据乘上这N个特征向量而将它转换到新的空间。
在NumPy中实现PCA
将数据转换成前N个主成分的伪代码大致如下:
去除平均值
计算协方差矩阵
计算协方差矩阵的特征值和特征向量
将特征值从大到小排序
保留最上面的N个特征向量
将数据转换到上述N个特征向量构建的新空间中
实际代码实现:
from numpy import *
def loadDataSet(fileName,delim='\t'):
fr=open(fileName)
stringArr=[line.strip().split(delim) for line in fr.readlines()]
datArr=[list(map(float,line)) for line in stringArr]
return mat(datArr)
def pca(dataMat,topNfeat=9999999):
meanVals=mean(dataMat,axis=0)
#去除平均值
meanRemoves=dataMat-meanVals
covMat=cov(meanRemoves,rowvar=0)
eigVals,eigVects=linalg.eig(mat(covMat))
eigValInd=argsort(eigVals)
#从小到大对N个值进行排序
eigValInd=eigValInd[:-(topNfeat+1):-1]
redEigVects=eigVects[:,eigValInd]
#将数据转换到新空间
lowDDateMat=meanRemoves*redEigVects
reconMat=(lowDDateMat*redEigVects.T)+meanVals
return lowDDateMat,reconMat上述代码包含了通常的NumPy导入和loadDataSet()函数。这里的loadDataSet使用了两个list comprehension来构建矩阵。
pac()函数有两个参数:第一个参数适用于进行PCA操作的数据集,第二个参数topNfeat则是一个可选参数,即应用的N个特征。如果不指定topNfeat的值,那么函数就会返回前9999999个特征,或者原始数据中全部非特征。
pca()函数首先计算并减去原始数据集的平均值。然后,计算协防差矩阵及其特征值,接着利用argsort()函数对特征值进行从小到大的排序。根据特征值排序结果的逆序就可以得到topNfeat个最大的特征向量。这些特征向量将构成后面对数据进行转换的矩阵,该矩阵则利用N个特征将原始数据转换到新空间中。最后,原始数据被重构后返回用于调试,同时降维之后的数据集也被返回了。
我们在testSet.txt文件中加入一个由1000个数据点组成的数据集,在这个数据集上进行PCA操作查看运行效果:
dataMat=loadDataSet('test/testSet.txt')
lowDMat,reconMat=pca(dataMat,1)
print(shape(lowDMat))![]()
可以看到,降维之后的矩阵是一个一维矩阵。
我们将降维后的数据和原始数据一起绘制出来:
import matplotlib.pyplot as plt
fig=plt.figure()
ax=fig.add_subplot(111)
ax.scatter(dataMat[:,0].flatten().A[0],dataMat[:,1].flatten().A[0],marker='^',s=90)
ax.scatter(reconMat[:,0].flatten().A[0],reconMat[:,1].flatten().A[0],marker='o',s=50,c='red')
plt.show()
调整参数,观察不同的结果:
dataMat=loadDataSet('test/testSet.txt')
lowDMat,reconMat=pca(dataMat,2)
import matplotlib
import matplotlib.pyplot as plt
fig=plt.figure()
ax=fig.add_subplot(111)
ax.scatter(dataMat[:,0].flatten().A[0],dataMat[:,1].flatten().A[0],marker='^',s=90)
ax.scatter(reconMat[:,0].flatten().A[0],reconMat[:,1].flatten().A[0],marker='o',s=50,c='red')
plt.show()
可以看到,设置两个特征值的话,数据就会和原始数据重合。






![【动态规划、dp】P1091 [NOIP2004 提高组] 合唱队形 题解](https://img-blog.csdnimg.cn/img_convert/e6c2c6a9da18ff3b2254251f173ed7a3.jpeg#pic_center)