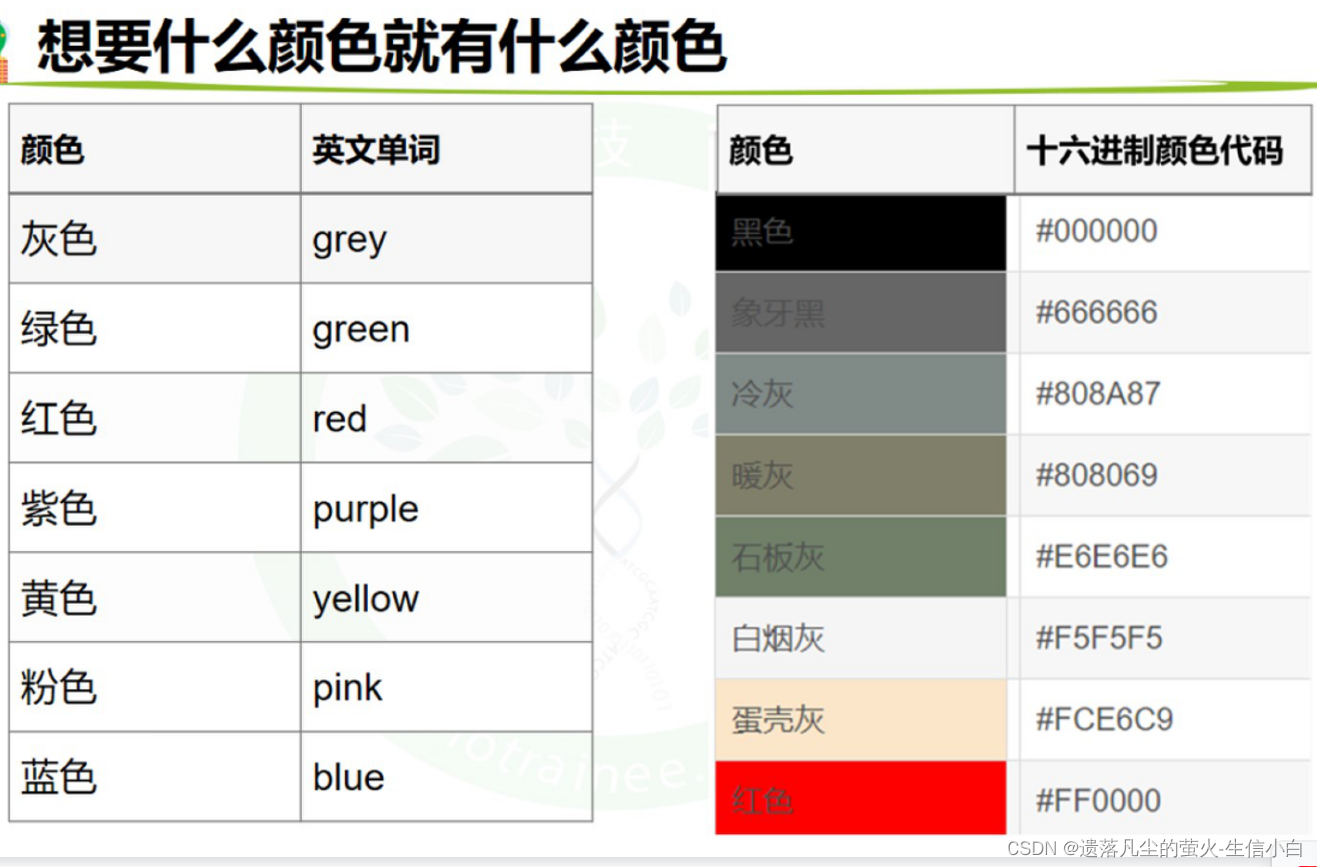
From生物技能树(R第五节)
文章目录
- 一、文件读写
- 1.注意用project管理工作目录
- 2、文件读取
- 1、读取.txt文件
- 2、读取.csv文件
- 注意:数据框不允许重复的行名
- 3.数据框的导出
- 4.读取文件的其他方式(用于读取/导出文件的R包)--经验
- 1.base
- 2.readr
- 3.data.table
- 4.rio:读取xlsx比较推荐!
- 5、练习
- 6.总结
- 二、掉包
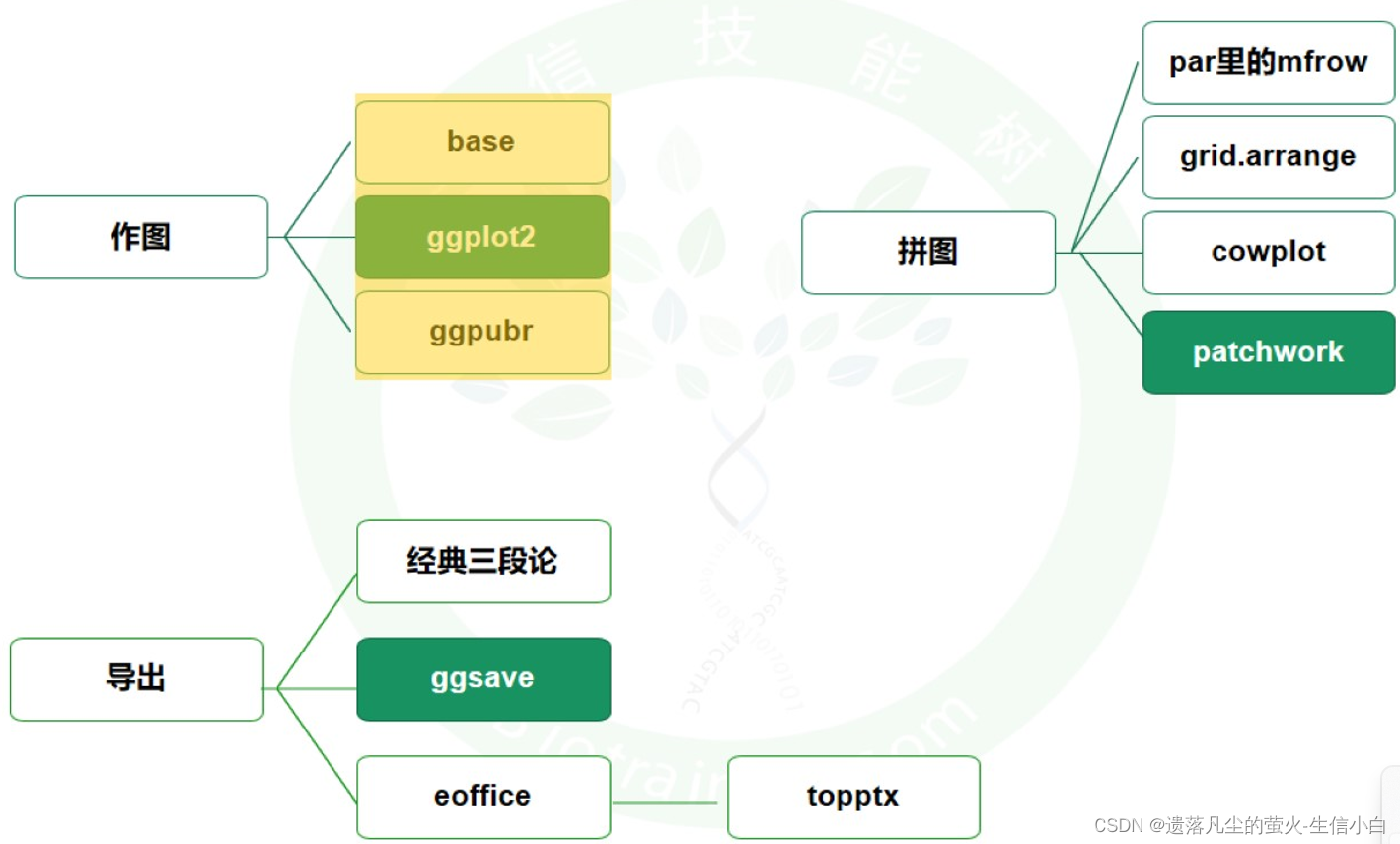
- 三、R语言作图:分三类
- 1.常见R包和函数
- 2._plots.R
- 1.基础包-绘图函数
- 2.ggplot2与ggpubr
- 3.-ggplot2.R
- ggplot2语法:
- 1.ggplot2:绘图、颜色、形状
- 2.入门级绘图模板:作图数据,横纵坐标
- 3.属性设置(颜色、大小、透明度、点的形状,线型等)
- 1.手动设置
- 2.映射:按照数据框的某一列来定义图的某个属性
- 3.映射 vs 手动设置
- 4.能不能自行指定映射的具体颜色?
- 5.区分color和fill两个属性
- 1.空心形状和实心形状都用color设置颜色
- 2.既有边框又有内心的,才需要color和fill两个参数
- 4.拼图
- 5.导出
- 四、练习1(5_exercise.R)
- 练习2
一、文件读写
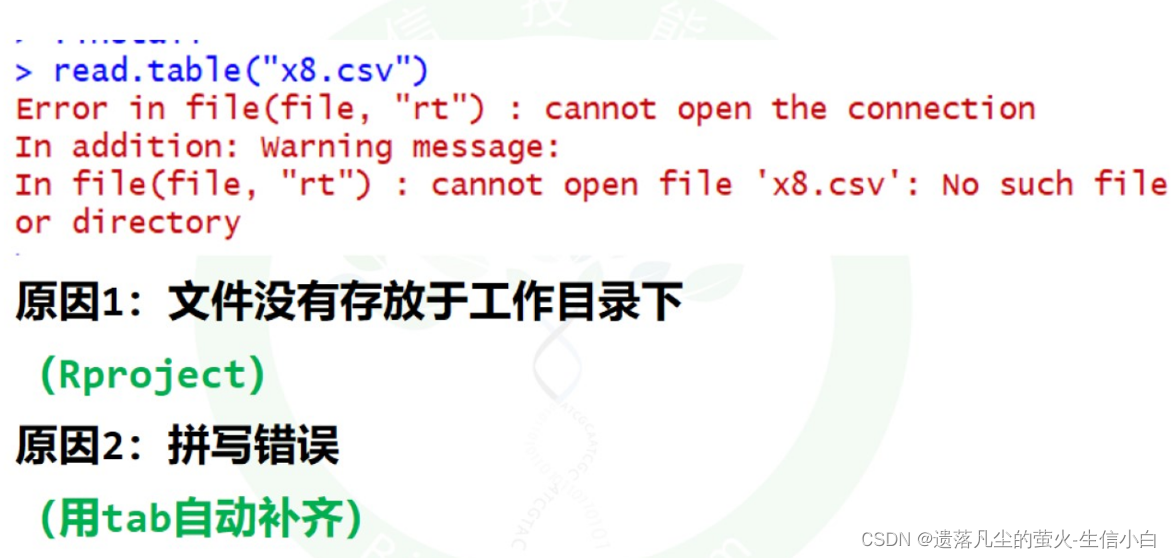
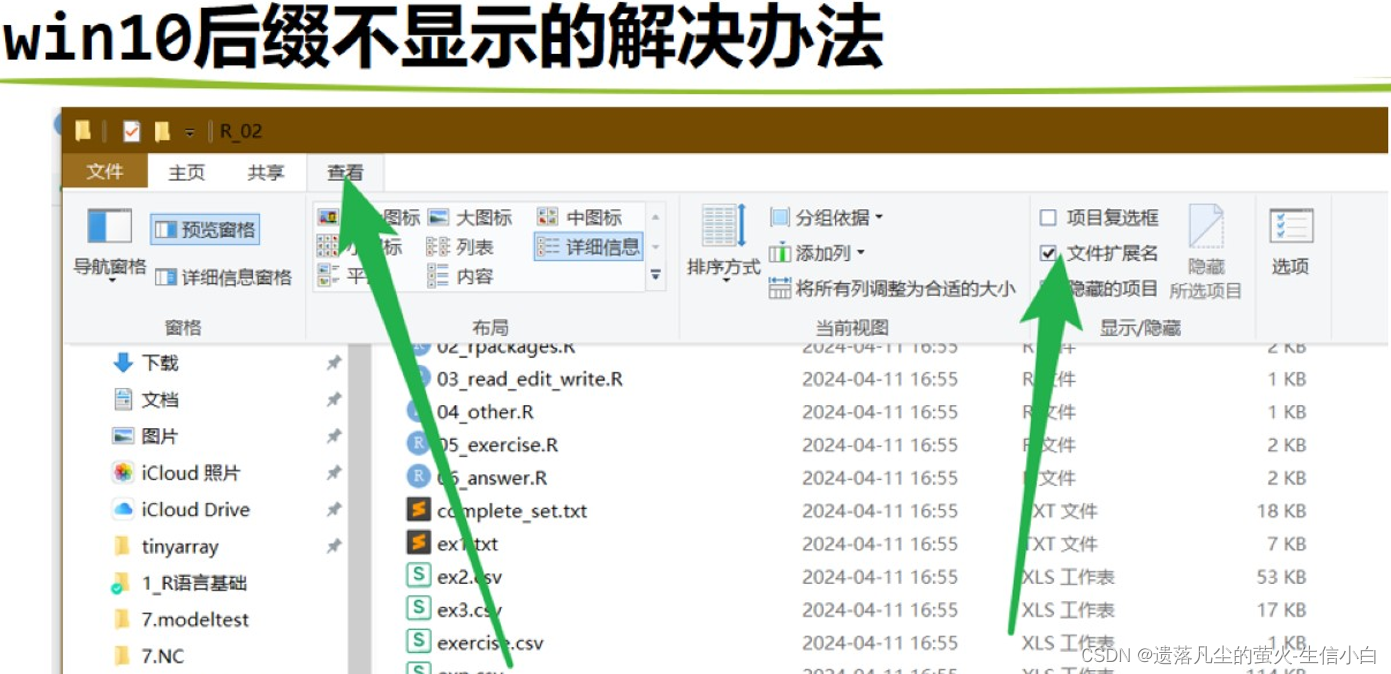
1.注意用project管理工作目录
不然就会报错
2、文件读取
#read.csv() 函数是用来读取CSV(逗号分隔值)文件并将其转换为数据框(data frame)的函数。CSV文件是一种常见的数据存储格式,广泛应用于数据交换和数据分析。
例如,如果你有一个名为 data.csv 的文件,它位于当前工作目录中,并且第一行包含列名,你可以使用以下命令读取这个文件:
my_data <- read.csv("data.csv")
这将创建一个名为 my_data 的数据框,其中包含了CSV文件中的数据。
如果CSV文件使用分号作为字段分隔符,你可以指定 sep 参数:
my_data <- read.csv("data.csv", sep = ";")
#通常用于读取txt格式,read.table() 是R语言中的一个基础函数,用于读取文本文件并将其转换为数据框(data frame)。这个函数非常灵活,可以处理多种不同的数据格式,不仅仅是表格数据。它通常用于读取那些没有内置读取函数的文本文件格式。
read.table()
#read.delim()也可以读取txt文件,不常用但是读txt文件时比read.table不容易出错
#两者都属于read.系列函数
?read.table()两者的帮助文档都在一起,都属于read系列函数,参数通用,默认值不同函数有所不同,如header列名参数,seq分隔符参数等。

如果想知道文件读入后是什么数据结构,应该class(?)
A.test
B.“ex3.csv”
答案:应该选A,因为"ex3.csv"是个文件名,文件名加双引号,不是变量
文件和变量的区别,从工作目录下可以找到"ex3.csv"这个文件名,而关掉R找不到test,test是r语言里的数据
1、读取.txt文件
#查看,发现问题,读取的时候第一行变成了列名,列名不属于表格的正式内容,
ex1 <- read.table("ex1.txt")
#因为数据框的一列属于向量,而一列只能有一种类型的向量,字符的包容性更大,这样的话剩下的数值型都会被强制转换为字符型,因为对于数据框来说一列只能有一种数据类型
> ex1[2,4]
[1] "45351"
#遇到报错,查看帮助文档,寻找解决方案,把列名归位,read.table中header的默认值为F,意思是文件内不包括列名,如果包括的话要改为T
x1 <- read.table("ex1.txt",header = TRUE)
2、读取.csv文件
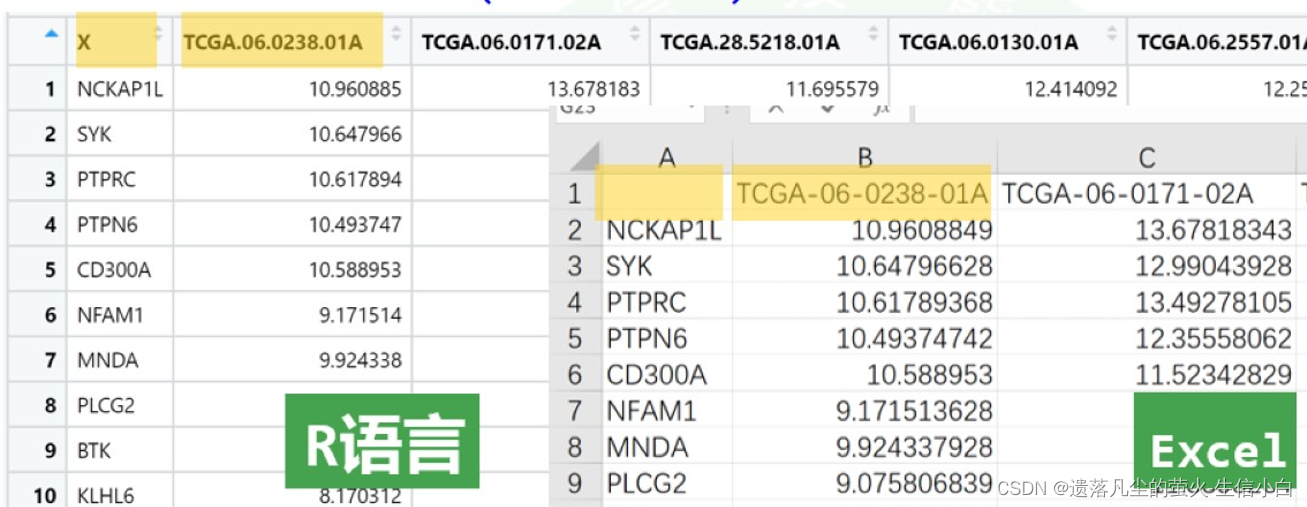
#文件查看,和excel查看相比,因为R语言列名不允许出现特殊字符,列名中的-变成了.,因为列名中有特殊字符,被强制转换了,行名变成了第一列,没有变成行名,上面多了一个X,查看帮助文档,
> #2.读取ex2.csv
> ex2 <- read.csv("ex2.csv")
> View(ex2)
> ex2 <- read.csv("ex2.csv",row.names = 1,check.names = F)
>
> row.names = 1的作用是把第一列设置成行名
> check.names = F意思是不检查列名中的特殊字符
注意:数据框不允许重复的行名
如有重复的话会报错,解决方法:需要先不按行名读进来,去重复,然后再row.names=rod$A
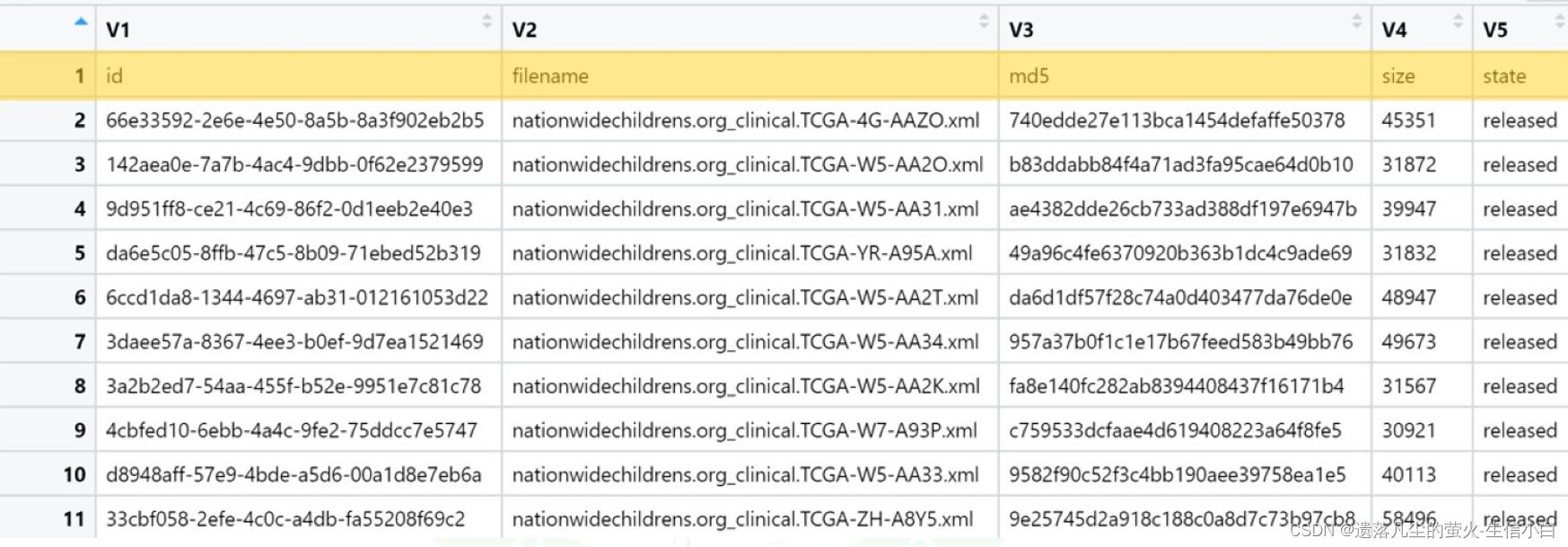
rod = read.csv("rod.csv")

3.数据框的导出
将数据框导出为一个表格文件
write.csv(ex2,file = "example.csv") #用的多,记得写后缀.csv
write.table(ex2,file = "example.txt") #用的少,记得写后缀.txt

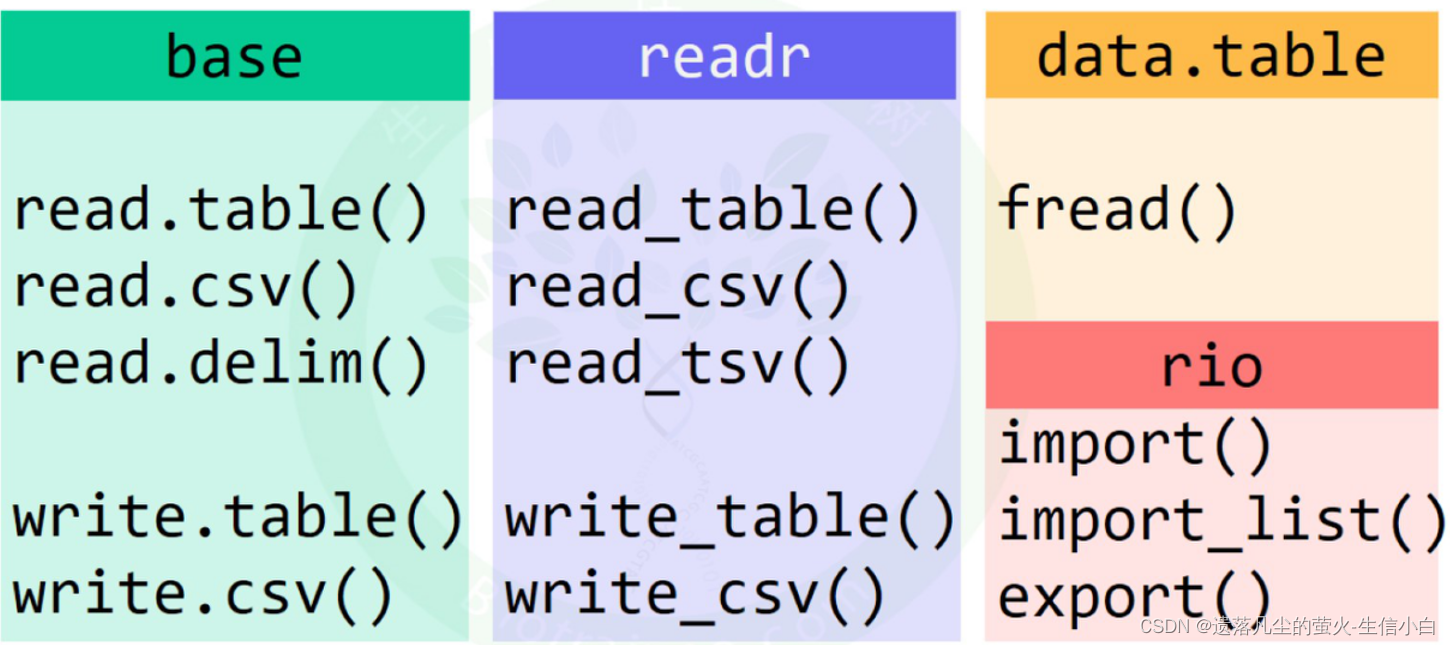
4.读取文件的其他方式(用于读取/导出文件的R包)–经验
用于读写文件的r包有很多,read.table、read.csv等都属于base,不用加载任何R包即可使用,有一些其他的R包也可以用于读写文件,参数更智能
1.base
2.readr
大神写的,打开会比base快,但不推荐用
3.data.table
> library(data.table)
> ex1 = fread("ex1.txt")
> View(ex1)
> class(ex1)
[1] "data.table" "data.frame"
#不用设置header=T
多了一个属性叫data.table,是一种特殊的数据框,不过不常用,因此可去除data.table这种属性
> ex1 = fread("ex1.txt",data.table = F)
> class(ex1)
[1] "data.frame"
#加上后面的参数之后data.table属性就消失了
ex2 = fread("ex2.csv",data.table = F)
#缺点:fread没有设置行名的参数,因为大神觉得行名是个不应该存在的东西…………
library(tibble)
ex2 = column_to_rownames(ex2,"V1")
#又想用fread,又想设置行名,于是只能用另一个函数来设置行名,在tibble的R包里,函数column_to_rownames()可以设置行名,即第一列设置为行名
4.rio:读取xlsx比较推荐!
#一个函数支持读取很多格式,见帮助文档,#如果没有特殊情况,excel文件推荐用import()来读取
library(rio)
ex1 = import("ex1.txt")
#如果excel文件含有多个工作博,会用到函数import_list,这种场景用的较少,用时现学即可
#一个函数支持导出很多格式,见帮助文档
export(ex1,file = "ex1.xlsx")
5、练习
练习5-1:
1.读取GSE217012_Normalized_RPKM_LOG2_matrix.txt.gz(已保存在工作目录)
注 :gz是压缩格式,可以不用解压,直接读取
#加上check.name = F,是因为有特殊字符,经常加着
x1 = read.delim("GSE217012_Normalized_RPKM_LOG2_matrix.txt.gz",check.name = F)
x1 = data.table::fread("GSE217012_Normalized_RPKM_LOG2_matrix.txt.gz",data.table = F)
x3 = rio::import("GSE217012_Normalized_RPKM_LOG2_matrix.txt.gz")
2.加载y.Rdata(已保存在工作目录),求gene1列的平均值
如果你看到 y.Rdata,这通常意味着这是一个包含了至少一个对象的文件,该对象的名称是 y。这个文件可以通过R语言中的 load() 函数来读取。以下是如何加载 .RData 文件的基本命令:
> load("y.Rdata")
> mean(y$gene1)
Error in h(simpleError(msg, call)) :
error in evaluating the argument 'x' in selecting a method for function 'mean': $ operator is invalid for atomic vectors
# $不让用,是因为它不是一个数据框
> class(y)
[1] "matrix" "array"
> mean(y[,1])
> #是个矩阵
[1] NA
Warning message:
In mean.default(y[, 1]) : argument is not numeric or logical: returning NA
> y[,1]
GSM1 GSM2 GSM3 GSM4 GSM5 GSM6
"40" "20" "51" "46" "38" "49"
#取出的是向量,可转换格式
> mean(as.numeric(y[,1]))
[1] 40.66667
> #why?
> y[,1] = as.numeric(y[,1])
> y[,1]
GSM1 GSM2 GSM3 GSM4 GSM5 GSM6
"40" "20" "51" "46" "38" "49"
#没改成功是因为y是一个矩阵,没办法单独改一列的内容,一个矩阵里有两种数据类型的时候还是会把他兼容回去,
注意:转换数据类型的前提条件是数据框里只能有一种数据类型,需要重视数据类型,数据结构
load("y.Rdata")
mean(as.numeric(y[,"gene1"]))
## [1] 40.66667
一定要经常检查数据
6.总结

header 管行名,check.names管列名,row.name管要不要检查列名
在R语言中,header 是一个参数,经常出现在读取数据文件的函数中,如 read.csv() 或 read.table()。这个参数决定了函数在读取文件时是否应该将文件的第一行作为列名。
以下是一些使用 header 参数的常见场景:
read.csv():读取CSV文件时,如果你设置 header = TRUE,R会将文件的第一行作为列名。默认情况下,read.csv() 函数的 header 参数就是 TRUE。
my_data <- read.csv("data.csv", header = TRUE)
如果CSV文件的第一行不是列名,你应该将 header 设置为 FALSE。
read.table():在读取文本文件时,read.table() 函数的 header 参数默认为 FALSE。如果你想让R将第一行作为列名,你需要将其设置为 TRUE。
my_data <- read.table("data.txt", header = TRUE)
read.delim() 和 read.delim2():这些函数是 read.table() 的变体,它们默认将第一行视为列名,即 header = TRUE
在R语言中,check.names 是 read.table()、read.csv() 和其他相关函数的一个参数,用于控制是否检查并自动修改数据框中的列名以确保它们是合法的R变量名。
默认情况下,read.table() 和 read.csv() 函数会将列名作为字符型读取,而不会检查它们是否是合法的R变量名。如果你希望函数自动将列名修改为合法的变量名,可以设置 check.names = TRUE。
在R语言中,rownames() 函数用于获取或设置数据框(data frame)、矩阵(matrix)或其他列表(list)对象的行名(row names)。
二、掉包
仿照:语雀上”小洁怎么会分身“
添加链接描述
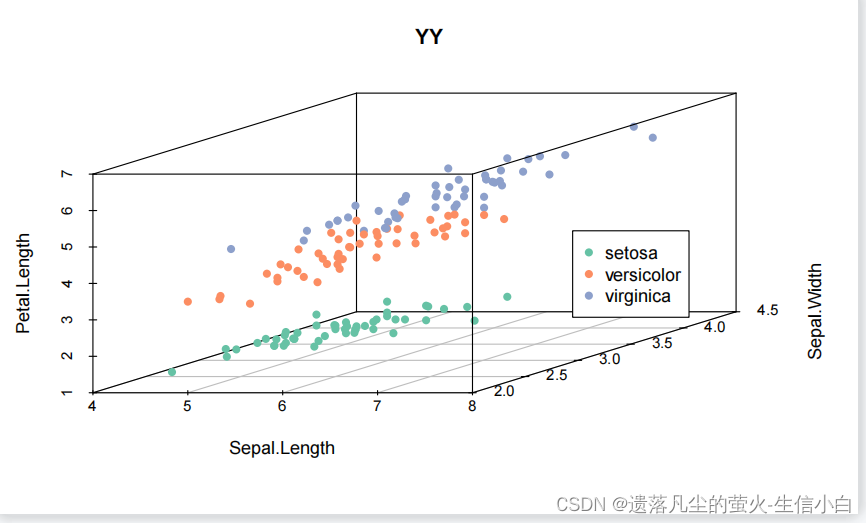
#install.packages("scatterplot3d")
library(scatterplot3d)
my_color = c("#66C2A5FF", "#FC8D62FF", "#8DA0CBFF")
colors = my_color[as.numeric(iris$Species)]
#因子转换为数值是可以转换的
p1 = scatterplot3d(iris[,1:3],color = colors,main="iris",pch = 16)
legend(p1$xyz.convert(8.5, 2.5, 5), legend = levels(iris$Species),
col = my_color, pch = 16)
#如果套用自己的数据
只需要更改iris$Species,代表分类的那一列,以及iris[,1:3]带入自己的数据
#install.packages("scatterplot3d")
library(scatterplot3d)
my_color = c("#66C2A5FF", "#FC8D62FF", "#8DA0CBFF","#fe4632")
dat = data.frame(a = rnorm(100),
b = rnorm(100),
c = rnorm(100),
d = as.factor(rep(c("m","n","o","p"),each = 25)))
colors = my_color[as.numeric(dat$d)]
p1 = scatterplot3d(dat[,1:3],color = colors,main="zyy",pch = 16)
legend(p1$xyz.convert(5, 2, 2), legend = levels(iris$Species),
col = my_color, pch = 16)
三、R语言作图:分三类
展示自己的数据
1.常见R包和函数
2._plots.R
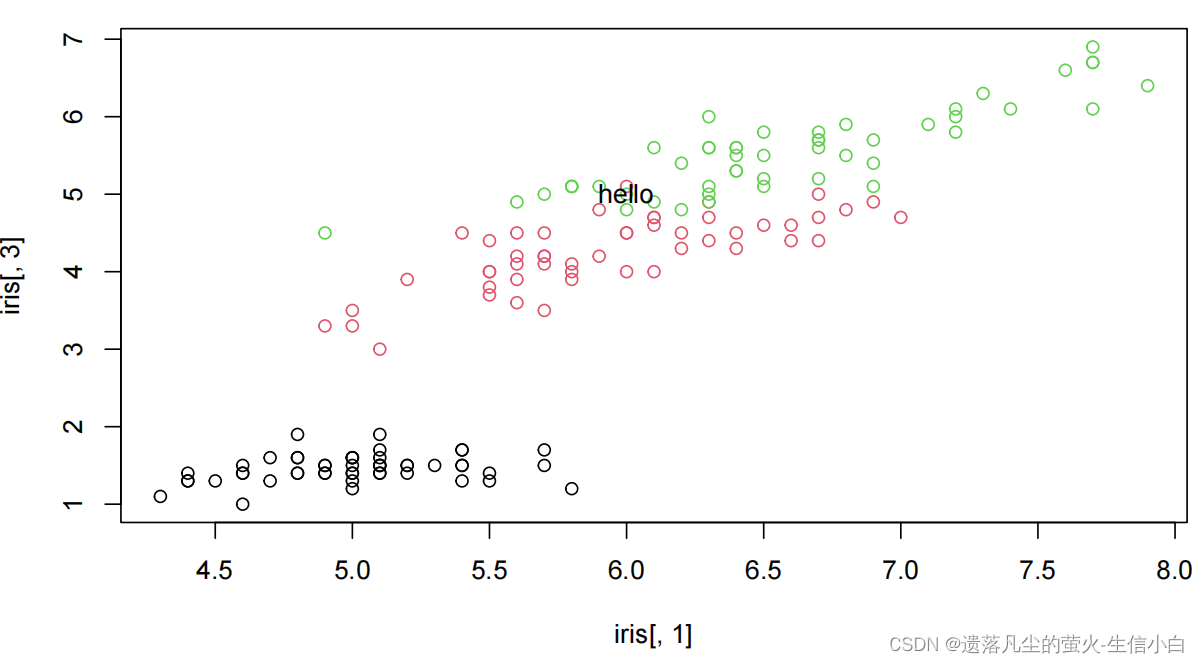
1.基础包-绘图函数
高级:画一整张图,低级:修改图中某个元素
plot(iris[,1],iris[,3],col = iris[,5])
text(6.5,4, labels = 'hello')
2.ggplot2与ggpubr
ggplot2 中坚力量,语法有个性,更加强大
library(ggplot2)
## Warning: 程辑包'ggplot2'是用R版本4.3.2 来建造的
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species))
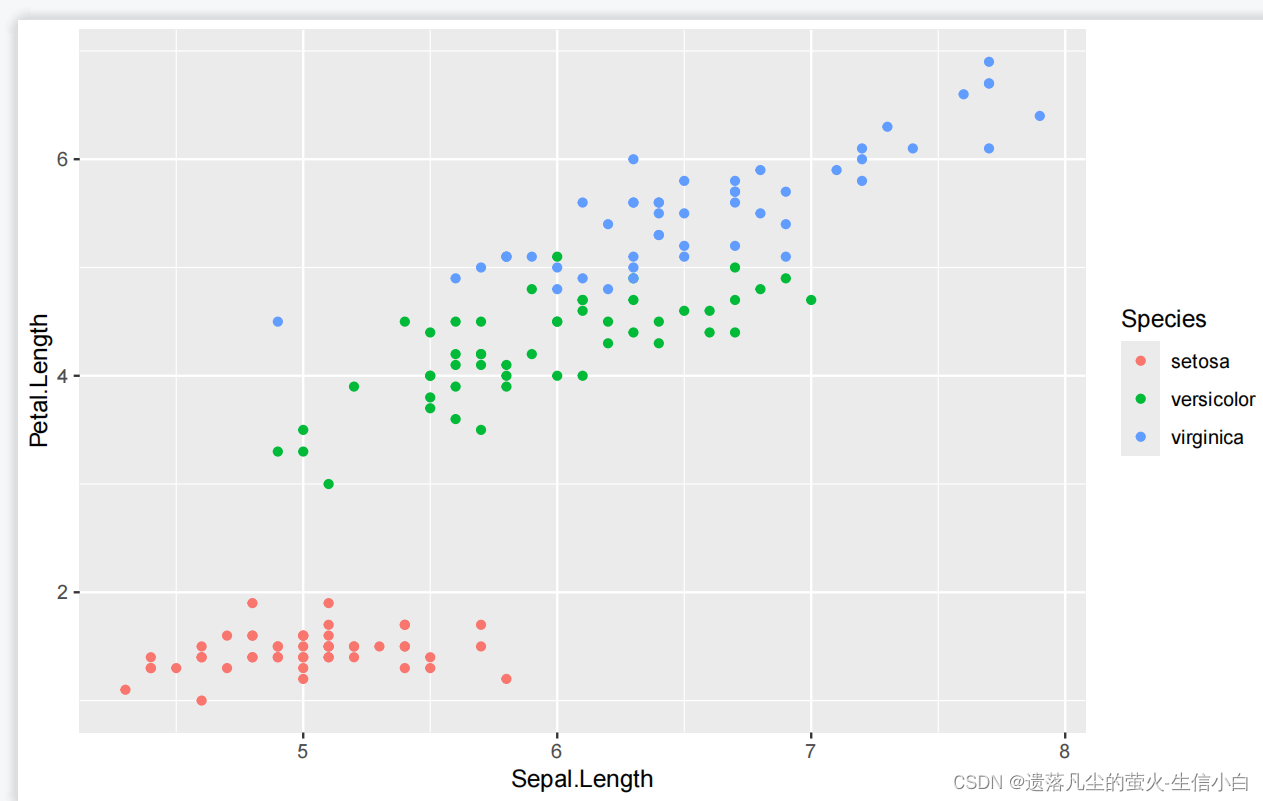
ggpubr 新手友好型,在ggplot2的基础上进行了简化和美化,没有灵魂,可扩展性没有那么强
library(ggpubr)
## Warning: 程辑包'ggpubr'是用R版本4.3.2 来建造的
ggscatter(iris,
x="Sepal.Length",
y="Petal.Length",
color="Species")
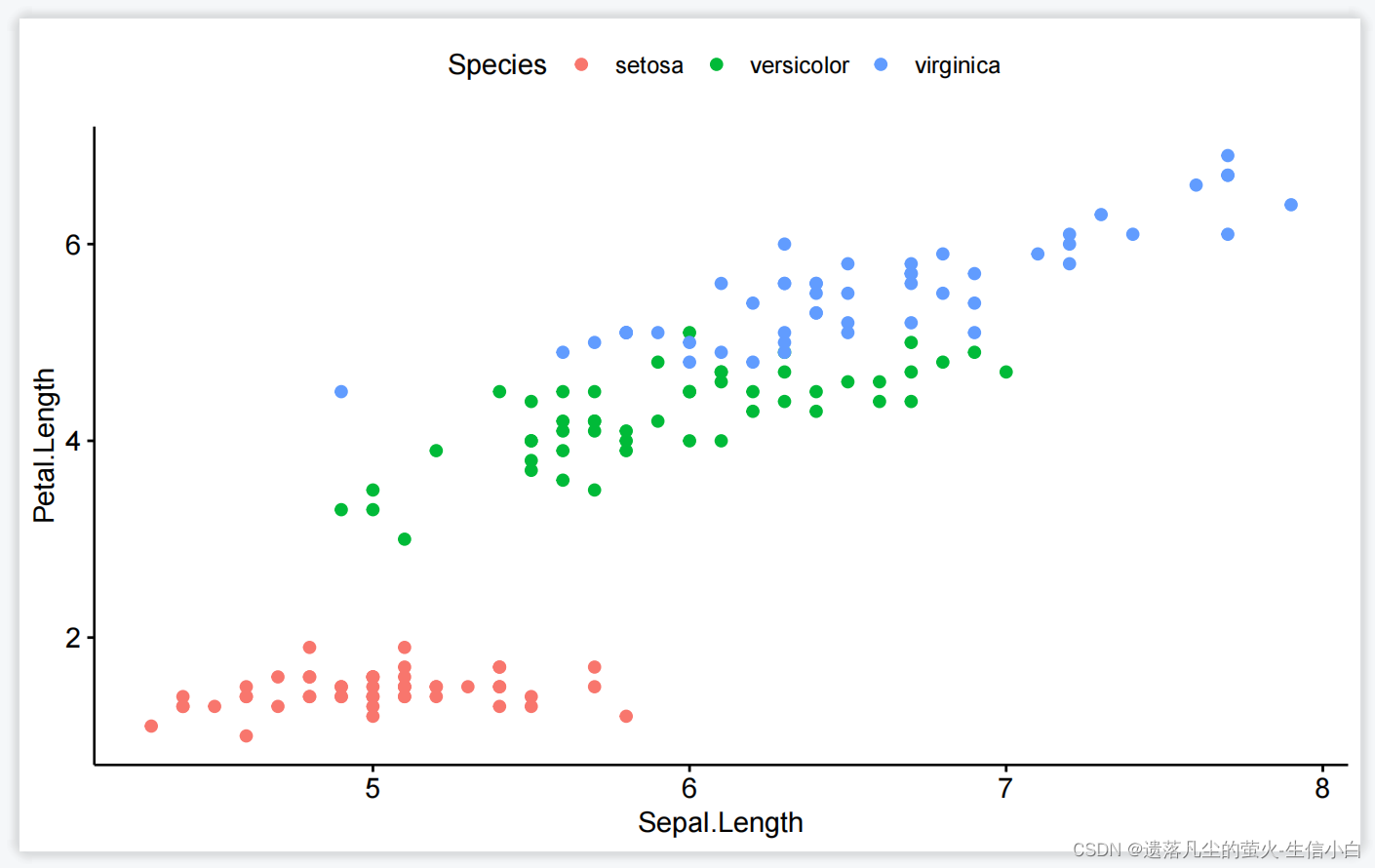
3.-ggplot2.R
ggplot2语法:

1.ggplot2:绘图、颜色、形状
library(ggplot2)
2.入门级绘图模板:作图数据,横纵坐标
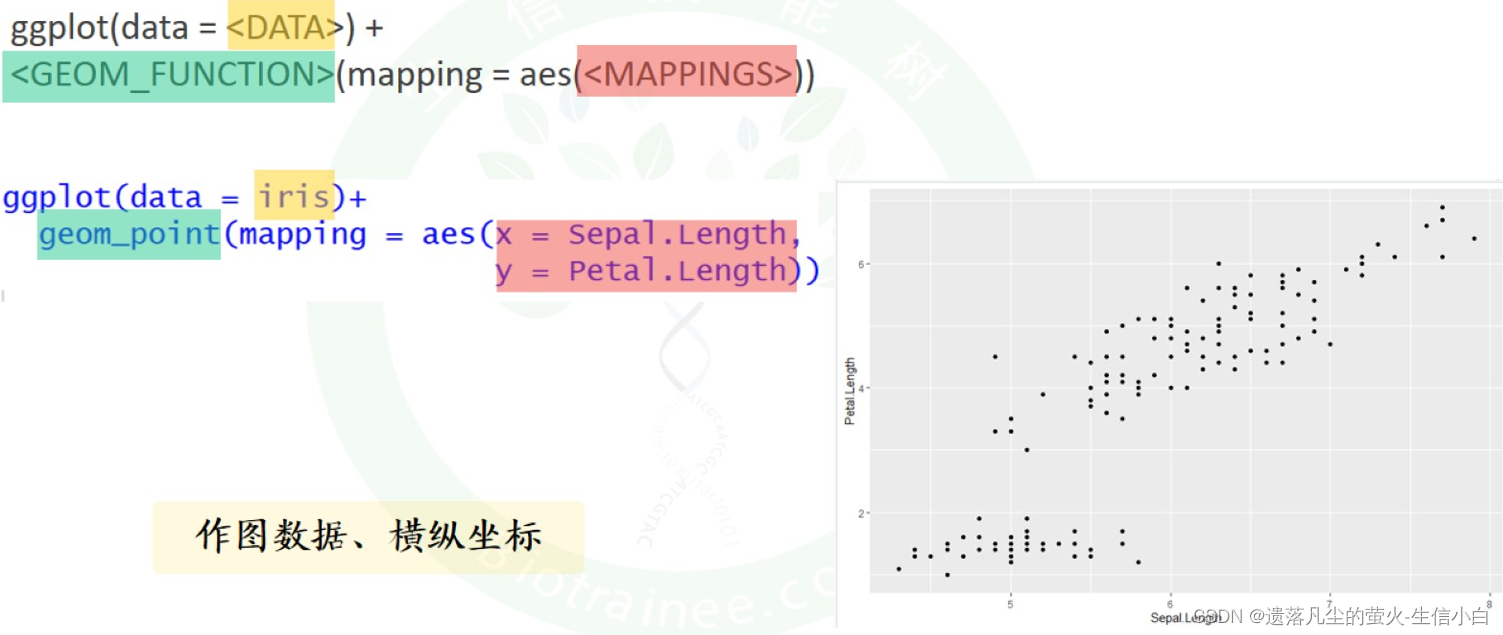
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length))
数据是iris,geom_point是绘图函数,两个列名分别传给了x和y作为横纵坐标,
ggplot独有的语法特点:列名没有引号,+号用于连接两个函数,行末写加号,说明两个代码在画同一张图,+会使缩进两个字目
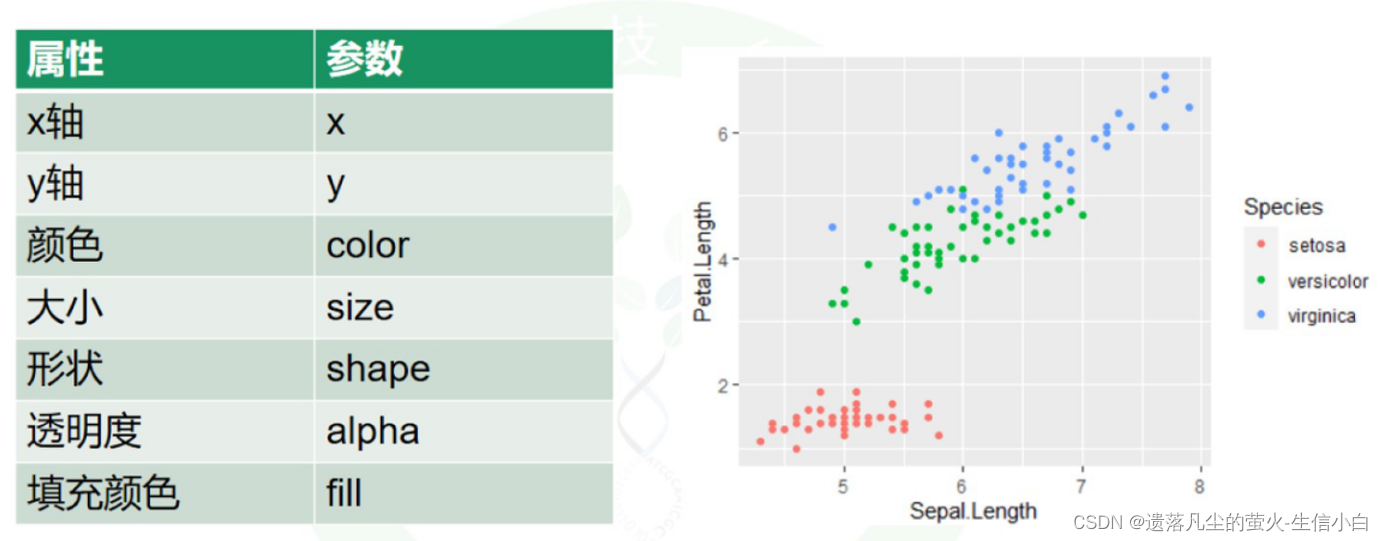
3.属性设置(颜色、大小、透明度、点的形状,线型等)
1.手动设置
手动设置,需要设置为有意义的值
和数据没有挂钩,只是手动设置,注意这部分不属于映射,因此在aes外面
ggplot(data = iris) +
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length),
color = "blue")
ggplot(data = iris) +
geom_point(mapping = aes(x = Sepal.Length, y = Petal.Length),
size = 5, # 点的大小5mm
alpha = 0.5, # 透明度 50%
shape = 8) # 点的形状,24个数字代表24个形状
2.映射:按照数据框的某一列来定义图的某个属性
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species))
根据Species分配颜色,注意要放到aes的括号里面去,表示映射,R包可以默认颜色
3.映射 vs 手动设置

映射:根据数据的某一列的内容分配为颜色
手动设置:把图形设置为一个或n个颜色,与数据内容无关
4.能不能自行指定映射的具体颜色?
color=颜色依据+(加号不要漏掉)scale_color_manual(values = c(“blue”,“grey”,“red”))
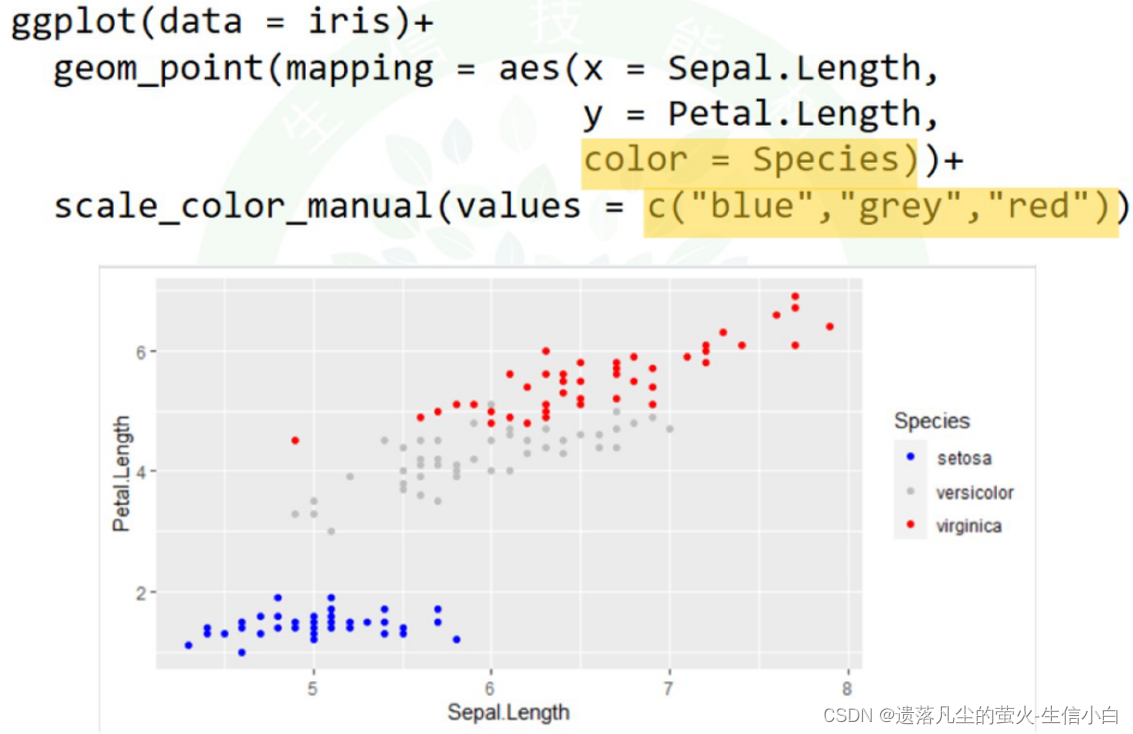
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species))+
scale_color_manual(values = c("blue","grey","red"))
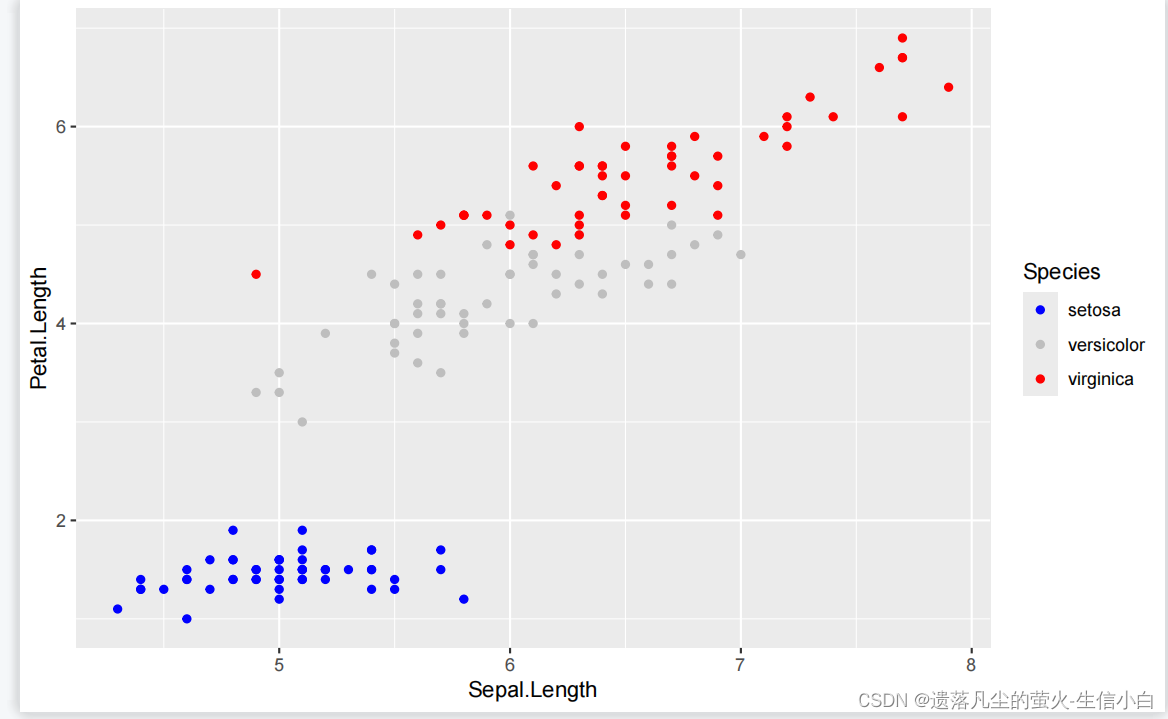
同理可得color可以换成别的属性,如shape,还可以用配色R包,如RColorBrewer,这个函数是可以直接+进ggplot的,其他的配色R包例如ggsci、paletteer
添加链接描述
#例如使用RColorBrewer R包,被加到了ggplot2里边,直接可用的颜色,scale_color_brewer()
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species))+
scale_color_brewer(palette = "Set1")
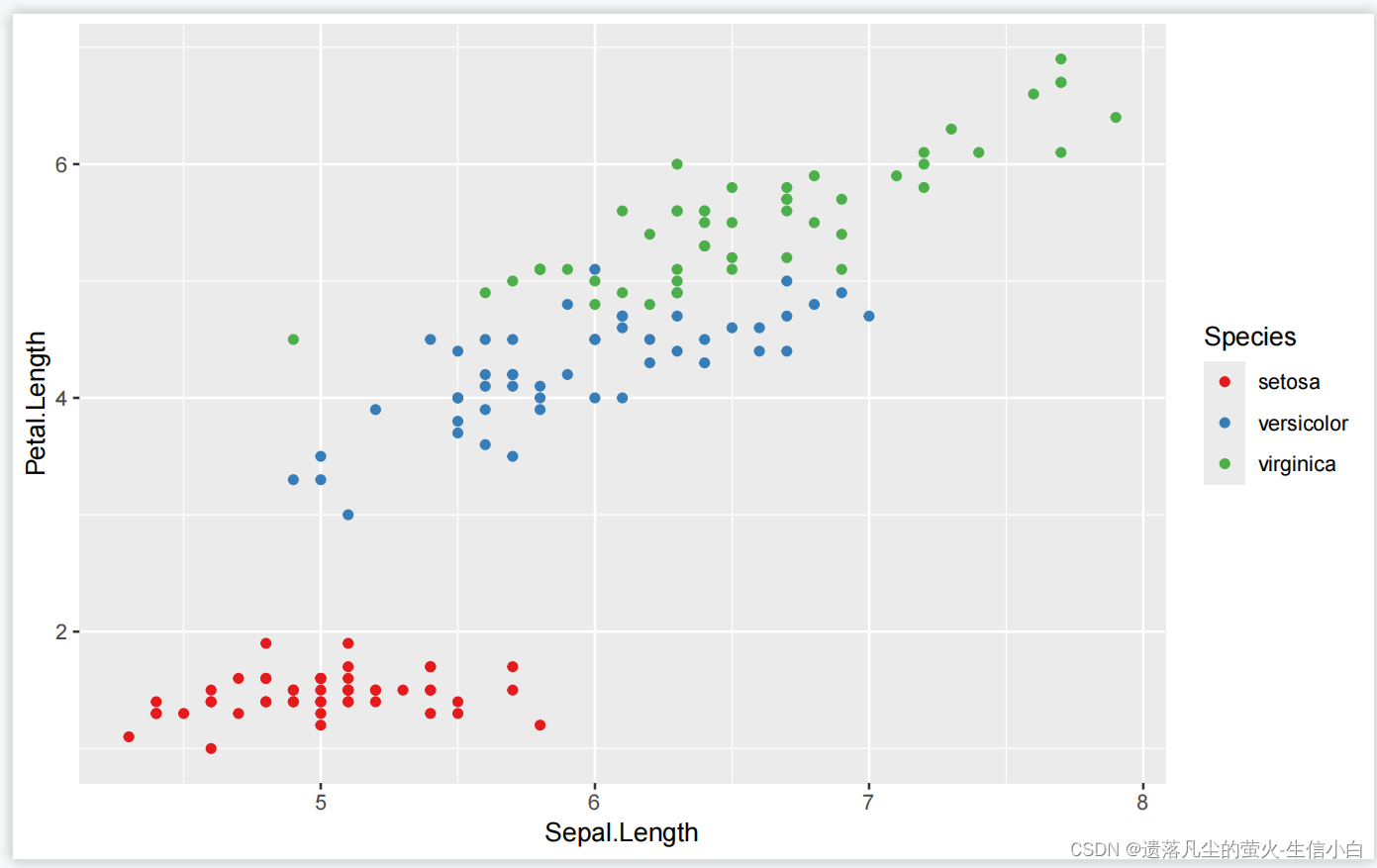
https://cloud.tencent.com/developer/article/1839444
#也可使用最强悍的配色R包paletteer
load("test.Rdata")
library(ggplot2)
library(paletteer)
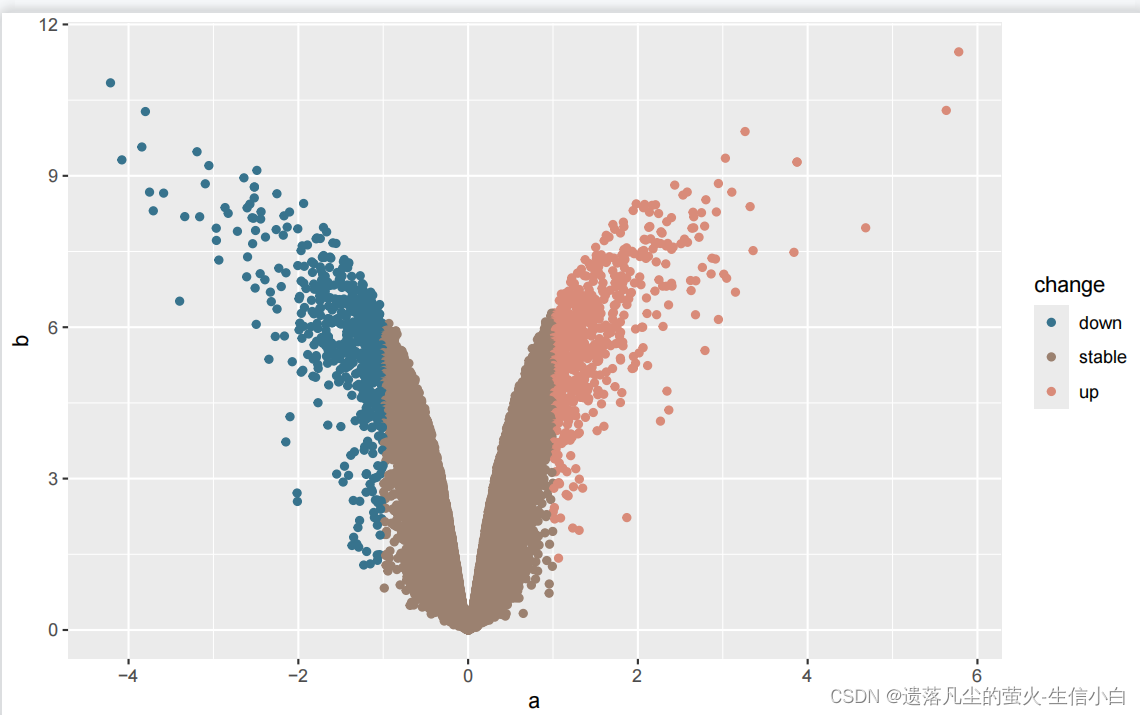
ggplot(data = test)+
geom_point(mapping = aes(x = a,
y = b,
color = change))+
scale_color_paletteer_d(`"ButterflyColors::parides_zacynthus_polymetus"`)
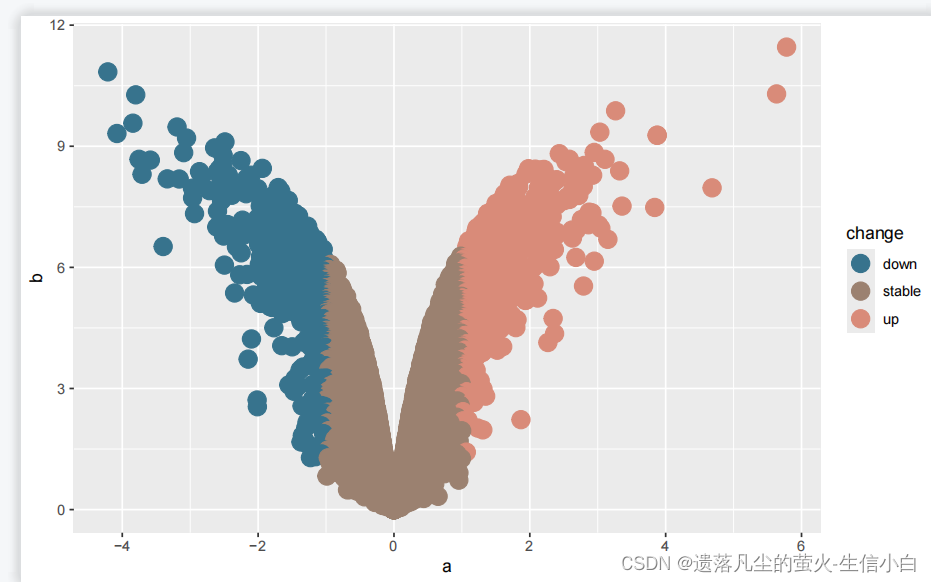
#改大小
ggplot(data = test)+
geom_point(mapping = aes(x = a,
y = b,
color = change),size = 5)+
scale_color_paletteer_d(`"ButterflyColors::parides_zacynthus_polymetus"`)
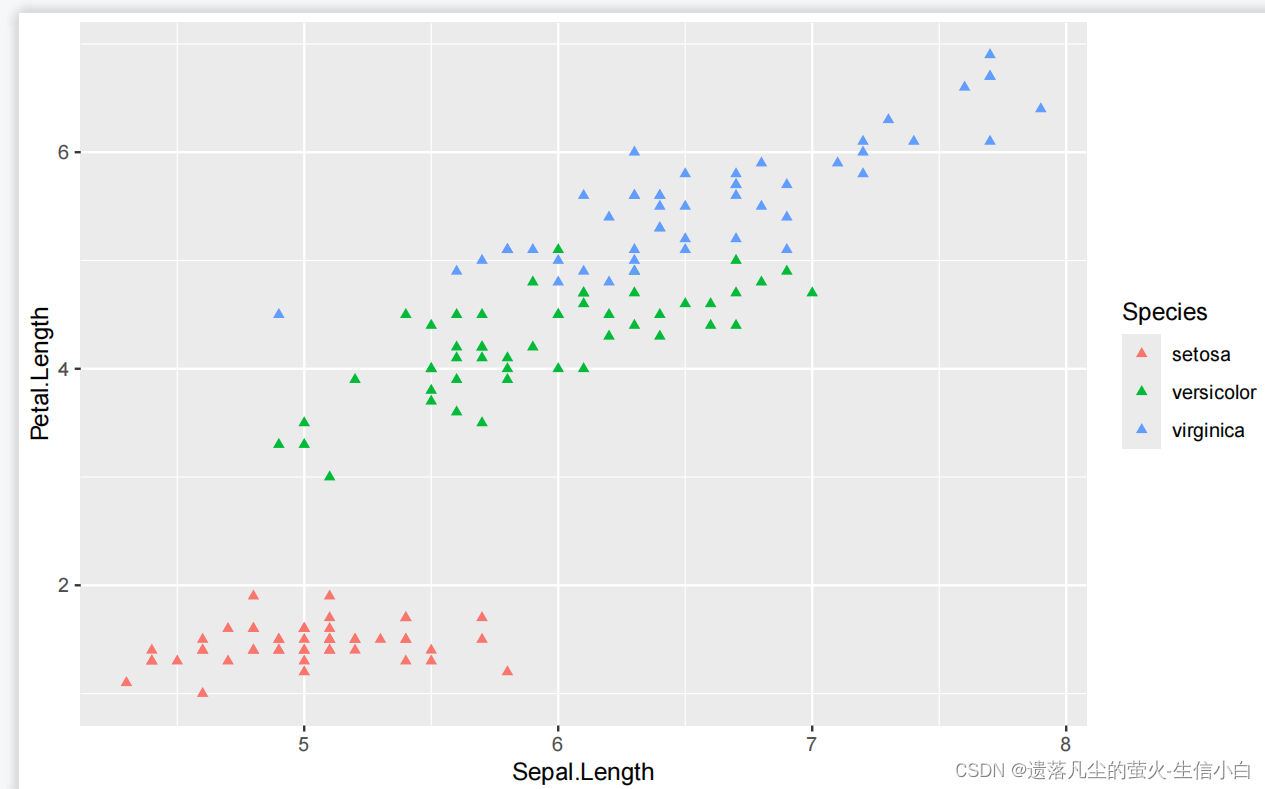
5.区分color和fill两个属性
1.空心形状和实心形状都用color设置颜色
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species),
shape = 17) #17号,实心的例子
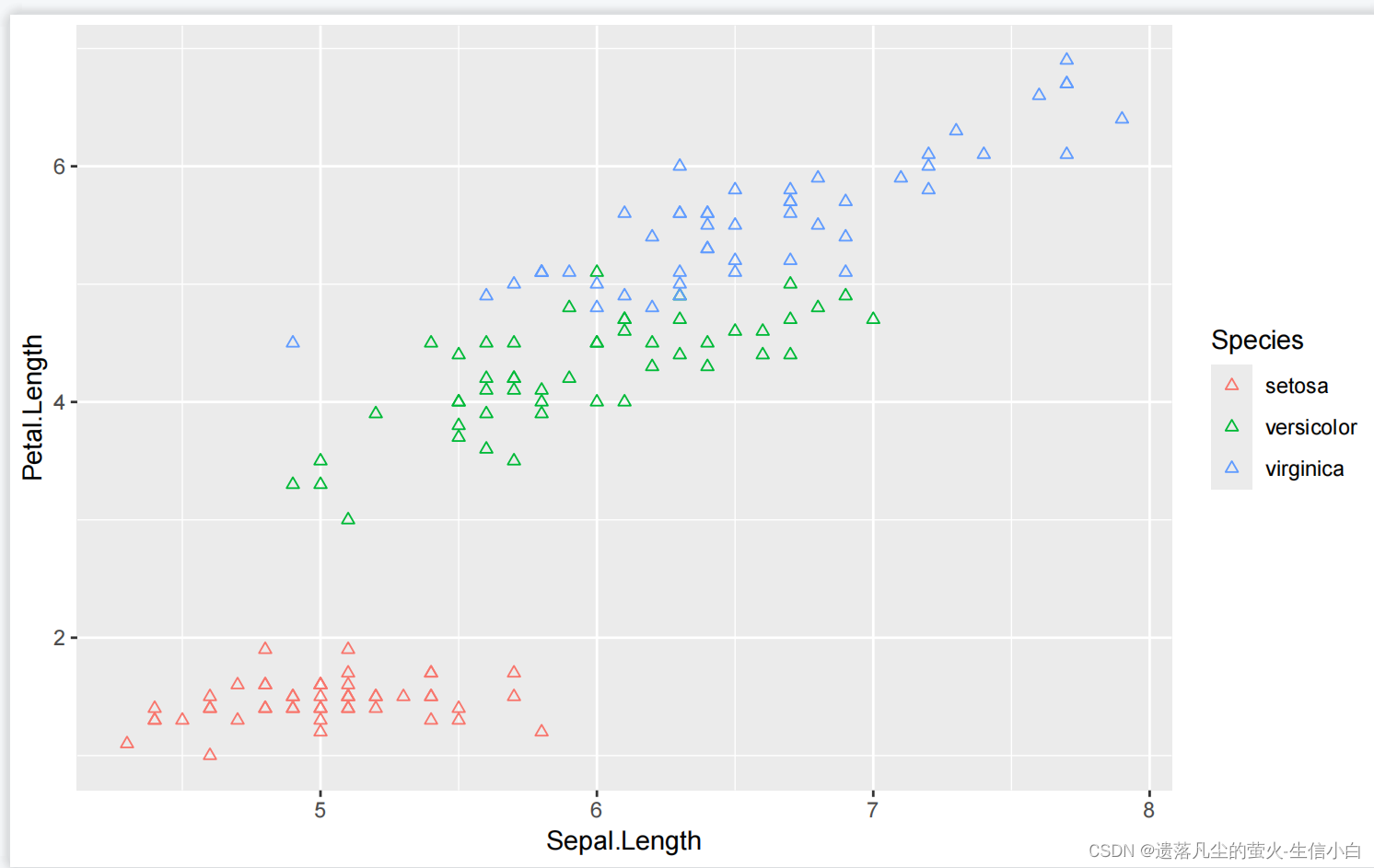
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species),
shape = 2) #2号,空心的例子
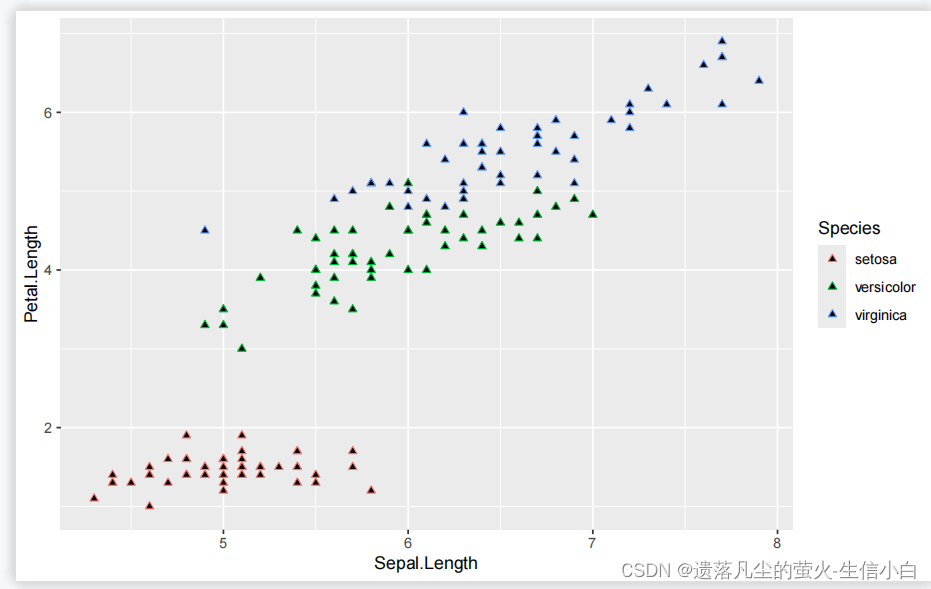
2.既有边框又有内心的,才需要color和fill两个参数
ggplot(data = iris)+
geom_point(mapping = aes(x = Sepal.Length,
y = Petal.Length,
color = Species),
shape = 24,
fill = "black") #24号,双色的例子
4.拼图
par里的mfrow
grid.arrage
cowplot
patchwork比较常用
5.导出
经典三段论
ggsave
eoffice-topptx
四、练习1(5_exercise.R)
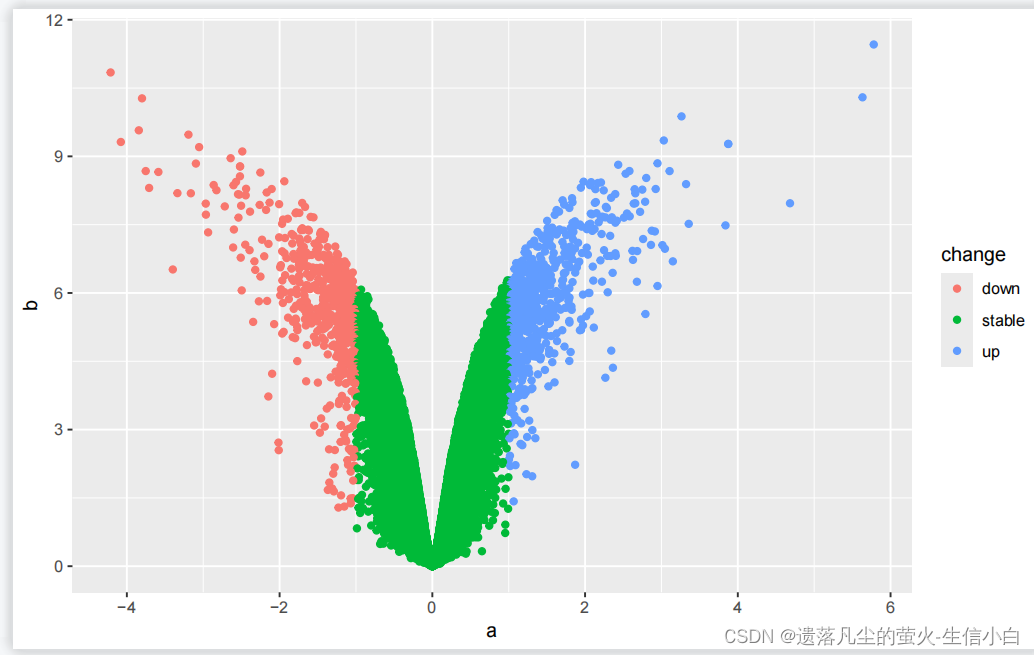
1.加载test.Rdata,分别test的以a和b列作为横纵坐标,change列映射颜色,画点图。
load("test.Rdata")
library(ggplot2)
ggplot(data = test)+
geom_point(mapping = aes(x = a,
y = b,
color = change))
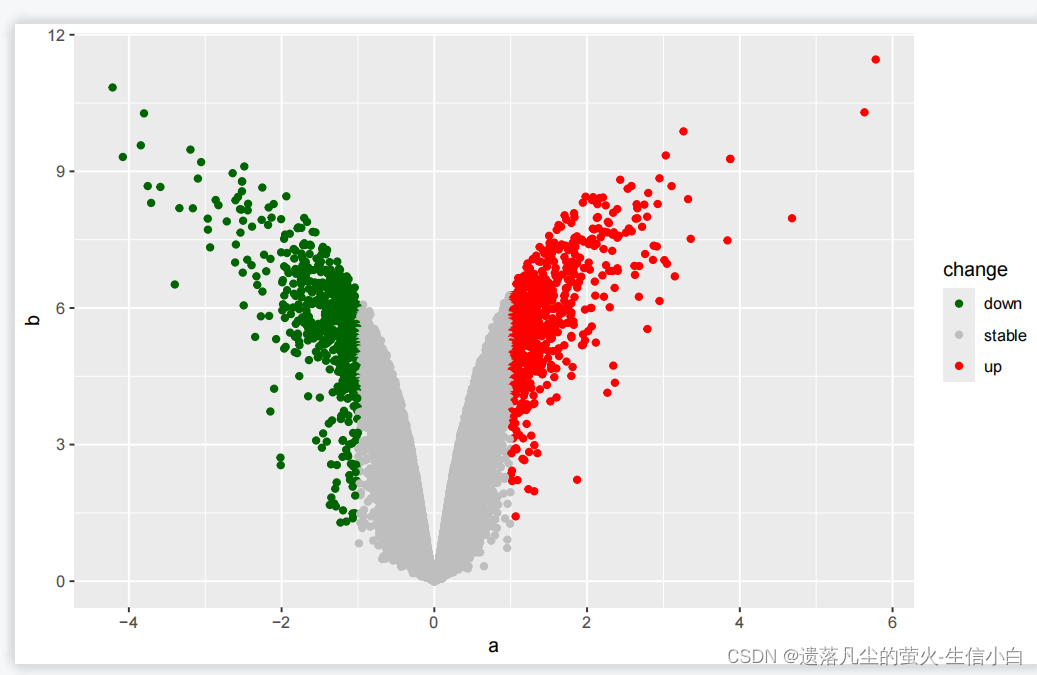
2.尝试修改点的颜色为暗绿色(darkgreen)、灰色、红色
ggplot(data = test)+
geom_point(mapping = aes(x = a,
y = b,
color = change))+
scale_color_manual(values = c("blue","grey","red"))
抽奖函数:
> sample(1:12,3)
[1] 11 2 10
#c实现首尾相连
paste0("挖掘", 1:14)
paste0("入门", 1:12)
c(paste0("挖掘", 1:14),paste0("入门", 1:12))
#抽奖
> a = c(paste0("挖掘", 1:14),paste0("入门", 1:12))
> sample(a,1)
[1] "挖掘8"
#抽奖作弊,使用set.seed
a = c(paste0("挖掘", 1:14),paste0("入门", 1:12))
set.seed(10)
sample(a,1)
练习2
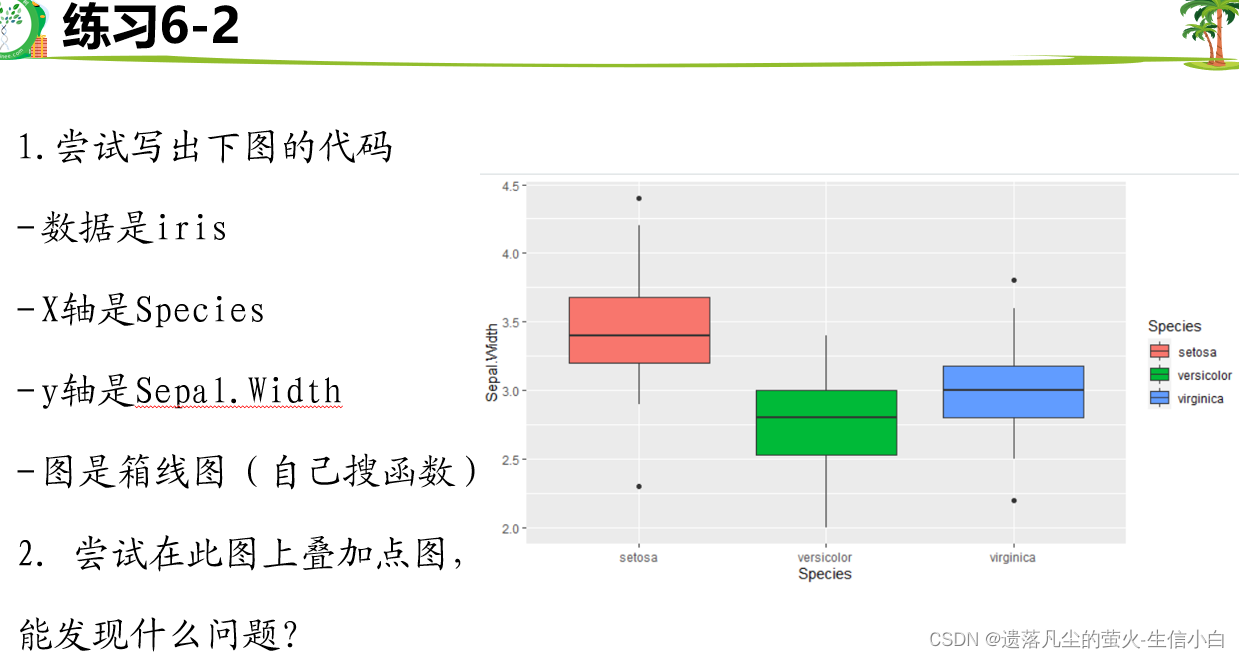
自己的答案:
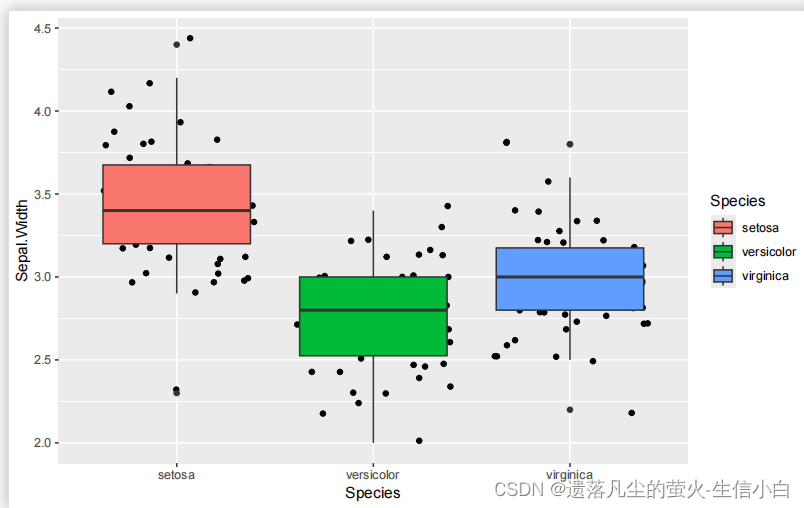
1.
library(ggplot2)
p = ggplot(iris, aes(x=Species, y=Sepal.Width)) +
geom_boxplot()
p
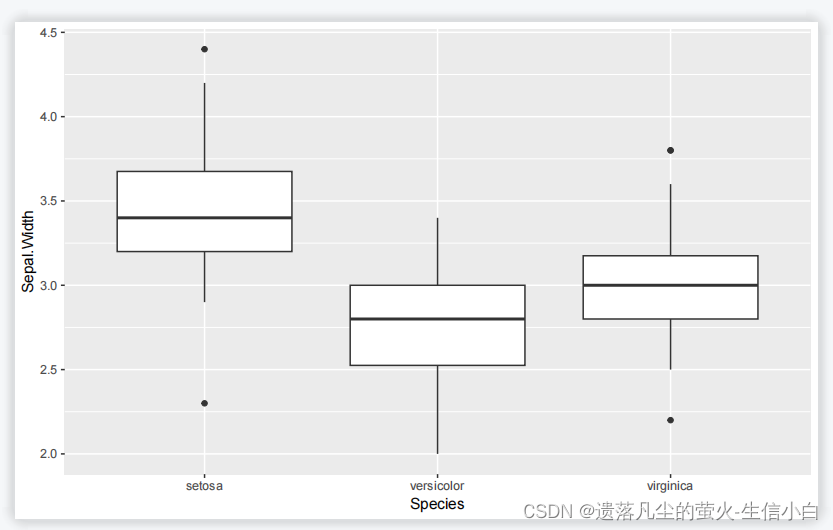
2.
#添加颜色
p5 = ggplot(iris, aes(x=Species, y=Sepal.Width,fill =Species)) +
geom_boxplot()
p5
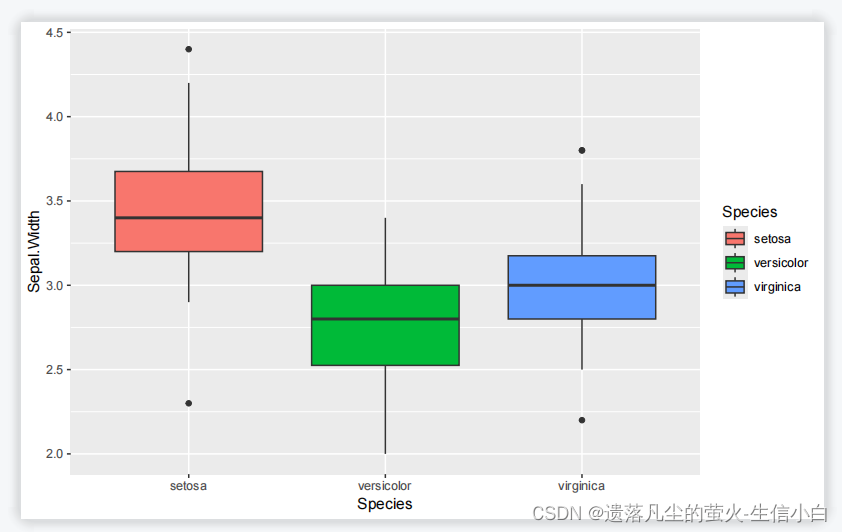
3.
#加点
p5 = ggplot(iris, aes(x=Species, y=Sepal.Width,fill =Species)) +
geom_jitter(shape=16, position = position_jitter(0.2)) +
geom_boxplot()
p5
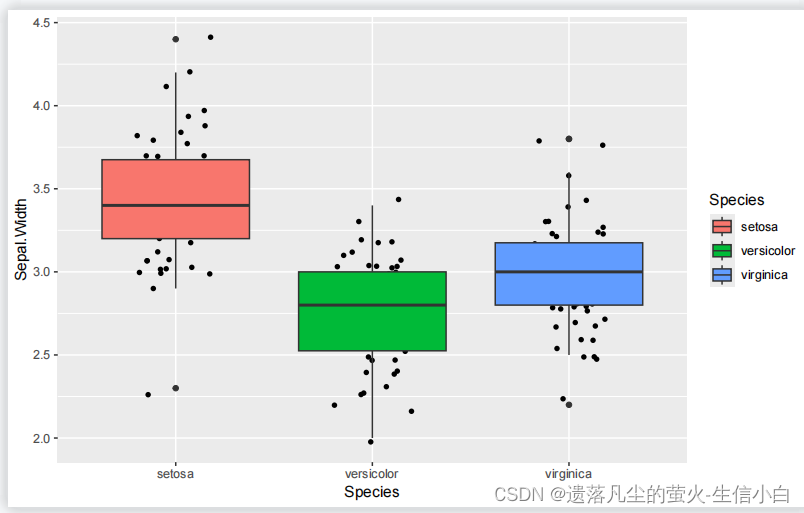
正确答案:
ggplot(data = iris,mapping = aes(x = Species,
y = Sepal.Width,
fill = Species)) +
geom_boxplot()
ggplot(data = iris,mapping = aes(x = Species,
y = Sepal.Width,
fill = Species)) +
geom_boxplot()+
geom_point()
#这样相同的点就不能分开
p5 = ggplot(iris, aes(x=Species, y=Sepal.Width,fill =Species)) +
geom_jitter() +
geom_boxplot()
p5
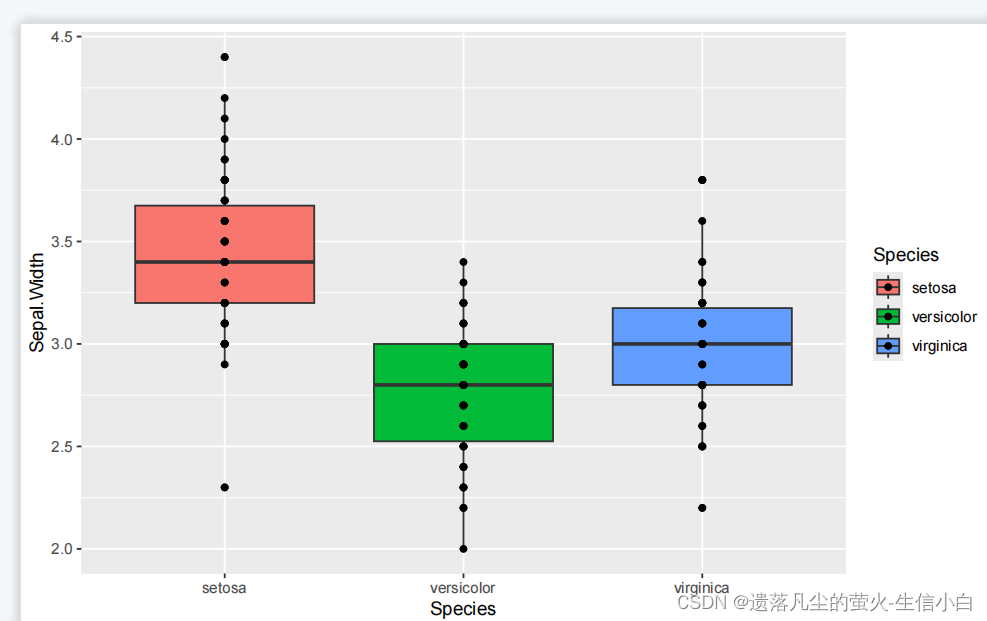
p5 = ggplot(iris, mapping =aes(x=Species, y=Sepal.Width,fill =Species)) +
geom_boxplot() +
geom_jitter()
p5
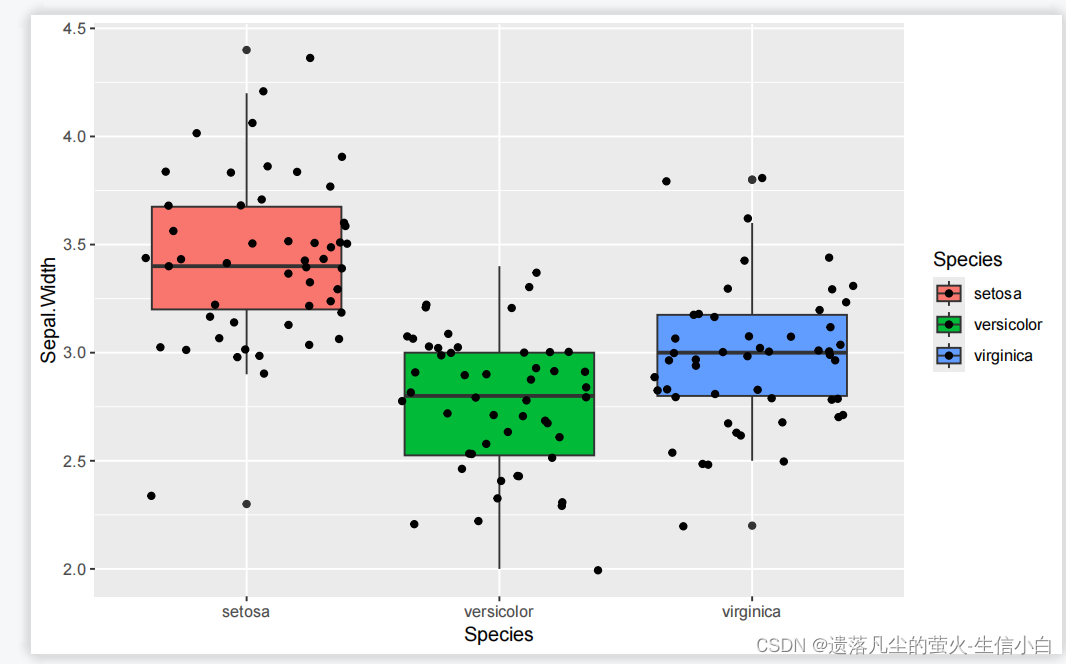
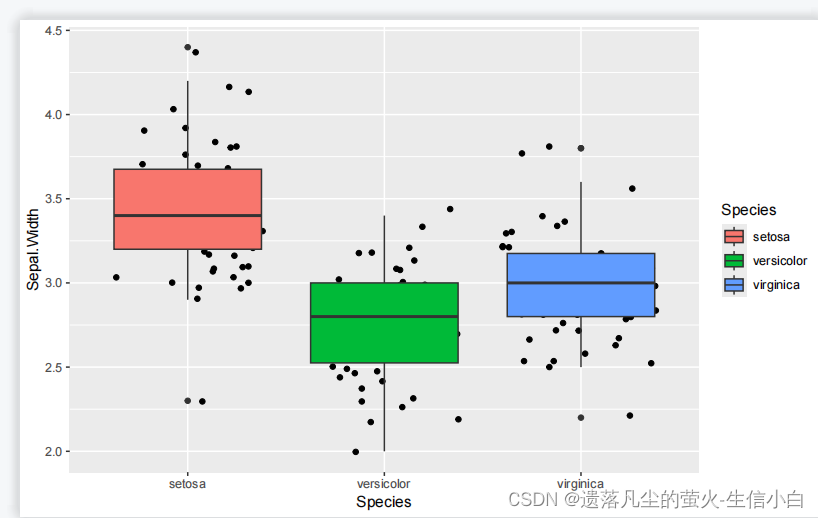
p5 = ggplot(iris, mapping =aes(x=Species, y=Sepal.Width,fill =Species)) +
geom_jitter() +
geom_boxplot()
p5