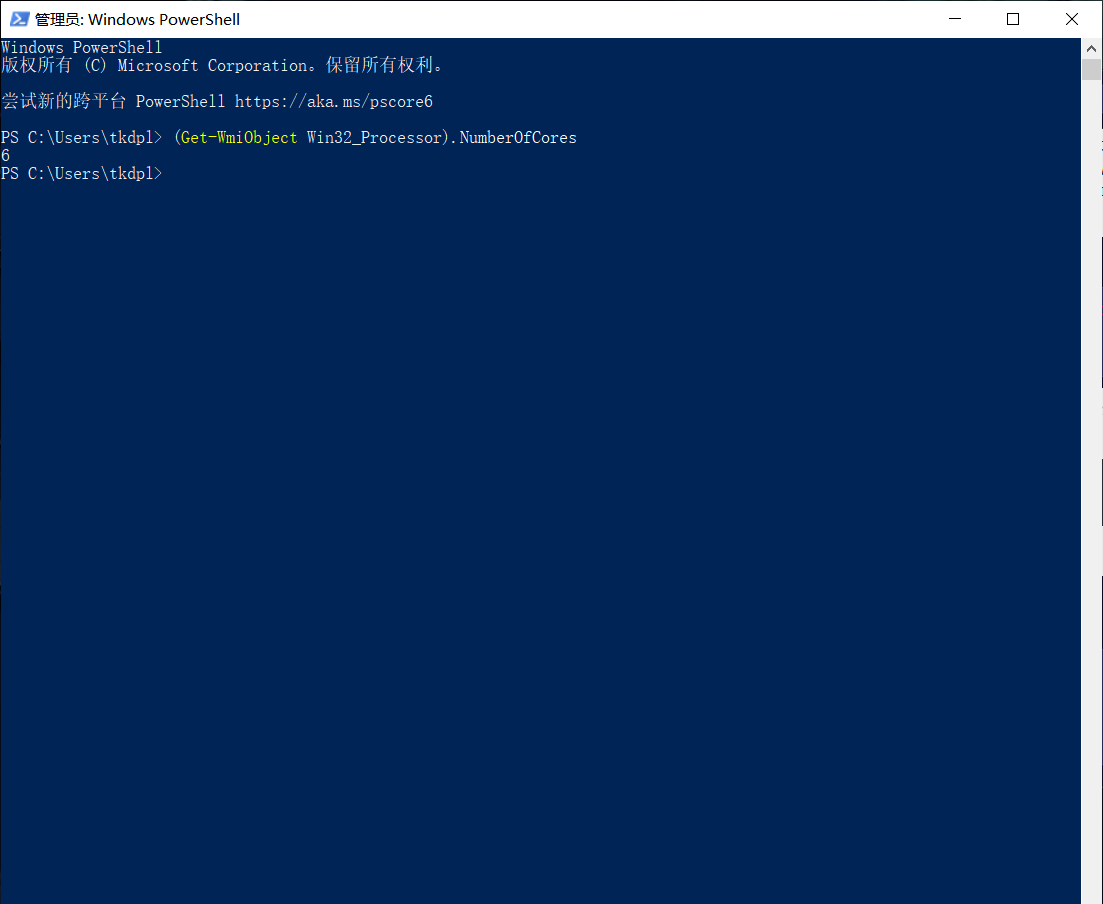
一. 使用R包“FactoMineR”进行主成分分析(PCA)
基本步骤如下:
- 安装和加载包:如果尚未安装,首先安装“FactoMineR”包,然后加载它:
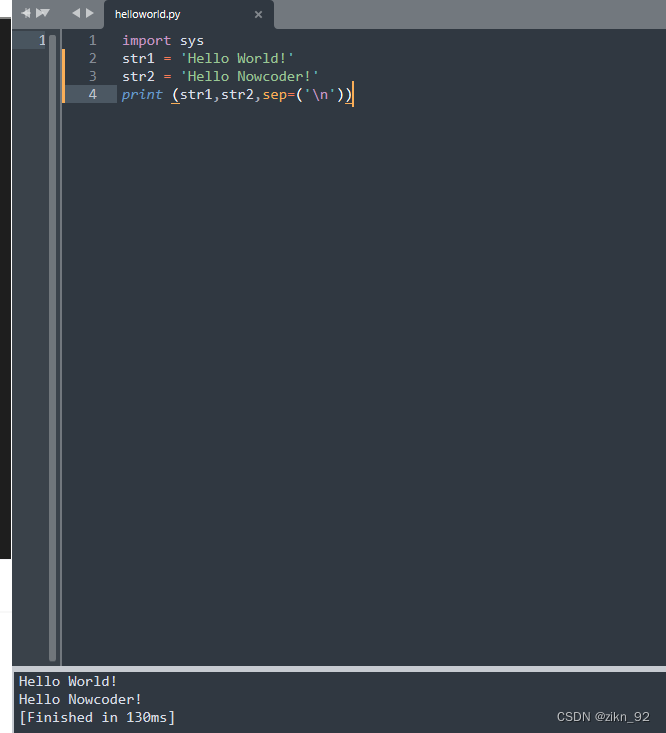
install.packages("FactoMineR")
library(FactoMineR)-
准备数据:确保你有一个数据框,其中包含你想要进行主成分分析的数值型变量。假设你的数据框名为
my_data。 -
执行主成分分析:使用
PCA()函数执行主成分分析。可以通过设置参数来指定一些选项,比如是否进行缩放、是否计算因子得分等。
# 执行主成分分析 pca_result <- PCA(my_data, scale.unit = TRUE, ncp = 5, graph = FALSE)在上述代码中:
my_data是你的数据框。scale.unit = TRUE将数据进行标准化。ncp = 5指定要提取的主成分数量,这里设定为5个主成分。graph = FALSE表示不生成默认的图形。
- 查看结果:主成分分析的结果存储在
pca_result对象中。你可以使用不同的函数来查看结果,比如summary()、eig()和dimdesc()等。
# 摘要 summary(pca_result)
# 查看每个主成分的解释方差 pca_result$eig
# 查看变量的因子载荷 pca_result$var$coord
# 查看变量的贡献度 pca_result$var$contrib- 绘制结果:你还可以使用
plot()函数绘制不同的图形,如散点图、因子载荷图等。
# 绘制散点图 plot(pca_result, choix = "ind")
# 绘制因子载荷图 plot(pca_result, choix = "var")通过这些步骤,你可以利用R包“FactoMineR”进行主成分分析,并从分析结果中获取所需的信息。
二. R包“Factoextra”是一个用于在R中进行因子分析和主成分分析结果可视化的工具
它提供了一些方便的函数,可以帮助用户更好地理解和呈现多元数据分析的结果。
以下是使用“Factoextra”包的一般步骤:
- 安装和加载包:如果尚未安装“Factoextra”包,首先安装它,然后加载它:
install.packages("factoextra") library(factoextra)-
准备数据:确保你已经进行了因子分析或主成分分析,并且已经有了相应的结果对象。例如,假设你的主成分分析结果存储在名为
pca_result的对象中。 -
可视化结果:使用“Factoextra”包中的函数来可视化分析结果。它提供了许多函数来绘制不同类型的图形,比如散点图、因子载荷图、变量贡献图等。
# 绘制个体散点图 fviz_pca_ind(pca_result)
# 绘制变量贡献图 fviz_contrib(pca_result, choice = "var")
# 绘制因子载荷图 fviz_pca_var(pca_result)这些只是“Factoextra”包中一些可用的函数。根据你的需要,你可能会使用其他函数来可视化其他方面的分析结果。该包提供了很多定制选项,可以帮助你创建具有专业外观的图形,以便更好地传达分析结果。
三. 使用“Factoextra”包中的函数绘制浓度图
在“Factoextra”包中,可以使用fviz_dist()函数来绘制浓度图。这个函数用于可视化样本之间的距离或相似度矩阵。
下面是使用fviz_dist()函数绘制浓度图的一般步骤:
-
准备距离矩阵:确保你有一个距离或相似度矩阵。可以使用一些距离度量或相似度度量来计算样本之间的距离或相似度,比如欧几里得距离、曼哈顿距离、相关系数等。
-
绘制浓度图:使用
fviz_dist()函数绘制浓度图。可以通过设置参数来调整图形的外观。
# 假设你有一个距离矩阵dist_matrix
# 绘制浓度图 fviz_dist(dist_matrix, gradient = list(low = "blue", mid = "white", high = "red"))在上述代码中:
dist_matrix是你的距离或相似度矩阵。gradient参数用于指定颜色渐变,你可以自定义颜色的低、中、高值,以便更好地表示样本之间的距离或相似度。
通过这些步骤,你可以使用“Factoextra”包中的fviz_dist()函数绘制浓度图来可视化样本之间的距离或相似度。