数据处理
数据质控
随机挑出九个序列进行比对,结果如下:
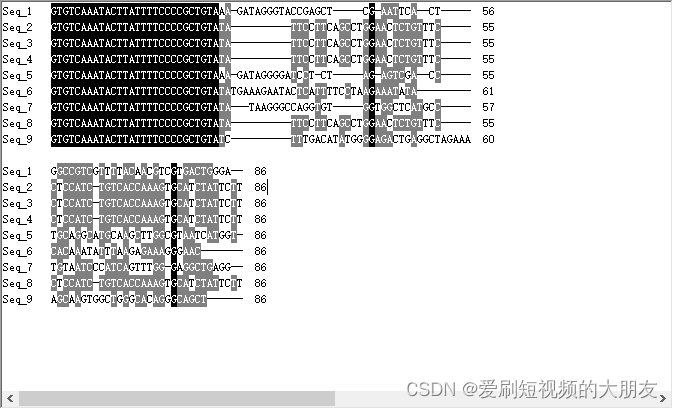 所有序列前面的部分序列均完全相同,怀疑是插入的转座子序列,再随机挑选9个序列进行比对,结果如下:
所有序列前面的部分序列均完全相同,怀疑是插入的转座子序列,再随机挑选9个序列进行比对,结果如下:

结果相同,使用cutadapt将该段序列修剪。
nohup cutadapt -g GTGTCAAATACTTATTTTCCCCGCTGTA -o ps_fastpTrimmed.cutadapt.fq PS_fastpTrimmed.fastq &
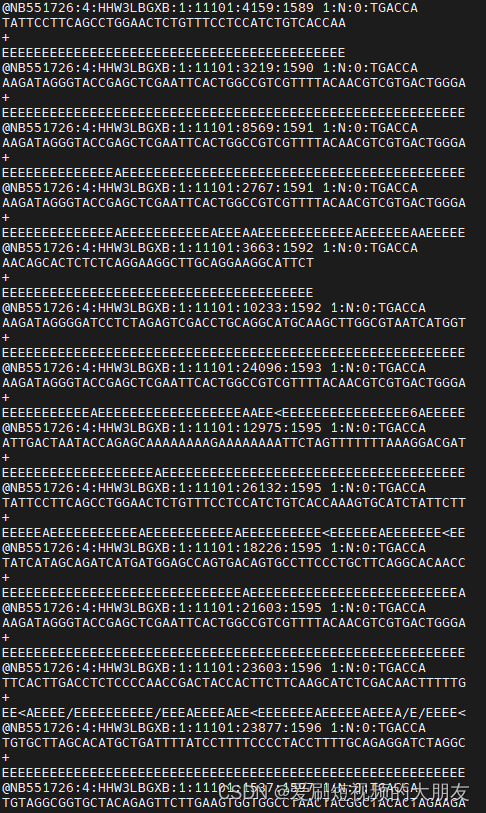 可以看到,大部分数据均经历了修剪,我们再随机选择部分数据进行比对,查看修剪情况。
可以看到,大部分数据均经历了修剪,我们再随机选择部分数据进行比对,查看修剪情况。

结果表明修剪成功。
比对
bwa mem -t 32 ../../001.blastFORcgspy/human.ref.bwaindex/GCF_000001405.40_GRCh38.p14_genomic.fna 002.ps_fastpTrimmed.cutadapt.fq > 003.ps_fastpTrimmed.cutadapt.sam &
(这里遇到了一点小bug,使用nohup挂后台的时候,居然把处理的过程信息输入到了我的结果文件中,导致我后面将sam文件转换为bam文件的时候一直在报错,去掉nohup后重新运行程序bug解除,可恶可恶!)
去除表头
awk '!/@SQ/' 003.ps_fastpTrimmed.cutadapt.sam > 003.ps_fastpTrimmed.cutadapt.1.sam &
查看比对结果
 可以看到,一些数据的比对质量良好,但也有部分数据未能成功比对。
可以看到,一些数据的比对质量良好,但也有部分数据未能成功比对。
反向验证1
公司提供的结果中,总共有6107个插入位点,并且未对其支持的reads数进行质控,见下图
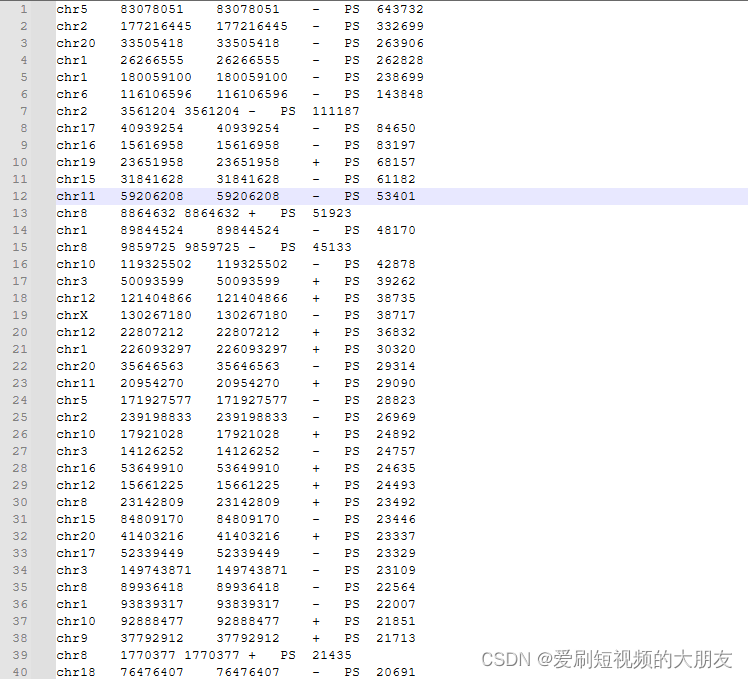 前三列是插入位点的染色体以及物理位置,第四列是正负链信息,第五列是插入的转座子名称,第六列是插入位点支持reads数。
前三列是插入位点的染色体以及物理位置,第四列是正负链信息,第五列是插入的转座子名称,第六列是插入位点支持reads数。
由于该数据中给出了插入位点的物理位置,我们可以根据给出的物理位置去验证是否存在对应的序列,可以使用bedtools工具的coverage功能统计给定的物理位置上覆盖的reads数。
数据预处理
首先,我们需要将比对结果的sam文件进行转换,使用samtools工具将比对结果的sam文件转换为bam文件
samtools view -bS 003.ps_fastpTrimmed.cutadapt.sam > 004.ps_fastpTrimmed.cutadapt.bam &
接着对bam文件进行排序
samtools sort 004.ps_fastpTrimmed.cutadapt.bam -o 005.psseq.sorted.bam &
然后,使用samtools的depth命令统计所有位点覆盖的reads数:
samtools depth 005.psseq.sorted.bam > 006.depth.txt &
公司的结果文件中,给的位点是以染色体号的形式命名的,而参考基因组是以染色体编码的形式命名的,所以需要进行替换。(为了方便,此步骤直接使用excel完成的,需要注意的是选择单元格匹配即可)
替换后,使用awk将位点的染色体号与物理位置合并,获得一个只有物理位置的结果文件(文件1),和一个基与比对文件生成的具有物理位置和reads数的文件(文件2)。(实现类似于excel中vlookup函数的功能)
#准备需要查找(第一列)并返回对应值(第二列)文件
awk '{print $1 "+"$2 , $3}' 006.psseq.depth.txt > 006.psseq.depth.1.txt
#准备待查找的文件(仅一列)
awk '{print $1 "+"$2}' ps_reducedIns.1.txt > ps_reducedIns.merged.txt
#可选项,不影响查找,但是排序后更加便于观察
sort -o 006.psseq.depth.1.sorted.txt -t $'\t' -k1,1 006.psseq.depth.1.txt
使用awk实现excel中的vlookup函数的功能。
awk 'FNR==NR {arr[$1] = $2; next} {print $0, arr[$1]}' 006.psseq.depth.1.txt ps_reducedIns.merged.txt > psseq.psresult.overlap.txt
观察结果
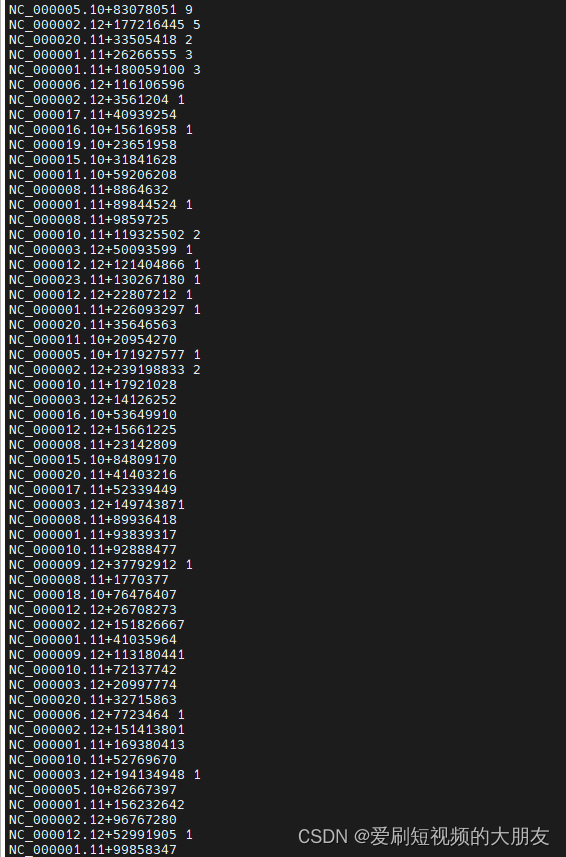
使用excel进行统计分析

在ps的6107个结果中,仅有183(2.9%)个位点能找到对应的reads数,在之前生成的depth文件中手动查找验证那些找不到reads数的位点用以核实是否代码是否有问题,结果表明:这些空值位点在depth文件中都找不到。
使用ZB的结果对PS的测序比对结果进行匹配,流程如上所属
#准备待查找的文件(仅一列)
awk '{print $1 "+"$2}' zb_reducedIns.1.txt > zb_reducedIns.merged.txt
awk 'FNR==NR {arr[$1] = $2; next} {print $0, arr[$1]}' 006.psseq.depth.1.txt zb_reducedIns.merged.txt > psseq.zbresult.overlap.txt
查看结果
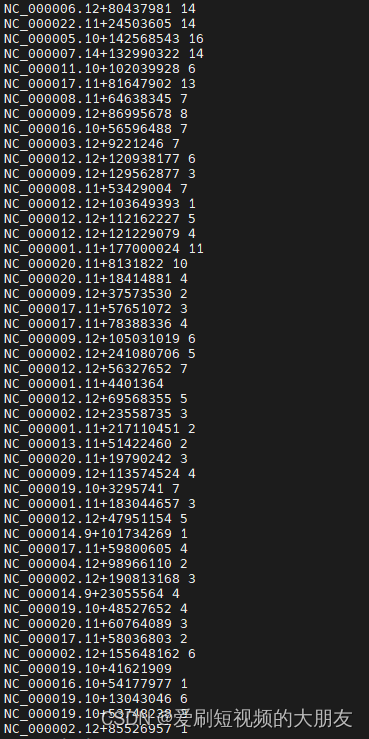
居然很多都能找到对应的位点,使用excel统计一下
在142694个位点中,有21795(15.2%)个位点能够找到,提示PS和ZB的结果与测序文件结果给反了。继续进行多重验证
分析ZB的结果
观察ZB的测序结果

同样存在转座子序列,进行修剪
cutadapt -g TGCGCCTTATAGTGCGGAAAATACGGTA -o zb_fastpTrimmed.cutadapt.fq ../ZB_w_118_fastpTrimmed.fastq &
表明修剪成功
进行比对
bwa mem -t 32 ../../001.blastFORcgspy/human.ref.bwaindex/GCF_000001405.40_GRCh38.p14_genomic.fna zb_fastpTrimmed.cutadapt.fq > 003.zb_fastpTrimmed.cutadapt.sam &
samtools view -bS 003.zb_fastpTrimmed.cutadapt.sam > 004.zb_fastpTrimmed.cutadapt.bam &
samtools sort 004.zb_fastpTrimmed.cutadapt.bam -o 004.zbseq.sorted.bam &
查找
awk 'FNR==NR {arr[$1] = $2; next} {print $0, arr[$1]}' 006.zbseq.depth.merged.txt zb_reducedIns.merged.txt > zbseq.zbresult.overlap.txt &
awk 'FNR==NR {arr[$1] = $2; next} {print $0, arr[$1]}' 006.zbseq.depth.merged.txt ps_reducedIns.merged.txt > zbseq.psresult.overlap.txt &
查看结果
在公司给出的142694个ZB位点中,在ZB测序数据中仅能找到196个(0.13%)位点具有reads数支持。
而在公司给出的6107个PS位点中,在ZB测序数据中能找到2042个(33.4%)位点具有reads数支持。
反向验证2
由于反向验证1的结果提示PS和ZB的测序数据与给出的位点结果可能反了,使用bedtools的coverage功能直接调出该位点覆盖的reads数。
数据预处理
需要将公司给出的位点的结果的染色体号和物理位置信息提取出来,做成bed格式,用于bedtools数据处理。
bedtools coverage -a ps_reducedIns.1bp.txt -b 005.psseq.sorted.bam > psseq.psresult.readsnumber.bed &
bedtools coverage -a zb_reducedIns.1bp.txt -b 005.psseq.sorted.bam > psseq.zbresult.readsnumber.bed &
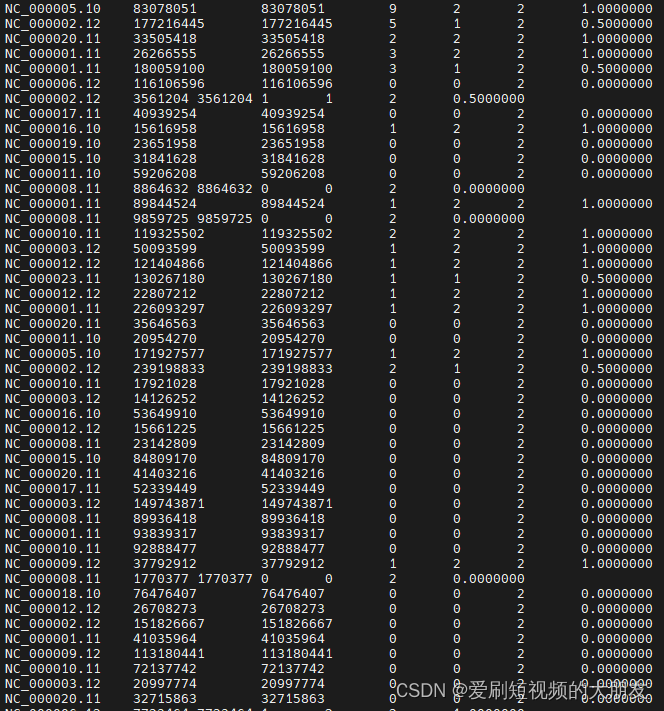
这里是公司给的ps位点对应的ps测序数据的reads结果,可以看到很多位点都没有对应的reads数覆盖。使用excel进行统计,发现在公司给出的6107个位点中,在ps测序数据中仅能找到191(3.1%)个位点具有覆盖的reads。与之前的结果类似。
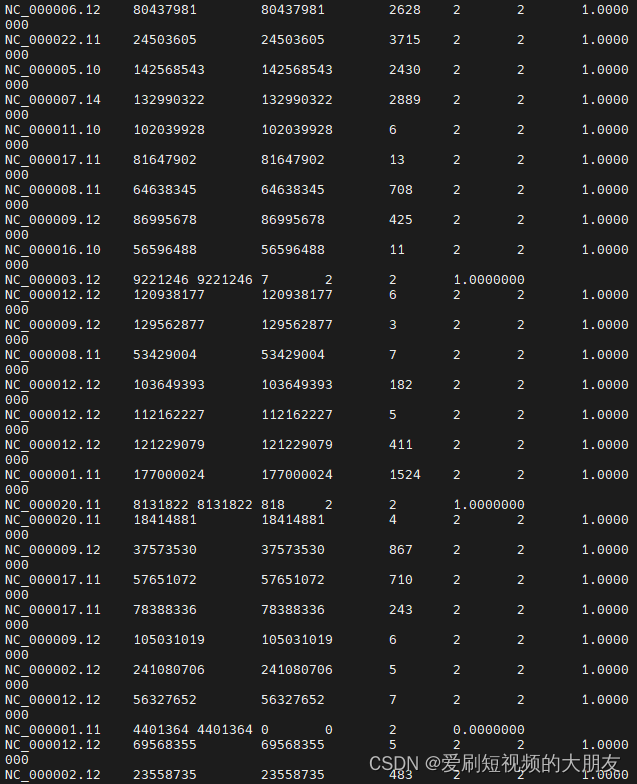
这里是公司给的zb位点对应的ps测序数据的reads结果,可以看到reads数覆盖的位点明显变多。使用excel进行统计,发现在公司给出的142694个位点中,在ps测序数据中能找到42302(29.6%)个位点具有覆盖的reads。与之前的结果类似。
对ZB的测序数据开展类似处理
在公司的给出的6107个ps位点中,有2102(34.4%)个位点在zb测序数据中具有覆盖的reads,而在公司给出的142694个zb微电子红,有200(0.14%)个位点在zb测序数据中具有覆盖的reads,与反向验证1的结果类似。
由于怀疑是基因组版本不一样导致位点数的物理位置出现了变化,进而导致了这些位点找不到覆盖的reads数,尝试扩展位点附近的序列进行覆盖书统计。
先查看插入位点极其上下5bp的序列的覆盖情况。数据准备流程如前所述
在公司给出的6107个ps位点中,在ps测序数据中仅能找到212个(3.4%)位点具有覆盖的reads。而在公司给出的142694个zb位点中,在ps测序数据中能找到142518个(99.8%)位点具有覆盖的reads。
在公司给出的6107个ps位点中,在zb测序数据中能找到6105个(99.9)位点具有覆盖的reads。而在公司给出的142694个zb位点中,在zb测序数据中仅能找到230(0.16%)位点具有覆盖的reads。
以上结果表明,结合插入位点附近的位点用于查看,覆盖率明显上升(zbseq-psresult,psseq-zbresult),表明可能确实是由基因组版本导致插入位点的统计出现了微小差异,并且测序数据与对应的额结果给反了。于是,我们查询了原文给出的参考基因组版本,使用的UCSC数据中的参考基因组,稍后正向验证的时候下载UCSC数据库中的参考基因组进行正向分析
正向分析
正向分析使用统计比对上的reads的边缘位点的策略,由于sam文件仅给出了比对到基因组上的最左边碱基的物理位置,所以对于匹配到正脸的数据,取其最匹配上的第一个碱基的物理位置即可,而对于匹配到负链的数据,使用匹配上的第一个碱基的物理位置加上比对上的碱基数即可得出匹配到负链的实际插入位点的物理位置。分析流程如下:
接之前的比对结果
#去除表头
awk '!/@SQ/' 003.ps_fastpTrimmed.cutadapt.sam > 004.psseq.1.sam &
#去除首行
awk '!/@PG/' 004.psseq.1.sam > 005.psseq.2.sam &
#去除没有比对上的数据
awk '$4 != 0' 005.psseq.2.sam > 006.psseq.3.sam
#数据从42497873行变为了31422261,约22%的数据没有比对上#
#提取数据的前六行信息用于分析
awk '{print $1, $2, $3, $4, $5, $6}' 006.psseq.3.sam > 007.psseq.4.sam &
统计一下比对到±链的reads数量
awk '{count[$2]++} END {for (word in count) print word, count[word]}' 007.psseq.4.sam > 008.strandnumber &
0 8280752
16 23141011
2048 292
2064 206
分别提取比对到正链和负链的信息用于分析
#提取正链
awk '$2 == 0' 007.psseq.4.sam > 009.psseq.5.+.sam &
#提取负链
awk '$2 == 16' 007.psseq.4.sam > 009.psseq.5.-.sam &
统计位于正链的插入位点的数量
#将染色体号和物理位置连接起来,防止将不同染色体的相同物理位置统计到一起
awk '{print $1 , $2 , $3 "+" $4 , $5 ,$6}' 009.psseq.5.+.sam > 009.psseq.5.+.1.sam &
#统计插入位点的数量
awk '{count[$3]++} END {for (word in count) print word, count[word]}' 009.psseq.5.+.1.sam > 009.psseq.5.+.2.txt &
#去除reads数小于5的位点
awk '$2 > 4 {print}' 009.psseq.5.+.2.txt > 009.psseq.5.+.3.txt &
将结果比对到公司给的ps结果中,仅有2个位点有reads数
而在比对到公司给的zb结果中,有70个位点有reads数
数量实在太少,可能是之前的质控丢失了部分数据。
@SQ SN:NC_000001.11 LN:248956422
@SQ SN:NC_000002.12 LN:242193529
@SQ SN:NC_000003.12 LN:198295559
@SQ SN:NC_000004.12 LN:190214555
@SQ SN:NC_000005.10 LN:181538259
@SQ SN:NC_000006.12 LN:170805979
@SQ SN:NC_000007.14 LN:159345973
@SQ SN:NC_000008.11 LN:145138636
@SQ SN:NC_000009.12 LN:138394717
@SQ SN:NC_000010.11 LN:133797422
@SQ SN:NC_000011.10 LN:135086622
@SQ SN:NC_000012.12 LN:133275309
@SQ SN:NC_000013.11 LN:114364328
@SQ SN:NC_000014.9 LN:107043718
@SQ SN:NC_000015.10 LN:101991189
@SQ SN:NC_000016.10 LN:90338345
@SQ SN:NC_000017.11 LN:83257441
@SQ SN:NC_000018.10 LN:80373285
@SQ SN:NC_000019.10 LN:58617616
@SQ SN:NC_000020.11 LN:64444167
@SQ SN:NC_000021.9 LN:46709983
@SQ SN:NC_000022.11 LN:50818468
@SQ SN:NC_000023.11 LN:156040895
@SQ SN:NC_000024.10 LN:57227415
awk '{count[$7]++} END {for (word in count) print word, count[word]}' 002.readsNUMBER.ZB.bed > freq.txt