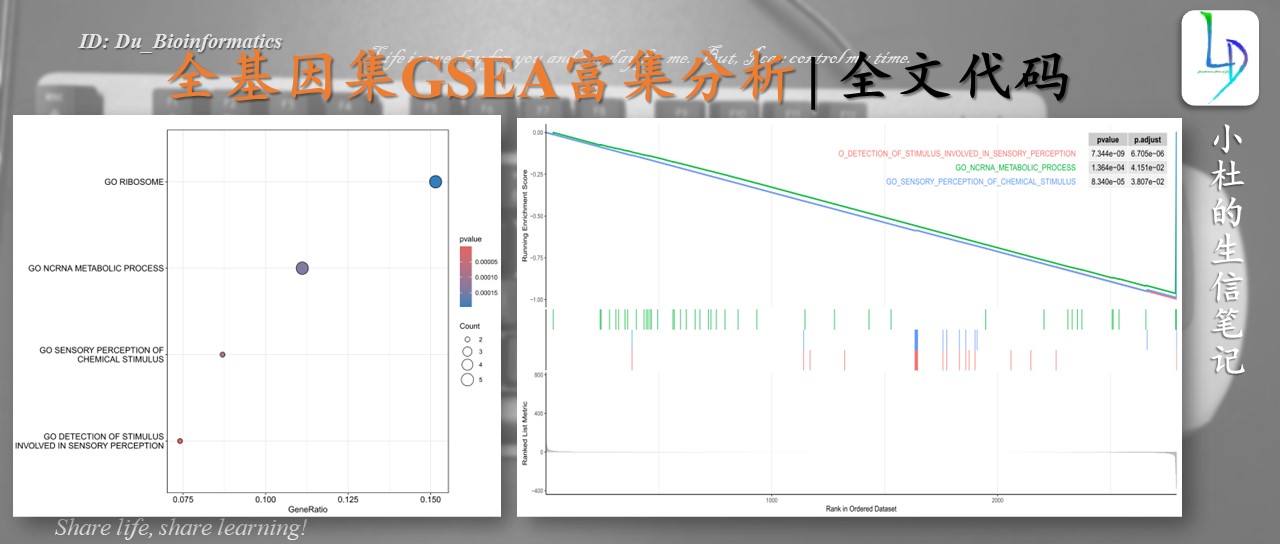
原文链接:一文完成全基因集GSEA富集分析
本期内容
写在前面
我们前面分享过一文掌握单基因GSEA富集分析的教程,主要使用单基因的角度进行GSEA富集分析。
我们社群的同学咨询,全基因集的GSEA如何分析呢??其实,原理都是大同小异的,那么今天我们就简单的整理一下吧。
若我们的分享对你有用,希望您可以点赞+收藏+转发,这是对小杜最大的支持。
GSEA知识回顾
GSEA网址
https://www.gsea-msigdb.org/gsea/index.jsp

分析原理
GSEA的分析原理,我们这里使用“生信宝典”陈同老师分享的教程,一文掌握GSEA,超详细教程。
1. GSEA定义
GSEA用来评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。其输入数据包含两部分,一是已知功能的基因集 (可以是GO注释、MsigDB的注释或其它符合格式的基因集定义),二是表达矩阵,软件会对基因根据其于表型的关联度(可以理解为表达值的变化)从大到小排序,然后判断基因集内每条注释下的基因是否富集于表型相关度排序后基因表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。
2. GSEA原理
给定一个排序的基因表L和一个预先定义的基因集S (比如编码某个代谢通路的产物的基因, 基因组上物理位置相近的基因,或同一GO注释下的基因),GSEA的目的是判断S里面的成员s在L里面是随机分布还是主要聚集在L的顶部或底部。这些基因排序的依据是其在不同表型状态下的表达差异,若研究的基因集S的成员显著聚集在L的顶部或底部,则说明此基因集成员对表型的差异有贡献,也是我们关注的基因集。

3. GSEA计算的关键概念
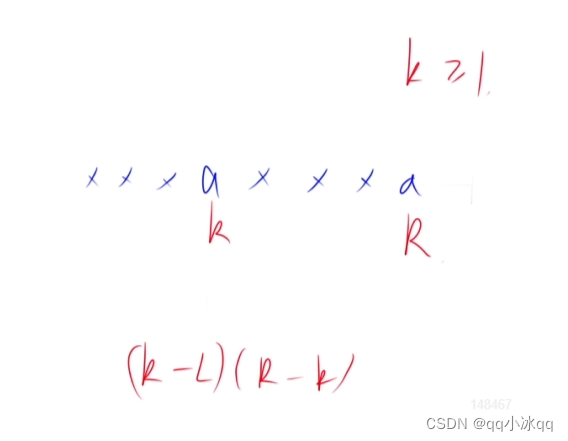
- 计算富集得分 (ES, enrichment score). ES反应基因集成员s在排序列表L的两端富集的程度。计算方式是,从基因集L的第一个基因开始,计算一个累计统计值。当遇到一个落在s里面的基因,则增加统计值。遇到一个不在s里面的基因,则降低统计值。每一步统计值增加或减少的幅度与基因的表达变化程度(更严格的是与基因和表型的关联度)是相关的。富集得分ES最后定义为最大的峰值。正值ES表示基因集在列表的顶部富集,负值ES表示基因集在列表的底部富集。

- 评估富集得分(ES)的显著性。通过基于表型而不改变基因之间关系的排列检验 (permutation test)计算观察到的富集得分(ES)出现的可能性。若样品量少,也可基于基因集做排列检验 (permutation test),计算p-value。

- 多重假设检验矫正。首先对每个基因子集s计算得到的ES根据基因集的大小进行标准化得到Normalized Enrichment Score (NES)。随后针对NES计算假阳性率。(计算NES也有另外一种方法,是计算出的ES除以排列检验得到的所有ES的平均值)

- Leading-edge subset,对富集得分贡献最大的基因成员。
4. 与GO或KEGG富集分析的区别
GO富集分析是先筛选差异基因,再判断差异基因在哪些注释的通路存在富集;这涉及到阈值的设定,存在一定主观性并且只能用于表达变化较大的基因,即我们定义的显著差异基因。
GSEA则不局限于差异基因,从基因集的富集角度出发,理论上更容易囊括细微但协调性的变化对生物通路的影响。
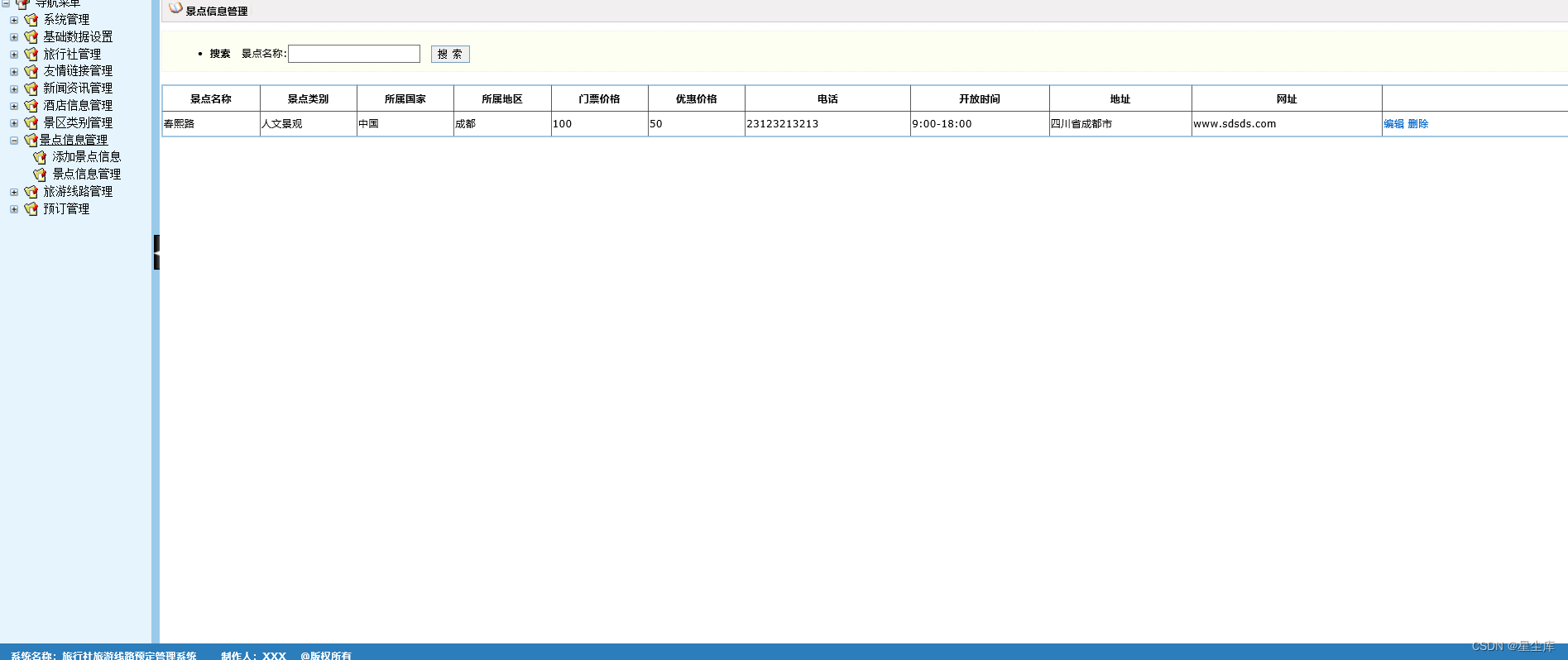
GSEA富集结果如何解读
GSEA分析的标准图形,该图分为3部分,最上面部分为基因EnrichmentScore的折线图,从左至右,每个基因计算一个ES值,连成线。横轴为该基因下的每个基因,纵轴为对应基因的RunningES。 在折线图中有个峰值,该峰值就是这个基因集的Enrichemntscore。一般关注基因集的Enrichemntscore,峰出现在前端还是后端(ES值大于0在前端,小于0在后端),和Leading-edge subset (即对富集贡献最大的部分,领头亚集)。如果ES值大于0,则峰值前的基因为Leading-edge subset ;如果ES值小于0,则峰值后的基因为Leading-edge subset。在ES图中出现领头亚集的形状,表明这个功能基因集在某处理条件下具有更显著的生物学意义。如果峰值出现在正值处,认为峰值之前的基因就是该基因集下的核心基因。图中我们一般关注ES值,峰出现在前端还是后端(ES值大于0在前端,小于0在后端)以及Leading-edge subset(即对富集贡献最大的部分);在ES图中出