| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
文章目录
前言
本文是UCSF DOCK的使用案例分享,关于DOCK中基于配体-靶标相互作用的遗传算法用于分子生成,模块名称 DOCK_GA。利用进化原理的构建方法, 包括分子重组、突变和自然选择, 以指导与靶标有特定相互作用的配体的从头设计。DOCK_GA可用于:
(1)生成与靶标具有特定相互作用的结构类似物;
(2)作为从分子片段生长分子的从头设计方法;
(3)作为筛选靶向结合位点分子的方法的补充。
一、软件及操作环境
操作环境: Ubuntu 22.04
软件版本:
UCSF DOCK 6.11,安装可以参考这篇博文;
UCSF Chimera 1.17.3,UCSF ChimeraX 1.7.1,安装可以参考这篇博文。
作业管理Slurm:可参考作业调度开源软件Slurm的安装与使用
二、遗传算法
遗传算法是达尔文关于生物进化理论的一种算法,在个体突变的基础上控制引导种群进化方向,在算法上用来解决全局优化的问题。
遗传算法的介绍可参考:遗传算法详解 附python代码实现
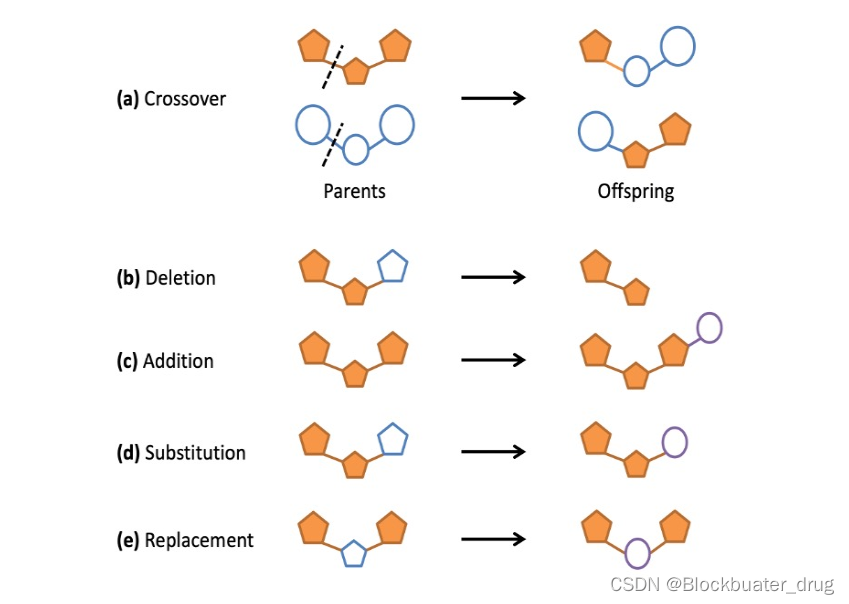
在本实例中,我们将继续以布鲁顿氏酪氨酸激酶(BTK)蛋白作为靶点,使用DOCK_GA模块,完成从配体生成片段到构建新配体。
三、结构文件准备
输入文件来自于前一篇案例分享:UCSF DOCK 分子对接详细案例(01)
新建本案例的目录:
cd DOCK_workdir
mkdir 013_DOCK_GA && cd 013_DOCK_GA
cp ../001_structure/up9_rec_withH.mol2 ./
四、 DOCK_GA
使用DOCK_GA,首先生成配体片段库。
片段库是具有连接点(Dummy atom,Du表示)的基团,可以用Chimera或者ChimeraX查看,Du分别用洋红色与灰色表示。DOCK为了以实现更好的可视化,使用fragment_library_trans_origin参数将所有输出片段平移到原点,并以容易观察的方式展示给用户。
4.1 Fragment Library Generation
与之前的运行方式类似,我们首先生成一个参数输入文件,然后用dock6运行根据需要完善参数,检查运行,最后正式运行。
mkdir fraglib && cd fraglib
建立输入空文件:
touch 8u2e.fraglib.in
参考一下参数,设置输入参数。
conformer_search_type flex
write_fragment_libraries yes
fragment_library_prefix 8u2e_fraglib
fragment_library_freq_cutoff 1
fragment_library_sort_method freq
fragment_library_trans_origin no
use_internal_energy no
ligand_atom_file up9_rec_withH.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd no
use_database_filter no
orient_ligand no
bump_filter no
score_molecules no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix 8u2e_fraglib_output
write_orientations no
num_scored_conformers 1
rank_ligands no
运行:
dock6 -i 8u2e.fraglib.in
产生与该片段文库相关的扭转环境文件,其名称为8u2e_fraglib_torenv.dat。
利用combine_torenv.py合并:
python ${DOCKHOME}/bin/combine_torenv.py 8u2e.fraglib_torenv.dat ${DOCKHOME}/parameters/fraglib_torenv.dat
生成一个新的列表:unique_full_sorted_fraglib. dat,这个文件记录了GA运行需要的Torsion Environments。
借助可合成性的原子环境(atom environments)的概念,DOCK使用Torsion Environments来表示分子等拓扑与键连接,并表示为一个正则字符串。用户配体分子的Torsion Environments可以与DOCK自带的合并,以提高遗传算法的成功率。可以使用自带的combine_torenv.py程序用于合并。
4.2 运行 GA
没有RDKit编译的情况
与4.1运行类似,产生一个空的输入文件 8u2e_GA.in:
```powershell
touch 8u2e_GA.in
运行:
dock6 -i 8u2e_GA.in
可以借鉴以下参数,完成交互式输入,完成后生成8u2e_GA.in文件。其中up9_rec_withH.mol2文件为,之前制备的配体输入文件。
conformer_search_type genetic
ga_molecule_file up9_rec_withH.mol2
ga_utilities no
ga_fraglib_scaffold_file $DOCKHOME/parameters/fraglib_ga_scaffold.mol2
ga_fraglib_linker_file $DOCKHOME/parameters/fraglib_linker.mol2
ga_fraglib_sidechain_file $DOCKHOME/parameters/fraglib_sidechain.mol2
ga_torenv_table unique_full_sorted_fraglib.dat
ga_max_generations 100
ga_xover_on yes
ga_xover_sampling_method_rand yes
ga_xover_max 150
ga_bond_tolerance 0.5
ga_angle_cutoff 0.14
ga_check_overlap no
ga_mutate_addition yes
ga_mutate_deletion yes../run_RF2.sh
ga_mutate_substitution yes
ga_mutate_replacement yes
ga_mutate_parents yes
ga_pmut_rate 0.3
ga_omut_rate 0.7
ga_max_mut_cycles 5
ga_mut_sampling_method rand
ga_num_random_picks 10
ga_max_root_size 5
ga_energy_cutoff 100
ga_heur_unmatched_num 2
ga_heur_matched_rmsd 2
ga_constraint_mol_wt 500
ga_constraint_rot_bon 10
ga_constraint_H_accept 10
ga_constraint_H_don 5
ga_constraint_formal_charge 4
ga_ensemble_size 200
ga_selection_method elitism
ga_elitism_combined no
ga_elitism_option max
ga_max_num_gen_with_no_crossover 1000
ga_name_identifier 8u2e.GA
ga_output_prefix 8u2e_GA_output
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100
use_database_filter no
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary no
multigrid_score_primary no
dock3.5_score_primary no
continuous_score_primary no
footprint_similarity_score_primary no
pharmacophore_score_primary no
hbond_score_primary no
internal_energy_score_primary no
descriptor_score_primary yes
descriptor_use_grid_score no
descriptor_use_multigrid_score no
descriptor_use_continuous_score no
descriptor_use_footprint_similarity no
descriptor_use_pharmacophore_score no
descriptor_use_tanimoto no
descriptor_use_hungarian no
descriptor_use_volume_overlap no
minimize_ligand yes
minimize_anchor yes
minimize_flexible_growth yes
use_advanced_simplex_parameters no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1
simplex_trans_step 1
simplex_rot_step 0.1
simplex_tors_step 10
simplex_anchor_max_iterations 200
simplex_grow_max_iterations 200
simplex_grow_tors_premin_iterations 00
simplex_random_seed 0
simplex_restraint_min yes
simplex_coefficient_restraint 10
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
运行:
dock6 -i 8u2e_GA.in -o 8u2e_GA.out
RDKit编译的情况
如果使用RDKit编译,可以在8u2e_GA.in加入以下参数:
sa_fraglib_path $DOCKHOME/parameters/sa_fraglib.dat
PAINS_path $DOCKHOME/parameters/pains_table_2019_09_01.dat
运行:
dock6.rdkit -i 8u2e_GA.in -o 8u2e_GA.out
结果文件中会增加RDKit描述符的数值。
在服务器上运行
创建slurm运行脚本:8u2e_GA.sh
输入以下内容:
#!/bin/bash
#SBATCH --time=48:00:00
#SBATCH --nodes=1
#SBATCH --ntasks=12
#SBATCH --job-name=8u2e_GA_input
#SBATCH --output=8u2e_GA_output
dock6.mpi -i 8u2e_GA.in -o 8u2e_GA.out
#dock6.rdkit.mpi -i 8u2e_GA.in -o 8u2e_GA.out
运行:
sbatch 8u2e_GA.sh
运行~ 2 h。结果文件,保留了每代产生的分子。
*restart0000.mol2文件是DOCK_GA采样的初始文件;
8u2e_GA_output.restart0100.mol2,即为第100代产生的分子,包含有2400个新结构的分子。
可以通过ViewDock查看;可以看到生成了一系列与固有配体具有类似空间分布的分子结构。
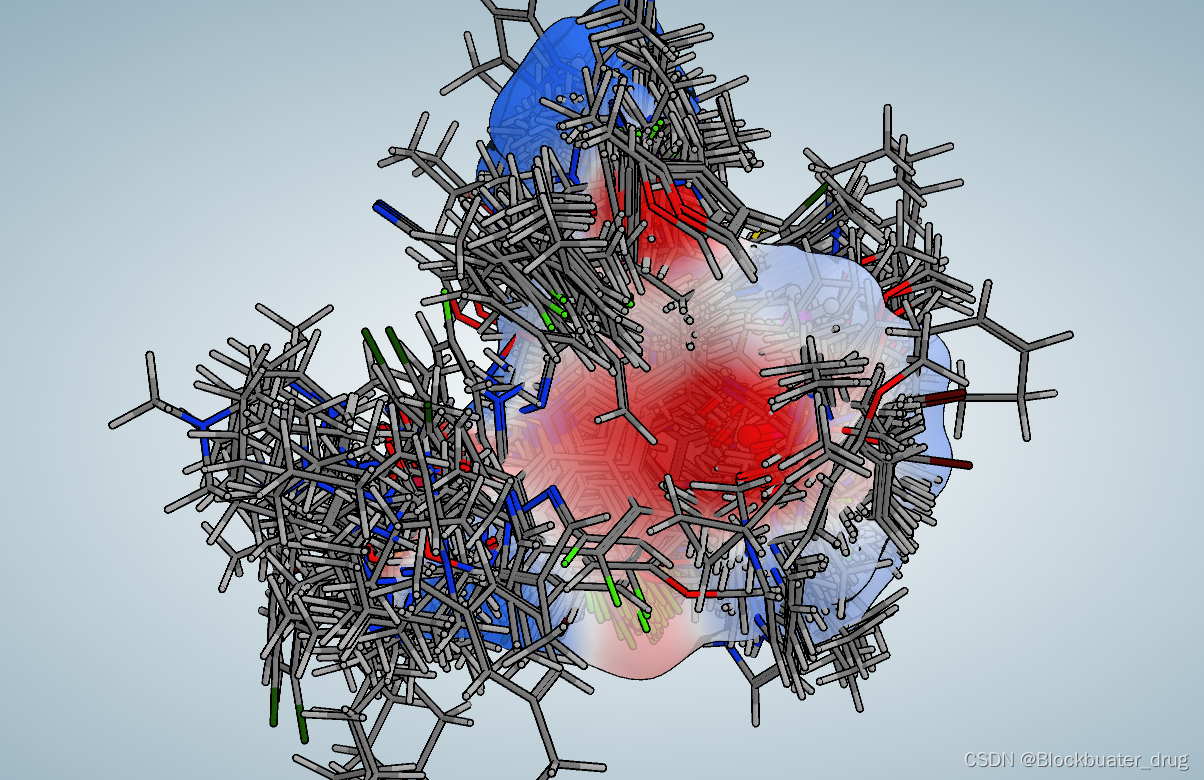 在8u2e_GA.in参数文件中,ga_max_generations控制遗传的代数;
在8u2e_GA.in参数文件中,ga_max_generations控制遗传的代数;
simplex_random_seed可以将某一代的生成分子作为起始分子, 比如用第100代分子做起始,可将其设置为100。
以上生成的分子可以采用rigid,flex等方法重新打分,合理的打分结果可用来排序,最终用于分子挑选。
总结
本文是UCSF DOCK的使用案例,关于DOCK中DOCK_GA模块,基于配体-靶标相互作用的遗传算法用于分子从头设计。
参考资料
- https://ringo.ams.stonybrook.edu/index.php/Main_Page
- https://dock.compbio.ucsf.edu/DOCK_6/dock6_manual.htm
- https://pubs.acs.org/doi/pdf/10.1021/acs.jcim.3c01031
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|