202107Using drug descriptions and molecular structures for drug-drug interaction extraction from literature
使用药物描述和分子结构从文献中提取药物-药物相互作用
Bioinformatics. 2021.07
有代码 https://github.com/tticoin/DESC_MOL-DDIE
目录
202107Using drug descriptions and molecular structures for drug-drug interaction extraction from literature
概述
研究背景
实验
输入句子表示
药物描述表示
分子结构表示
Ensemble
实验
概述
我们获得了以下结果。首先,大规模原始文本信息与现有模型相结合时可以大大提高提取DDI的性能,并显示出最先进的性能。其次,每种药物描述和分子结构信息都有助于进一步提高某些特定DDI类型的DDI性能。最后,同时使用药物描述和分子结构信息可以显著提高所有DDI类型的性能。结果表明,纯文本、药物描述信息和分子结构信息是互补的,它们的有效组合对于改进至关重要。
研究背景
本文是我们在ACL 2018(Asada等人,2018)中工作的重要扩展,有以下扩展:
-
将 word2vec 的标记表示替换为 SciBERT 获得的上下文化向量。因此,我们以最先进的性能显著提高了基线的性能。
-
采用了神经分子GNN(Tsubaki等人,2019),它考虑了相对较大的原子片段并更好地代表分子结构。
-
我们使用了在药物数据库中注册的药物描述,并且我们表明药物描述信息对于从某些DDI类型的语料库中提取DDI很有用。
-
研究发现,大规模训练前信息、药物描述和药物分子信息是互补的,它们的有效组合可以在很大程度上提高DDI提取性能。
实验
模型接收带有目标药物对的输入句子,并将该对分类为特定的DDI类型。使用SciBERT(Beltagy等人,2019)丰富了输入句子,这是一个在大规模生物医学和计算机科学文本上训练的BERT模型。
使用SciBERT获得靶药物的药物描述表示,并使用Tsubaki等人提出的分子图神经网络(GNN)模型获得靶药物的分子结构表示。
将这些药物描述和分子结构表示与丰富的输入句子表示相结合,并将目标药物对分类为特定的 DDI 类型。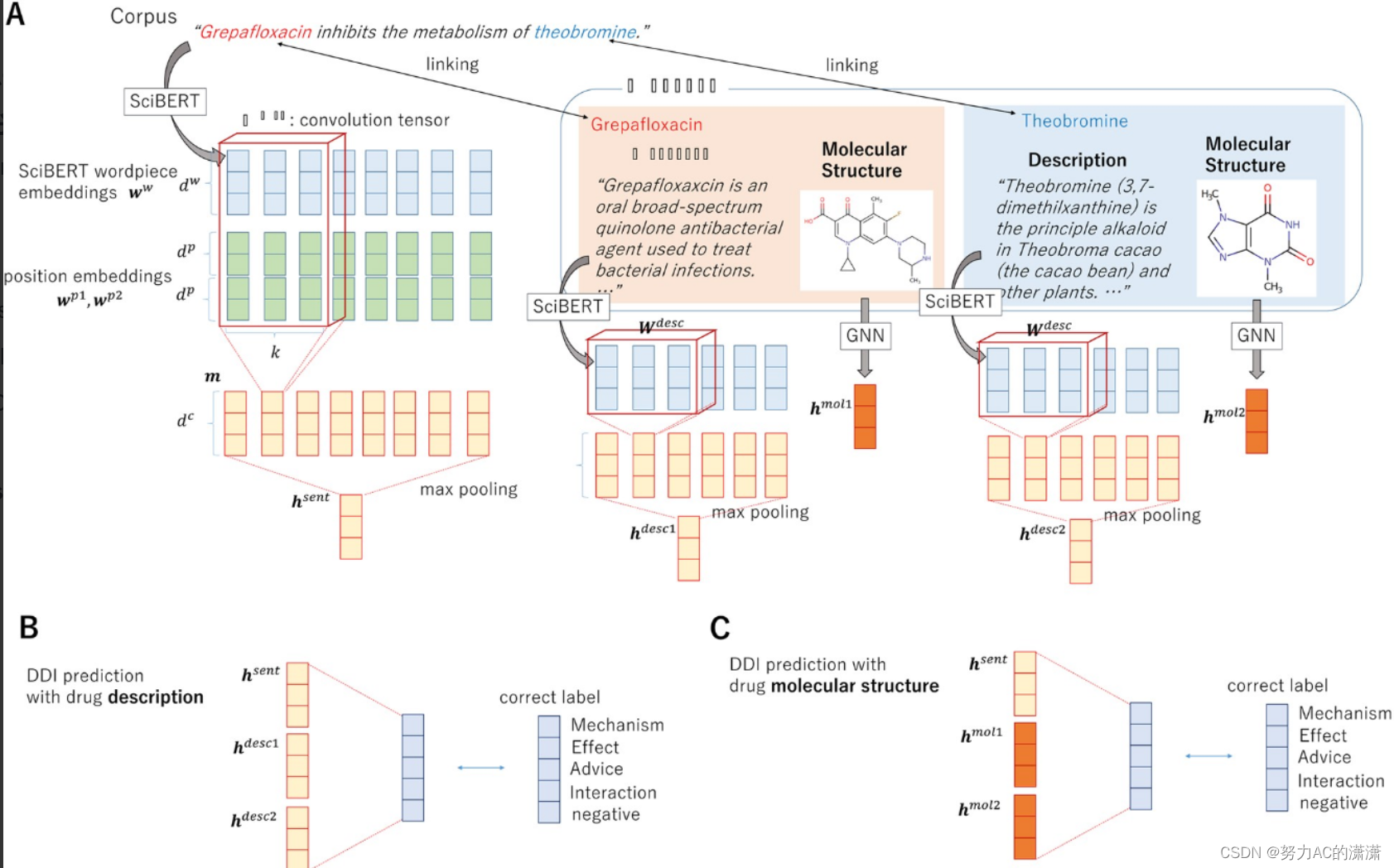
(A)说明如何编码输入句子,药物描述和药物分子结构。(B)和(C)显示使用药物描述表示和药物分子结构表示时的预测层
输入句子表示
图1A的左侧:给定一个输入句子S=(w1,⋯,wn)以及药物m1和m2,首先通过WordPiece算法将句子拆分为单词。然后,通过BERT模型(图1A中的浅蓝色向量)将每个单词wi转换为实值预训练的上下文嵌入;我们还准备d(p)维位置嵌入wp1和wp2,对于每个词片,分别对应于第一个和第二个目标提及的相对位置(图 1A 中的绿色向量)。我们连接词片嵌入和位置嵌入:
药物描述表示
与输入句子类似,药物的描述句子由BERT和CNN转换为实值固定大小向量。我们直接使用 BERT 的词片嵌入,无需词位置嵌入来准备卷积层的输入.![]()
卷积和最大池化与输入句子的处理方式相同
分子结构表示
在图中将原子表示为节点,将键表示为边缘。采用了GNN的方法。GNN使用相对较大的片段(称为r半径子图或分子指纹)来表示原子及其在图中的上下文。分子GNN采用指纹向量作为原子向量,随机初始化向量并考虑分子的图结构对其进行更新.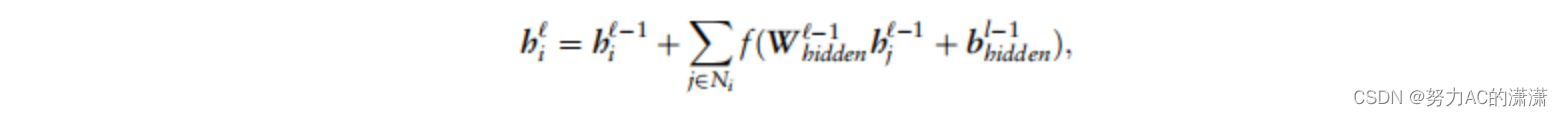
药物分子载体是通过将所有原子载体相加而获得的,然后将所得载体送入线性层。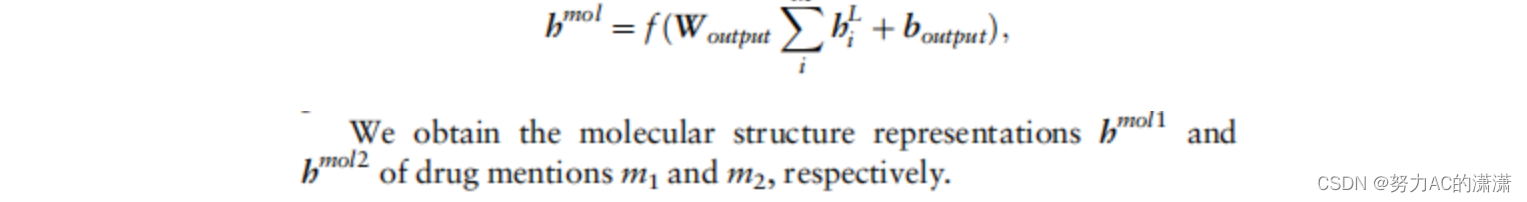
进行DDI提取
Ensemble
采用集成技术来组合来自不同模型的预测。具体来说,我们只是在分别训练每个模型后,将不同模型的预测分数汇总为融合。例如,当我们将模型的预测与描述信息以及分子结构信息相结合时,预测分数汇总如下: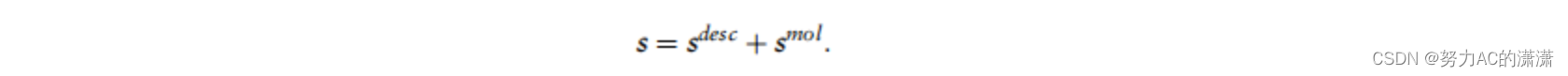
实验
数据集
数据集由两部分组成:MEDLINE和DrugBank。MEDLINE由MEDLINE/PubMed文章中的摘要组成,而DrugBank由DrugBank的FDA标签参考中的药物相互作用文本组成。
实验结果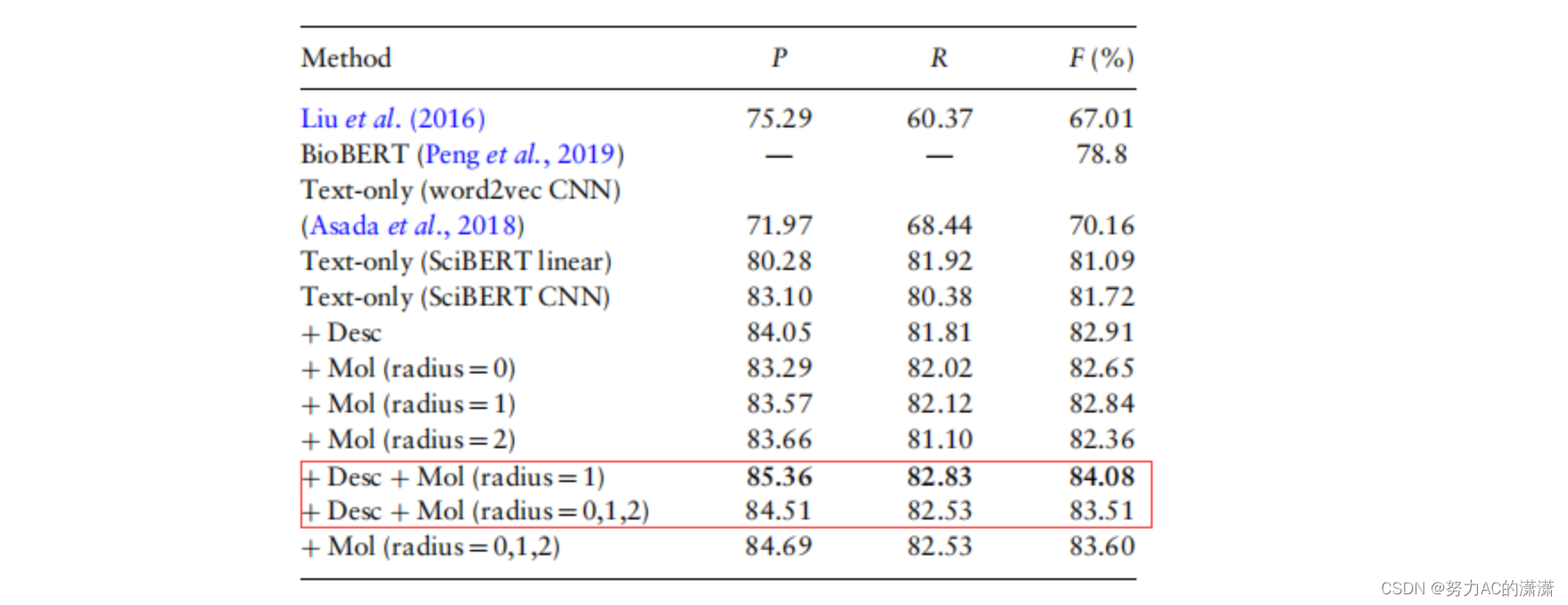
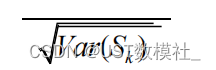





![Java中main函数里的String[] args详解](https://img-blog.csdnimg.cn/5fd07eeb27da4ee3aa75504f9af449a6.png)