目录
- 一、单个PET图像预处理
- 1、使用[MRIConvert](https://pan.baidu.com/s/1cn3kgeVRir8HvP6HHm0M0Q?pwd=5rt5)处理DCM
- 2、MRI和PET数据预处理过程
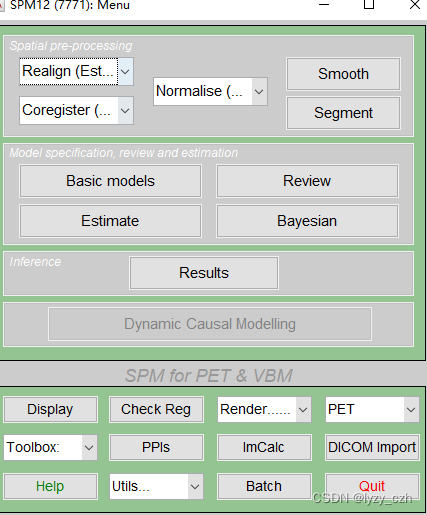
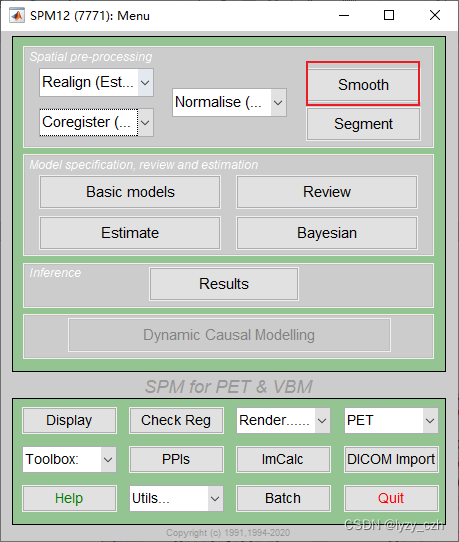
- 1) 打开matlab命令行输入spm pet,打开SMP12,界面如下
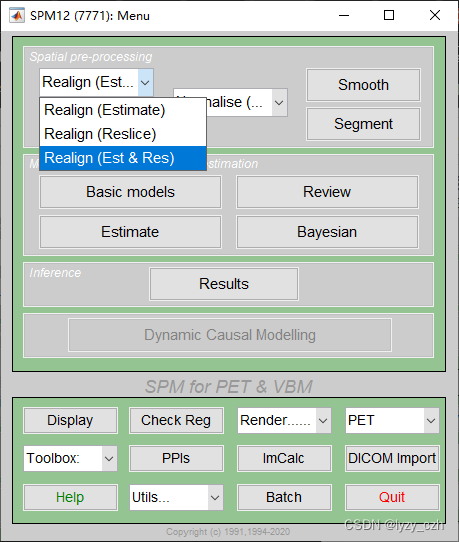
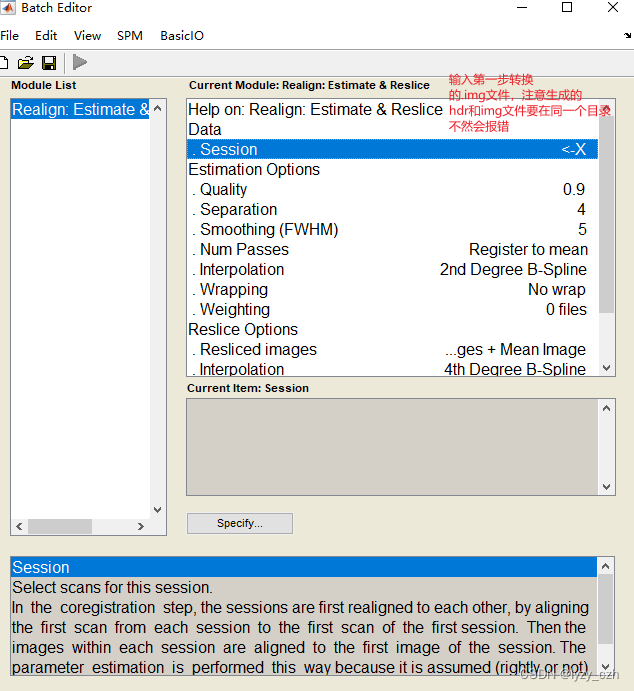
- 2) Realign,只需要编辑Session,其他参数不用改,编辑好后点击左上角绿色三角形按钮:
- 3)将mean002_s_0729的hdr数据配到PET模板上,点击Normalise(Est & Wri)
- 4)将wmeanxxxx.hdr配准到自己的MRI图像wmxxxx.nii上,打开Coregister(Est & Res),只修改Reference Image和Source Image;
- 5)去头颅
- 6)最后一步进行8mm的平滑
- 二、批量预处理
- 1、点击Batch
- 2、转到工作路径
- 3、选择预处理步骤
- 4、生成batch脚本
- 5、修改脚本代码
最近在做PET图像的预处理,相对于SMRI,PET的预处理要更加复杂,PET的预处理需要一步一步进行,每一步都需要前一步的输出结果,因此在批量预处理上也很麻烦。不过在和实验室小伙伴的共同努力下最终是完成了PET批量预处理。
自己在网上搜索教程的时候也没有找到很符合自己需要的教程,因此,把该问题的解决方法记录下来,供大家一起参考,如果觉得有用的话麻烦点个赞谢谢。
注:本方法使用的数据是从ADNI上下载的18F-FDG图像,下载的格式是DCM格式(PET图像会有其他格式的图像,我只使用了DCM格式的图像)环境使用了MATLABR2021a SPM12
一、单个PET图像预处理
为了更好理解批量预处理,有必要了解单个PET图像是如何预处理的。只想看批量预处理的直接跳到批量预处理即可
PET预处理整个流程如下:使用MRIConvert软件将DCM格式转为hdr格式→Realign→Normalise→Coregister→ImCalc→Smooth
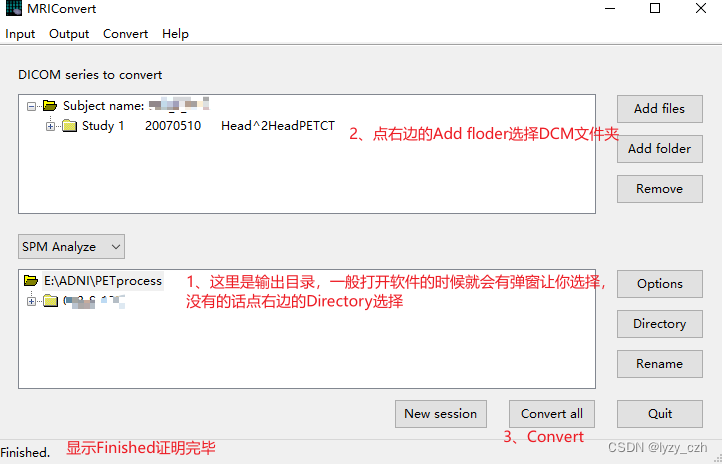
1、使用MRIConvert处理DCM
打开程序选择输出输入目录并且选择SPM Analyze如下图所示
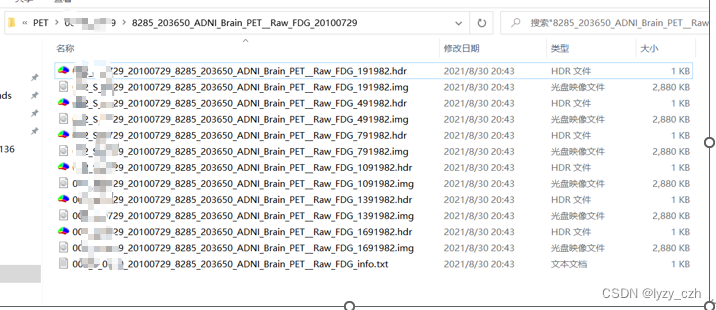
转换完成后输出如下:
这里生成了多个hdr/img的图像,只需要选择其中最好的一个即可(我看起来都是一样的)
使用MRIcron打开hdr文件如下:MRIcron在我的之前的文章有介绍

2、MRI和PET数据预处理过程
1) 打开matlab命令行输入spm pet,打开SMP12,界面如下
2) Realign,只需要编辑Session,其他参数不用改,编辑好后点击左上角绿色三角形按钮:
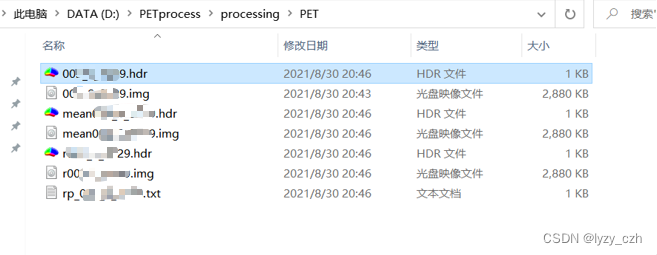
此时在输出目录下生成如下文件:
3)将mean002_s_0729的hdr数据配到PET模板上,点击Normalise(Est & Wri)
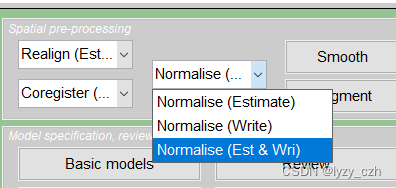
弹出以下窗口,只需编辑Source Image, Images to Write和Template Image;其他参数不动,最后点击左上角蓝色三角形运行
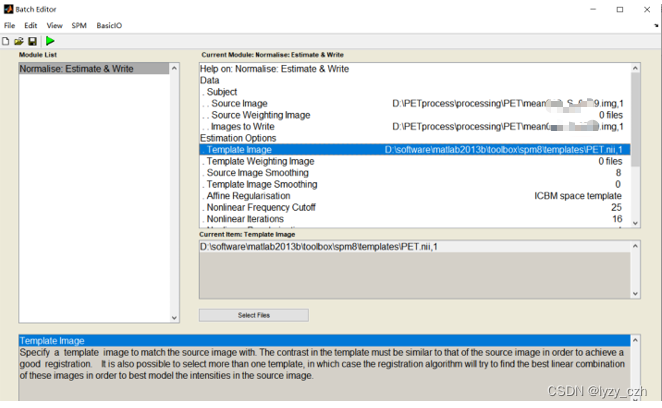
生成如下文件:
点开wmeanxxxx.hdr如下:
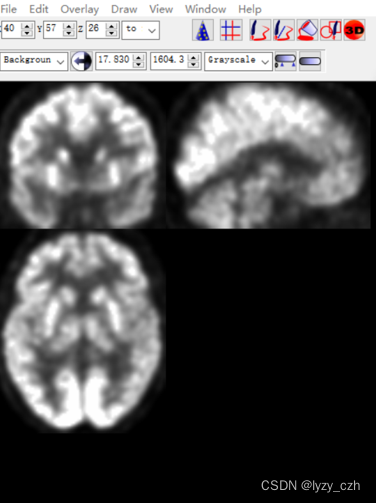
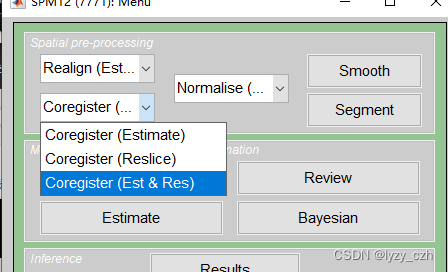
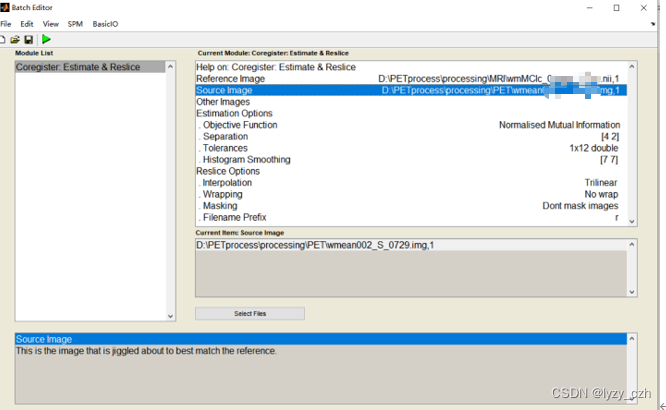
4)将wmeanxxxx.hdr配准到自己的MRI图像wmxxxx.nii上,打开Coregister(Est & Res),只修改Reference Image和Source Image;
注意:如果只需要处理PET图像,MRI可以使用模板来进行配准和去头骨影像(这里是配准到同一患者的SMRI预处理后的图像 wmxxxx.nii是SMRI预处理后的图像)
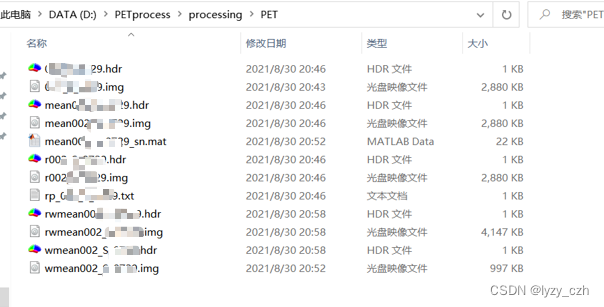
生成如下文件:
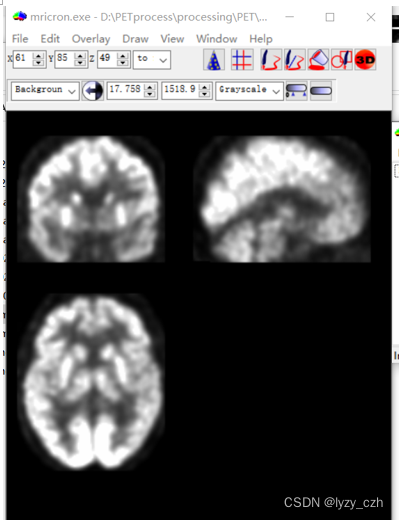
此时的PET与MRI大小相同
5)去头颅
点击ImCalc
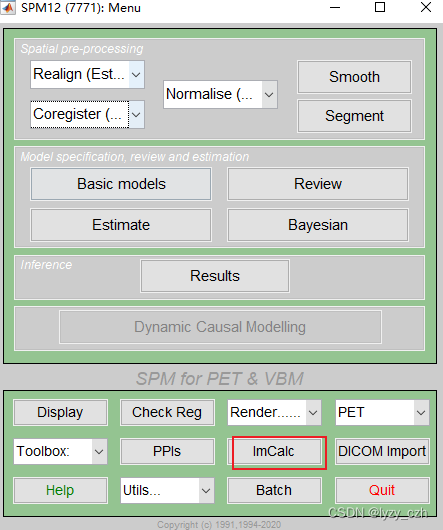
弹出如下界面,Input Images先选择MRI的图像,即wmxxxx.nii(也可以选MRI模板),再选rwmeanxxxx.img,Expression输入i2.*(i1>0.05),其中i1表示第一张图像,i2表示第二张,运行后会在matlab的当前路径输出结果,我这里是PETprocess\processing,所以在下面会生成output图像。
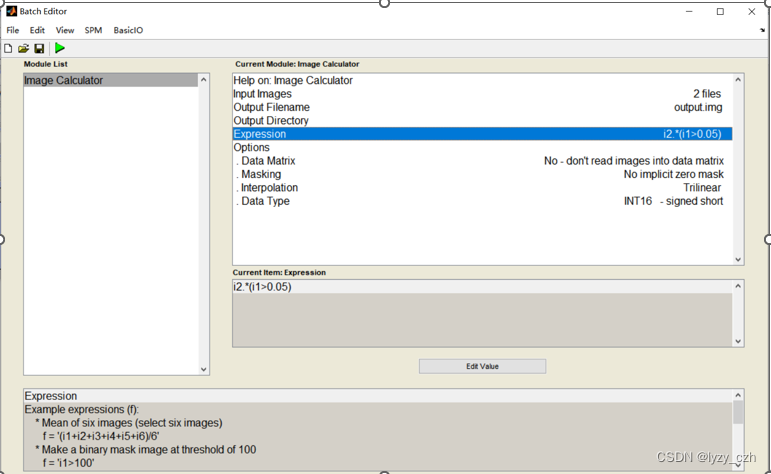
打开output图像如下:
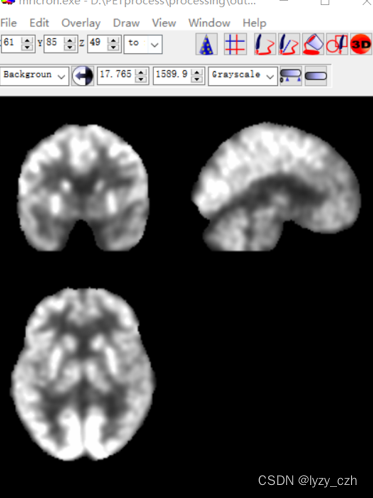
6)最后一步进行8mm的平滑
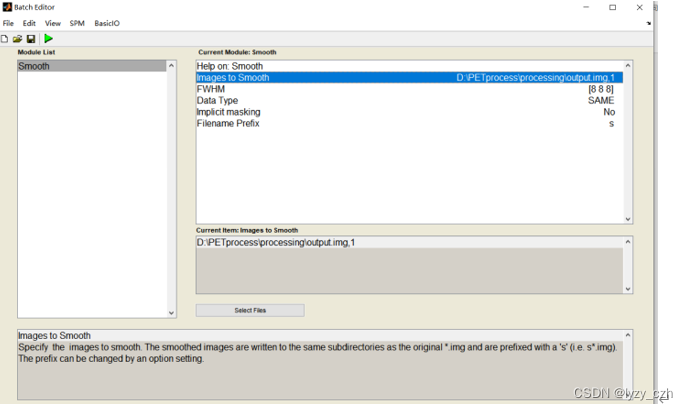
最后得到的图像如下:
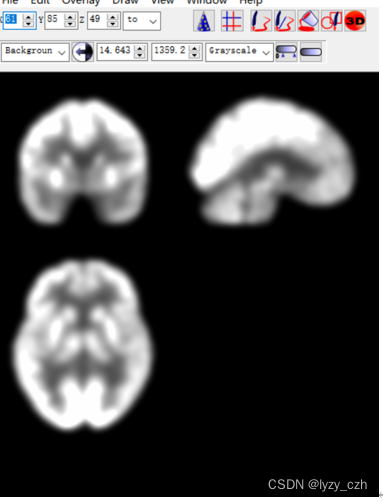
这便是PET预处理后的图像,到此整个PET预处理流程已经走完,做完后发现这也太复杂了,如果要处理几百张图像人估计都没了。。。所以接下来的才是重头戏,批量预处理。
二、批量预处理
我这里的方法是使用SPM12自带的batch工具选择需要的步骤之后生成脚本文件,然后根据实际情况修改脚本完成批量预处理!
1、点击Batch
2、转到工作路径

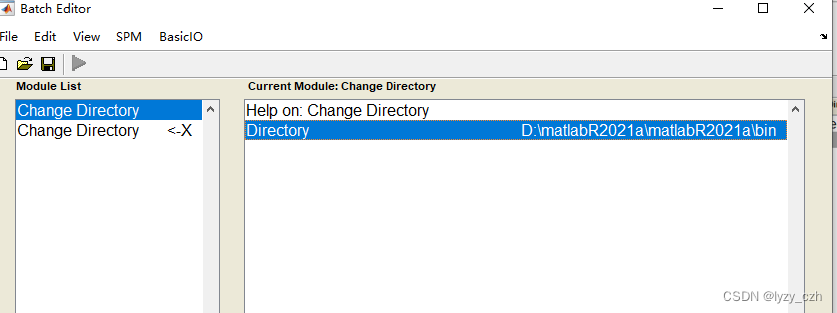
3、选择预处理步骤
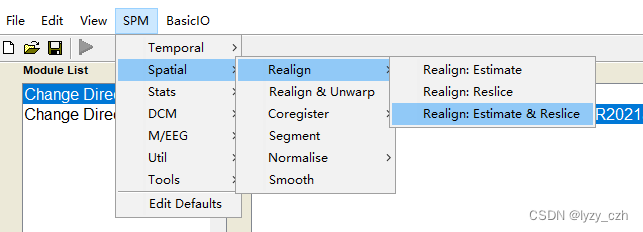
选择后要点一下要输入数据的地方不然可能会报错,如下图双击Data然后回多出一行Session这样就不会有问题了
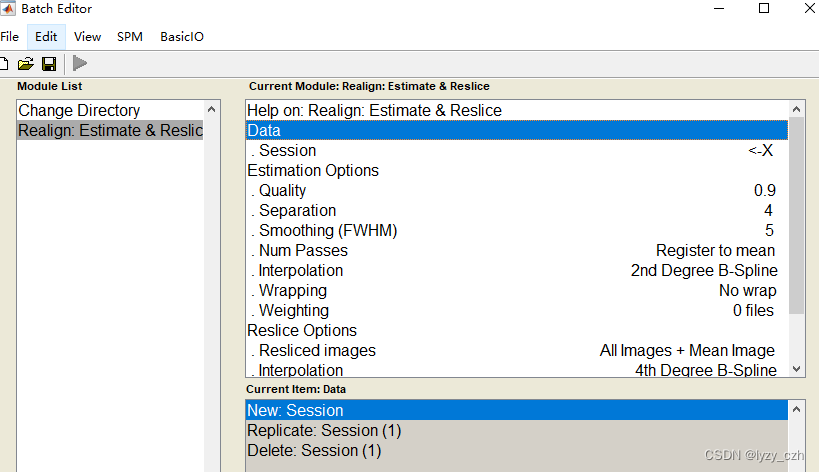
依次选择所有步骤如下一共5个步骤:
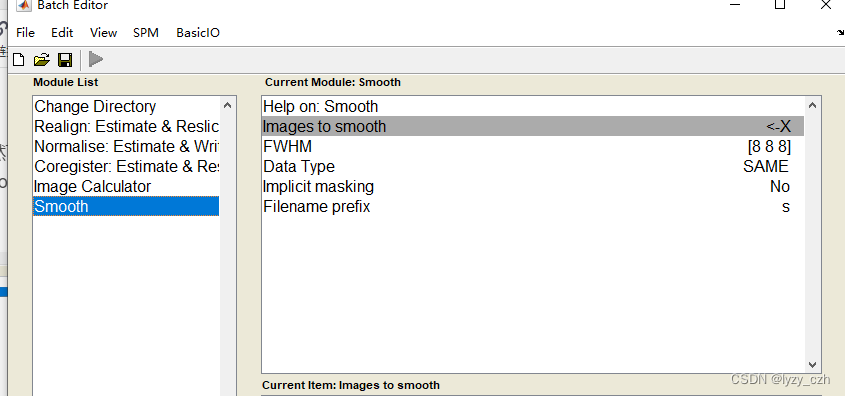
注意在Image Calculator的Expression输入是固定的因此可以直接在这里输入如下:
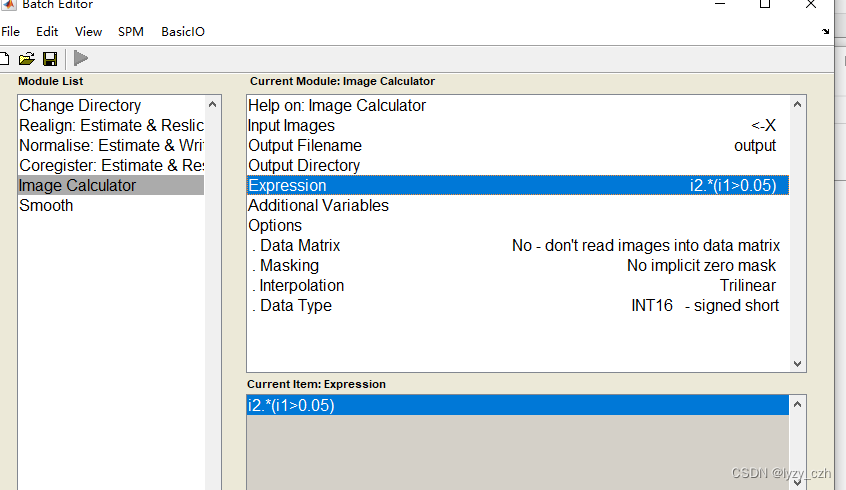
4、生成batch脚本
选择保存脚本的路径
点击保存后,会生成两个.m文件并且自动在matlab打开脚本文件如下:
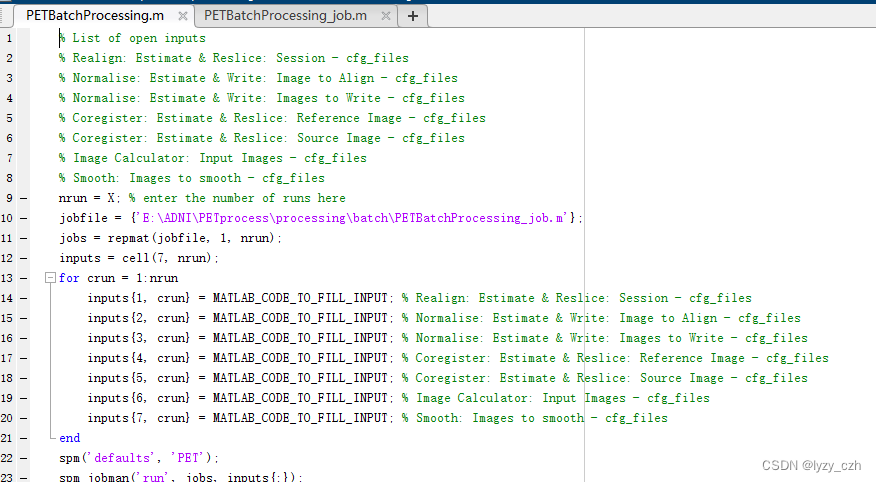
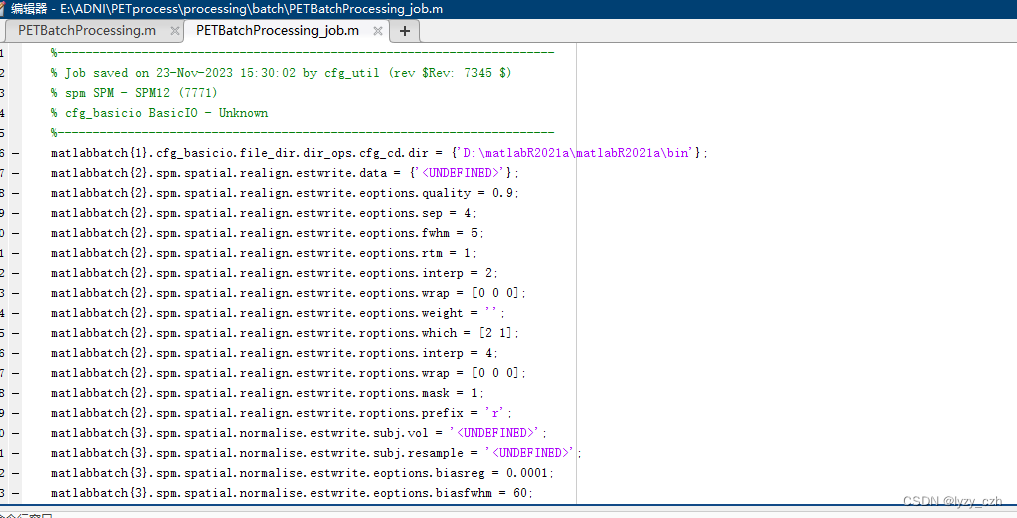
这里生成的只是模板文件,图像的输入输出还有一些细节是需要我们手动修改实现的。
5、修改脚本代码
1、xxx.m文件代码如下(xxx是你自己的命名)
% List of open inputs
% Realign: Estimate & Reslice: Session - cfg_files
% Normalise: Estimate & Write: Image to Align - cfg_files
% Normalise: Estimate & Write: Images to Write - cfg_files
% Coregister: Estimate & Reslice: Reference Image - cfg_files
% Coregister: Estimate & Reslice: Source Image - cfg_files
% Image Calculator: Input Images - cfg_files
% Smooth: Images to smooth - cfg_files
% 设置MRI文件夹路径
mri_folder = 'E:\ADNI\PETprocess\processing\batch\SMRI';
% 设置PET文件夹路径和待处理的图像数量
pet_folder = 'E:\ADNI\PETprocess\processing\batch\PET';
% 读取PET文件夹中的图像文件名列表
pet_files = dir(fullfile(pet_folder, '*.img'));
numFiles = length(pet_files);
pet_filenames = {pet_files.name};
nrun = numFiles; % 请将X替换为实际的图像数量
jobfile = {'E:\ADNI\PETprocess\processing\xxx_job.m'}; %注意这里要对应
jobs = repmat(jobfile, 1, nrun);
% 循环处理每个PET图像
for crun = 1:nrun
output_path = 'E:\ADNI\PETprocess\processing\batch\output\AD'; % 数据结果的目录,job文件也要添加这个路径
subjectID = strrep(pet_filenames{crun}, '.img', ''); % 获取当前处理的图像文件名(不含扩展名)
% Realign: Estimate & Reslice
inputs{1, crun} = cellstr(fullfile(pet_folder, pet_filenames{crun})); % 输入待处理的PET图像
% Normalise: Estimate & Write
inputs{2, crun} = cellstr(fullfile(pet_folder, ['mean', subjectID, '.img'])); % 输入mean图像
inputs{3, crun} = cellstr(fullfile(pet_folder, ['mean', subjectID, '.img'])); % 输入mean图像
% Coregister: Estimate & Reslice
inputs{4, crun} = cellstr(fullfile(mri_folder, [subjectID, '.nii'])); % MRI参考图像
inputs{5, crun} = cellstr(fullfile(pet_folder, ['wmean', subjectID, '.img'])); % 输入rwmean图像
% Image Calculator
i1 = fullfile(mri_folder, [subjectID, '.nii']);
i2 = fullfile(pet_folder, ['rwmean' subjectID, '.img']);
inputs{6, crun} = [cellstr(i1); cellstr(i2)]; % 将i1和i2组合成一个cell数组,然后赋值给inputs{6, crun}
inputs{7, crun} = [subjectID, '.nii'];
% Smooth
inputs{8, crun} = cellstr(fullfile(output_path, [subjectID, '.nii'])); % 输入文件为subjectID.nii图像
end
% 加载SPM默认设置和运行处理任务
spm('defaults', 'PET');
spm_jobman('run', jobs, inputs{:});
2、xxx_job.m文件代码如下(xxx是你自己的命名)
%-----------------------------------------------------------------------
% Job saved on 20-Nov-2023 19:01:02 by cfg_util (rev $Rev: 7345 $)
% spm SPM - SPM12 (7771)
% cfg_basicio BasicIO - Unknown
%-----------------------------------------------------------------------
matlabbatch{1}.cfg_basicio.file_dir.dir_ops.cfg_cd.dir = {'D:\matlabR2021a\matlabR2021a\bin'};
matlabbatch{2}.spm.spatial.realign.estwrite.data = {'<UNDEFINED>'};
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.quality = 0.9;
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.sep = 4;
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.fwhm = 5;
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.rtm = 1;
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.interp = 2;
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.wrap = [0 0 0];
matlabbatch{2}.spm.spatial.realign.estwrite.eoptions.weight = '';
matlabbatch{2}.spm.spatial.realign.estwrite.roptions.which = [2 1];
matlabbatch{2}.spm.spatial.realign.estwrite.roptions.interp = 4;
matlabbatch{2}.spm.spatial.realign.estwrite.roptions.wrap = [0 0 0];
matlabbatch{2}.spm.spatial.realign.estwrite.roptions.mask = 1;
matlabbatch{2}.spm.spatial.realign.estwrite.roptions.prefix = 'r';
matlabbatch{3}.spm.spatial.normalise.estwrite.subj.vol = '<UNDEFINED>';
matlabbatch{3}.spm.spatial.normalise.estwrite.subj.resample = '<UNDEFINED>';
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.biasreg = 0.0001;
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.biasfwhm = 60;
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.tpm = {'D:\matlabR2021a\matlabR2021a\toolbox\spm12\spm12\tpm\TPM.nii'};
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.affreg = 'mni';
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.reg = [0 0.001 0.5 0.05 0.2];
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.fwhm = 0;
matlabbatch{3}.spm.spatial.normalise.estwrite.eoptions.samp = 3;
matlabbatch{3}.spm.spatial.normalise.estwrite.woptions.bb = [-78 -112 -70
78 76 85];
matlabbatch{3}.spm.spatial.normalise.estwrite.woptions.vox = [2 2 2];
matlabbatch{3}.spm.spatial.normalise.estwrite.woptions.interp = 4;
matlabbatch{3}.spm.spatial.normalise.estwrite.woptions.prefix = 'w';
matlabbatch{4}.spm.spatial.coreg.estwrite.ref = '<UNDEFINED>';
matlabbatch{4}.spm.spatial.coreg.estwrite.source = '<UNDEFINED>';
matlabbatch{4}.spm.spatial.coreg.estwrite.other = {''};
matlabbatch{4}.spm.spatial.coreg.estwrite.eoptions.cost_fun = 'nmi';
matlabbatch{4}.spm.spatial.coreg.estwrite.eoptions.sep = [4 2];
matlabbatch{4}.spm.spatial.coreg.estwrite.eoptions.tol = [0.02 0.02 0.02 0.001 0.001 0.001 0.01 0.01 0.01 0.001 0.001 0.001];
matlabbatch{4}.spm.spatial.coreg.estwrite.eoptions.fwhm = [7 7];
matlabbatch{4}.spm.spatial.coreg.estwrite.roptions.interp = 4;
matlabbatch{4}.spm.spatial.coreg.estwrite.roptions.wrap = [0 0 0];
matlabbatch{4}.spm.spatial.coreg.estwrite.roptions.mask = 0;
matlabbatch{4}.spm.spatial.coreg.estwrite.roptions.prefix = 'r';
matlabbatch{5}.spm.util.imcalc.input = '<UNDEFINED>';
matlabbatch{5}.spm.util.imcalc.output = '<UNDEFINED>'; %这里要改
matlabbatch{5}.spm.util.imcalc.outdir = {'E:\ADNI\PETprocess\processing\batch\output\AD'}; %改成你自己的目录
matlabbatch{5}.spm.util.imcalc.expression = 'i2.*(i1>0.05)';
matlabbatch{5}.spm.util.imcalc.var = struct('name', {}, 'value', {});
matlabbatch{5}.spm.util.imcalc.options.dmtx = 0;
matlabbatch{5}.spm.util.imcalc.options.mask = 0;
matlabbatch{5}.spm.util.imcalc.options.interp = 1;
matlabbatch{5}.spm.util.imcalc.options.dtype = 4;
matlabbatch{6}.spm.spatial.smooth.data = '<UNDEFINED>';
matlabbatch{6}.spm.spatial.smooth.fwhm = [8 8 8];
matlabbatch{6}.spm.spatial.smooth.dtype = 0;
matlabbatch{6}.spm.spatial.smooth.im = 0;
matlabbatch{6}.spm.spatial.smooth.prefix = 's';
修完成后运行xxx.m即可
注:job.m大部分代码是自动生成的,如果你完全按照我的步骤运行代码后报错如下:
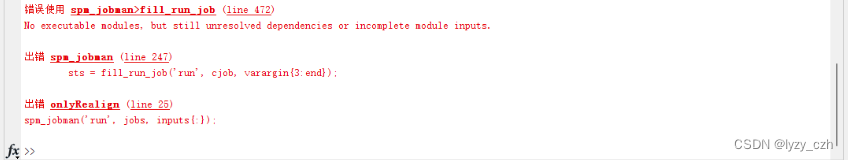
这大概率是job文件的错误,请仔细对照job文件是不是该有<UNDEFINED>地方没有<UNDEFINED>,上面步骤说要点击一下输入文件的地方就是为了防止这里不生成<UNDEFINED>。
OVER(点赞)!