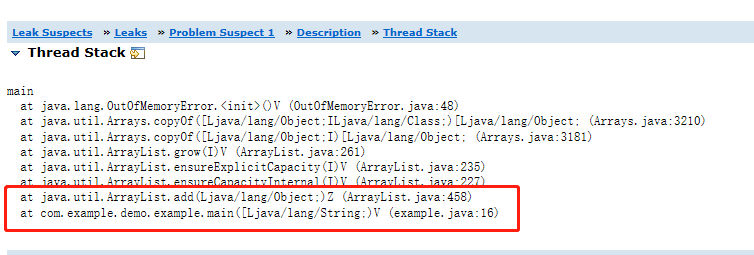
🌈个人主页:Sarapines Programmer
🔥 系列专栏:《人工智能奇遇记》
🔖少年有梦不应止于心动,更要付诸行动。
目录结构
1. 机器学习之聚类算法概念
1.1 机器学习
1.2 聚类算法
2. 聚类算法
2.1 实验目的
2.2 实验准备
2.3 实验原理
2.4 实验内容
2.4.1 K-means算法
2.4.2 K-mean++算法
2.4.3 K_medoids算法
2.4.4 DBScan算法
2.5 实验心得
致读者
1. 机器学习之聚类算法概念
1.1 机器学习
传统编程要求开发者明晰规定计算机执行任务的逻辑和条条框框的规则。然而,在机器学习的魔法领域,我们向计算机系统灌输了海量数据,让它在数据的奔流中领悟模式与法则,自主演绎未来,不再需要手把手的指点迷津。
机器学习,犹如三千世界的奇幻之旅,分为监督学习、无监督学习和强化学习等多种类型,各具神奇魅力。监督学习如大师传道授业,算法接收标签的训练数据,探索输入与输出的神秘奥秘,以精准预测未知之境。无监督学习则是数据丛林的探险者,勇闯没有标签的领域,寻找隐藏在数据深处的秘密花园。强化学习则是一场与环境的心灵对话,智能体通过交互掌握决策之术,追求最大化的累积奖赏。
机器学习,如涓涓细流,渗透各行各业。在图像和语音识别、自然语言处理、医疗诊断、金融预测等领域,它在智慧的浪潮中焕发生机,将未来的可能性绘制得更加丰富多彩。
1.2 聚类算法
聚类算法是一类无监督学习的算法,其目标是将数据集中的样本划分为若干个互不重叠的子集,每个子集被称为一个"簇",使得同一簇内的样本相似度较高,而不同簇之间的样本相似度较低。聚类的目标是在不事先知道数据的真实类别标签的情况下,发现数据中的内在结构和模式。
以下是一些常见的聚类算法:
K均值聚类(K-Means): 是最经典和常用的聚类算法之一。它通过将数据划分为K个簇,并使每个样本点到其所属簇的中心距离最小化来实现。K-Means算法迭代更新簇的中心,直至达到收敛条件。
层次聚类(Hierarchical Clustering): 层次聚类通过构建一颗树状结构(聚类树或谱系树)来刻画样本之间的层次关系。可以是自底向上(凝聚性层次聚类)或自顶向下(分裂性层次聚类)的方法。
DBSCAN(Density-Based Spatial Clustering of Applications with Noise): 是一种基于样本密度的聚类算法。它通过寻找高密度区域,将数据划分为不同的簇,并可以识别噪声点。DBSCAN不需要预先指定簇的数量,适用于不规则形状的簇和对噪声相对鲁棒的场景。
谱聚类(Spectral Clustering): 利用样本之间的相似度矩阵,将其转化为拉普拉斯矩阵,通过对拉普拉斯矩阵进行特征分解,得到样本的特征向量,再通过K-Means等方法对特征向量进行聚类。谱聚类通常对图结构的数据有较好的适应性。
高斯混合模型聚类(Gaussian Mixture Model, GMM): 假设数据是由若干个高斯分布混合而成,通过迭代优化参数,最大化观测数据的似然函数,从而进行聚类。GMM对于数据分布呈现出复杂结构的情况较为有效。
这些聚类算法在不同场景和数据特性下有各自的优势和局限性,选择合适的算法取决于问题的性质和对结果的需求。聚类在图像分割、客户细分、异常检测等领域都有广泛的应用。
机器学习源文件![]() https://download.csdn.net/download/m0_57532432/88521177?spm=1001.2014.3001.5503
https://download.csdn.net/download/m0_57532432/88521177?spm=1001.2014.3001.5503
2. 聚类算法
2.1 实验目的
(1)加深对非监督学习的理解和认识;
(2)掌握基于距离的和基于密度的动态聚类算法的设计方法。
2.2 实验准备
(1)安装机器学习必要库,如NumPy、Pandas、Scikit-learn等;
(2)配置环境用来运行 Python、Jupyter Notebook和相关库等内容。
2.3 实验原理
-
非监督学习的基础: 非监督学习是机器学习的重要分支,旨在从未标记的数据中发现模式、结构或关联关系。与监督学习不同,非监督学习通过聚类、降维或关联规则挖掘等技术学习,不依赖于预先标记的数据。其理论基础包括聚类、降维和异常检测等方法,揭示数据内在结构和规律。
-
动态聚类分析: 针对数据流或时序数据的聚类方法,旨在在变化的数据中发现和适应数据的演化。能够处理概念漂移、新类别出现和老类别消失等问题。基于数据序列性质,将数据划分为连续时间窗口,通过更新聚类模型适应数据变化。其理论依据包括时间窗口模型、漂移检测算法和增量聚类算法等。
-
聚类算法评价指标: 用于衡量聚类结果质量和性能的评价指标包括:
-
内部评价指标: 关注聚类结果的紧密性和分离性,如轮廓系数、Davies-Bouldin指数和Calinski-Harabasz指数。
-
外部评价指标: 用于将聚类结果与已知标签或真实类别进行比较,评估聚类准确性,如准确率、召回率、F1分数和调整兰德指数。
-
相似性度量: 用于比较不同聚类结果之间的相似程度,包括Jaccard系数、兰德指数和互信息等。
-
2.4 实验内容
1.选择一种聚类算法对鸢尾花做聚类;
2.读入要分类的数据;
3.设置初始聚类中心;
4.根据不同的聚类算法实现聚类;
5.显示聚类结果;
6.按照同样步骤实现学过的所有聚类算法。
在本次实验中,我使用了以下五种聚类方法来对数据进行分析和分类。其中,凝聚聚类算法(Agglomerative Clustering)是我自学的一种聚类方法。
1.K-means
K-means将数据分成K个簇,每个簇都以一个质心代表。该算法通过迭代的方式不断调整簇的质心位置,使得样本点到所属簇的质心的距离最小化。
2.K-means++
K-means++在选择初始质心时更加智能化。K-means++首先选择一个初始质心作为第一个簇的质心,然后根据距离选择下一个质心,直到选择完所有的质心。
3.K_medoids
K_medoids使用样本点作为簇的中心。与K-means算法不同,K_medoids选择的中心点必须是实际存在的样本点,而不仅仅是质心的位置。
4.DBScan
DBScan它将具有足够高密度的样本点划分为一个簇,并将低密度区域视为噪声。DBScan通过设置邻域半径和最小样本数来定义簇的形成条件。
5.凝聚聚类算法
凝聚聚类算法从每个样本点开始,逐步将最近的样本点聚合成簇,直到满足预设的聚类数目。凝聚聚类算法的特点是簇的形成是通过合并的方式进行的。
2.4.1 K-means算法
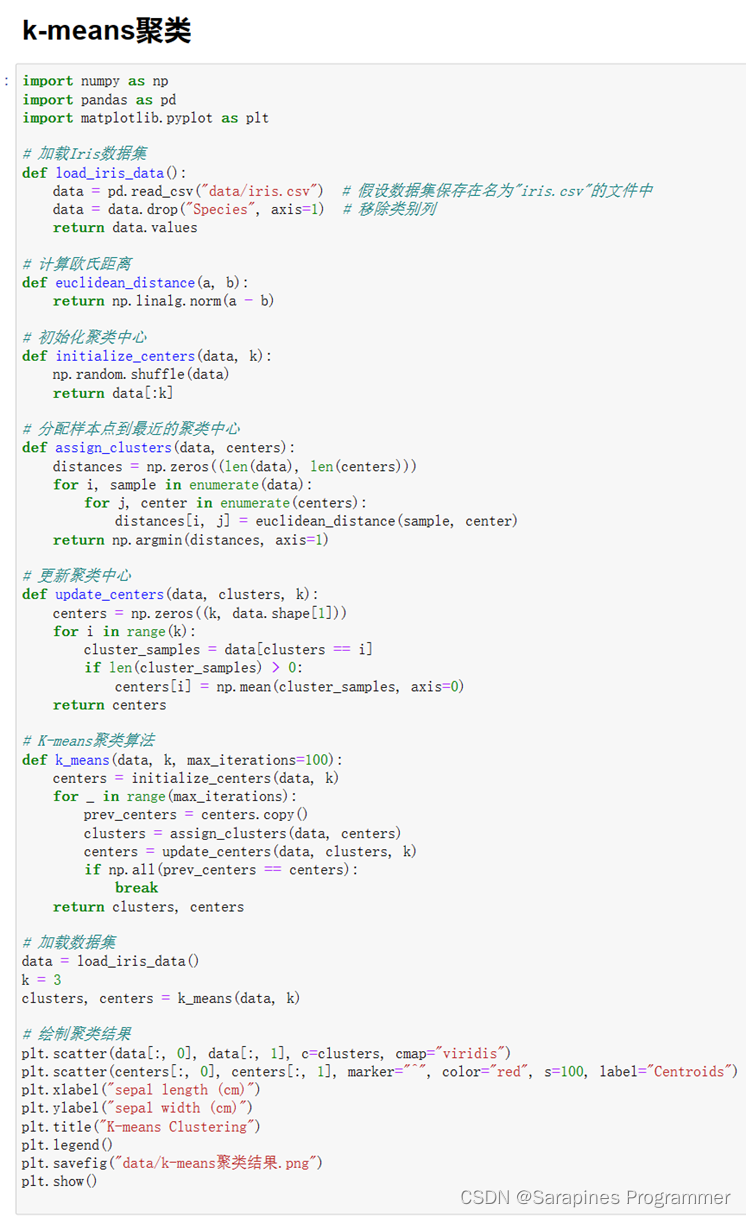
图2-1
运行结果

图2-2
实验代码:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 加载Iris数据集
def load_iris_data():
data = pd.read_csv("data/iris.csv") # 假设数据集保存在名为"iris.csv"的文件中
data = data.drop("Species", axis=1) # 移除类别列
return data.values
# 计算欧氏距离
def euclidean_distance(a, b):
return np.linalg.norm(a - b)
# 初始化聚类中心
def initialize_centers(data, k):
np.random.shuffle(data)
return data[:k]
# 分配样本点到最近的聚类中心
def assign_clusters(data, centers):
distances = np.zeros((len(data), len(centers)))
for i, sample in enumerate(data):
for j, center in enumerate(centers):
distances[i, j] = euclidean_distance(sample, center)
return np.argmin(distances, axis=1)
# 更新聚类中心
def update_centers(data, clusters, k):
centers = np.zeros((k, data.shape[1]))
for i in range(k):
cluster_samples = data[clusters == i]
if len(cluster_samples) > 0:
centers[i] = np.mean(cluster_samples, axis=0)
return centers
# K-means聚类算法
def k_means(data, k, max_iterations=100):
centers = initialize_centers(data, k)
for _ in range(max_iterations):
prev_centers = centers.copy()
clusters = assign_clusters(data, centers)
centers = update_centers(data, clusters, k)
if np.all(prev_centers == centers):
break
return clusters, centers
# 加载数据集
data = load_iris_data()
k = 3
clusters, centers = k_means(data, k)
# 绘制聚类结果
plt.scatter(data[:, 0], data[:, 1], c=clusters, cmap="viridis")
plt.scatter(centers[:, 0], centers[:, 1], marker="^", color="red", s=100, label="Centroids")
plt.xlabel("sepal length (cm)")
plt.ylabel("sepal width (cm)")
plt.title("K-means Clustering")
plt.legend()
plt.savefig("data/k-means聚类结果.png")
plt.show()
源码分析
该代码实现了K-means聚类算法对Iris数据集进行聚类,其中:
- load_iris_data()函数用于加载Iris数据集,假设数据集保存在名为"iris.csv"的文件中,并移除了类别列。
- euclidean_distance()函数计算两个样本点之间的欧氏距离。
- initialize_centers()函数用于初始化聚类中心,随机从数据中选择k个样本作为初始聚类中心。
- assign_clusters()函数将样本点分配到最近的聚类中心,计算每个样本点与所有聚类中心的距离,返回每个样本点所属的聚类索引。
- update_centers()函数更新聚类中心,计算每个聚类的样本点的均值,并将其作为新的聚类中心。
- k_means()函数是K means聚类算法的实现。它接收数据集、聚类数目k和最大迭代次数作为参数,并返回聚类结果和最终的聚类中心。
在k_means()函数中,首先使用initialize_centers()函数初始化聚类中心,然后进入迭代过程。每次迭代,首先将当前的聚类中心保存为prev_centers,然后使用assign_clusters()函数将样本点分配到最近的聚类中心,得到每个样本点所属的聚类索引。接下来,使用update_centers()函数根据每个聚类的样本点计算新的聚类中心。在每次迭代之后,检查当前的聚类中心是否与上一次迭代的聚类中心相同,如果相同,则说明聚类已经收敛,可以提前结束迭代。最终,返回聚类结果和最终的聚类中心。
2.4.2 K-mean++算法

图2-3
运行结果
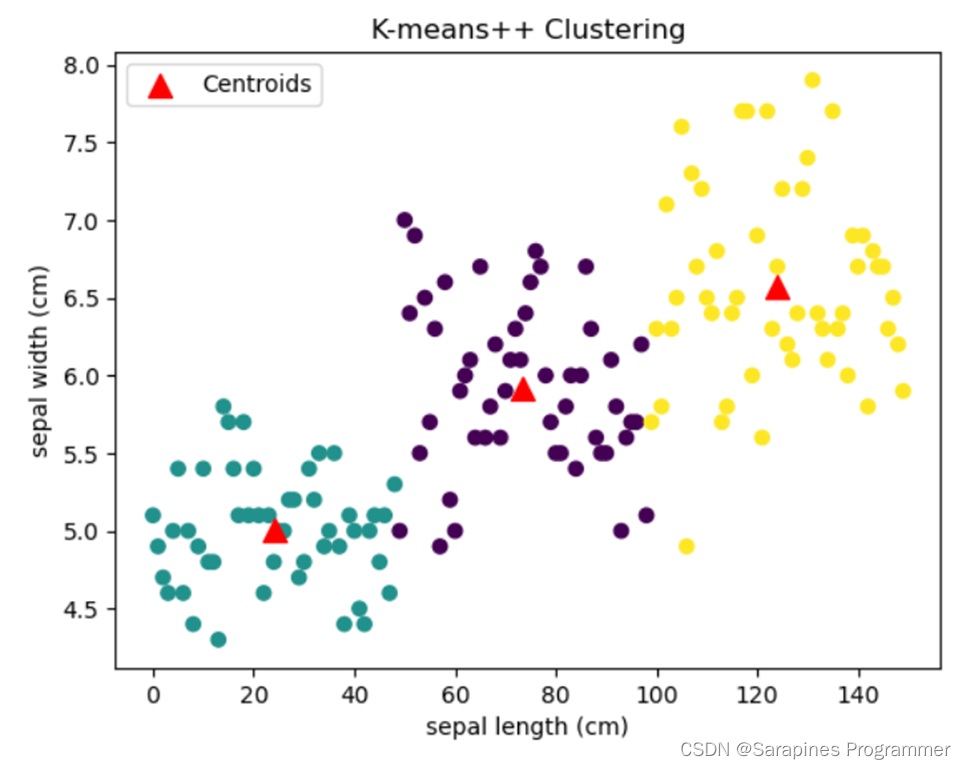
图2-4
实验代码:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 加载Iris数据集
def load_iris_data():
data = pd.read_csv("data/iris.csv")
# 假设数据集保存在名为"iris.csv"的文件中
data = data.drop("Species", axis=1)
# 移除类别列
return data.values
# 计算欧氏距离
def euclidean_distance(a, b):
return np.linalg.norm(a - b)
# 初始化聚类中心
def initialize_centers(data, k):
centers = np.zeros((k, data.shape[1]))
# 创建一个k行,每行包含数据的特征数列的零数组,用于存储聚类中心
centers[0] = data[np.random.choice(range(len(data)))]
# 随机选择一个数据点作为第一个聚类中心
for i in range(1, k):
# 对于每个后续聚类中心,计算它与已选定的聚类中心之间的距离,并使用距离构建一个概率分布
distances = np.array([min([euclidean_distance(c, x) for c in centers[:i]]) for x in data])
probabilities = distances / np.sum(distances)
# 计算概率
centers[i] = data[np.random.choice(range(len(data)), p=probabilities)]
# 根据概率选择下一个聚类中心
return centers
# 分配样本点到最近的聚类中心
def assign_clusters(data, centers):
distances = np.zeros((len(data), len(centers)))
# 创建一个数组,存储每个样本点到每个聚类中心的距离
for i, sample in enumerate(data):
for j, center in enumerate(centers):
distances[i, j] = euclidean_distance(sample, center)
# 计算样本点到聚类中心的距离
return np.argmin(distances, axis=1)
# 返回每个样本点所属的最近的聚类索引
# 更新聚类中心
def update_centers(data, clusters, k):
centers = np.zeros((k, data.shape[1]))
# 创建一个k行,每行包含数据的特征数列的零数组,用于存储新的聚类中心
for i in range(k):
cluster_samples = data[clusters == i]
# 提取属于第i个聚类的样本点
if len(cluster_samples) > 0:
centers[i] = np.mean(cluster_samples, axis=0)
# 计算属于第i个聚类的样本点的均值作为新的聚类中心
return centers
# K-means++聚类算法
def k_means(data, k, max_iterations=100):
centers = initialize_centers(data, k)
# 初始化聚类中心
for _ in range(max_iterations):
prev_centers = centers.copy()
# 复制当前的聚类中心
clusters = assign_clusters(data, centers)
# 分配样本点到聚类中心
centers = update_centers(data, clusters, k)
# 更新聚类中心
if np.all(prev_centers == centers):
# 如果当前的聚类中心与上一轮的聚类中心相同,则停止迭代
break
return clusters, centers
# 返回最终的聚类结果和聚类中心
data = load_iris_data()
k = 3
clusters, centers = k_means(data, k)
# 绘制聚类结果
plt.scatter(data[:, 0], data[:, 1], c=clusters, cmap="viridis")
plt.scatter(centers[:, 0], centers[:, 1], marker="^", color="red", s=100, label="Centroids")
plt.xlabel("sepal length (cm)")
plt.ylabel("sepal width (cm)")
plt.title("K-means++ Clustering")
plt.legend()
plt.savefig("data/k-means++聚类结果.png")
plt.show()
源码分析
导入NumPy、Pandas和Matplotlib库,用于数据处理、数值计算和可视化。
load_iris_data()函数加载"Iris.csv"中的数据集,移除"Species"列,返回NumPy数组。
euclidean_distance(a, b)函数计算a和b之间的欧氏距离,使用NumPy的linalg.norm()函数。
initialize_centers(data, k)函数初始化聚类中心,随机选择第一个中心,然后根据距离构建概率分布选择后续中心。
assign_clusters(data, centers)函数将样本点分配到最近的聚类中心,使用np.argmin()找到最近中心的索引。
update_centers(data, clusters, k)函数更新聚类中心为每个聚类的样本点均值。
k_means(data, k, max_iterations=100)函数执行K-means++聚类,通过initialize_centers()、assign_clusters()和update_centers()迭代更新聚类中心。
加载Iris数据集到变量data。
设置聚类数量k为3。
调用k_means()进行聚类,得到聚类结果clusters和聚类中心centers。
使用Matplotlib绘制散点图,表示数据点和聚类中心,设置标签、标题、图例,并保存图像。
通过实现K-means++聚类算法,并对Iris数据集进行了聚类分析,最终生成散点图展示聚类结果。
2.4.3 K_medoids算法

图2-5
运行结果

图2-6
实验代码:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 导入Iris数据集
def load_iris_data():
data = pd.read_csv("data/iris.csv") # 假设数据集保存在名为"iris.csv"的文件中
data = data.drop("Species", axis=1) # 移除类别列
return data.values
# 曼哈顿距离
def manhattan_distance(a, b):
return np.sum(np.abs(a - b))
# 初始化聚类中心(Medoids)
def initialize_medoids(data, k):
medoids = np.zeros(k, dtype=int)
n = len(data)
indices = np.arange(n)
np.random.shuffle(indices)
medoids = indices[:k]
return medoids
# 分配样本点到最近的聚类中心
def assign_clusters(data, medoids):
clusters = np.zeros(len(data), dtype=int)
for i, point in enumerate(data):
distances = [manhattan_distance(point, data[m]) for m in medoids]
clusters[i] = np.argmin(distances)
return clusters
# 更新聚类中心(Medoids)
def update_medoids(data, clusters, medoids):
new_medoids = np.copy(medoids)
for i in range(len(medoids)):
cluster_points = data[clusters == i]
cluster_distances = np.zeros(len(cluster_points))
for j, point in enumerate(cluster_points):
other_distances = np.sum(manhattan_distance(point, other) for other in cluster_points)
cluster_distances[j] = other_distances
new_medoid_index = np.argmin(cluster_distances)
new_medoids[i] = np.where(clusters == i)[0][new_medoid_index]
return new_medoids
# K-medoids聚类算法
def k_medoids(data, k, max_iterations=100):
medoids = initialize_medoids(data, k)
clusters = assign_clusters(data, medoids)
for _ in range(max_iterations):
new_medoids = update_medoids(data, clusters, medoids)
new_clusters = assign_clusters(data, new_medoids)
if np.array_equal(clusters, new_clusters):
break
medoids = new_medoids
clusters = new_clusters
return clusters, medoids
# 加载Iris数据集
data = load_iris_data()
# 设置聚类的数量k
k = 3
# 运行K-medoids聚类算法
clusters, medoids = k_medoids(data, k)
# 绘制聚类结果
plt.scatter(data[:, 0], data[:, 1], c=clusters, cmap="viridis") # 绘制数据点,颜色根据聚类结果clusters来区分,使用颜色映射"viridis"
plt.scatter(data[medoids, 0], data[medoids, 1], c="red", marker="^") # 绘制聚类中心,使用红色的三角形表示
plt.xlabel("sepal length (cm)") # 设置x轴标签
plt.ylabel("sepal width (cm)") # 设置y轴标签
plt.title("K-medoids Clustering") # 设置图的标题为"K-medoids Clustering"
plt.savefig("data/k-medoids聚类结果.png") # 保存图像为文件
plt.show()
源码分析
- load_iris_data()函数:从名为"iris.csv"的文件中加载Iris数据集,并移除其中的"Species"列。函数返回数据集的值部分(去除了标签列)。
- manhattan_distance(a, b)函数:计算两个向量a和b之间的曼哈顿距离,通过计算两个向量对应元素差的绝对值之和来实现。
- initialize_medoids(data, k)函数:根据指定的聚类数量k,从数据集中随机选择k个样本作为初始的聚类中心(medoids),并返回这些样本的索引。
- assign_clusters(data, medoids)函数:将数据集中的每个样本点分配到最近的聚类中心。对于每个样本点,计算它与每个聚类中心的曼哈顿距离,然后将该样本分配给距离最近的聚类中心的索引。
- update_medoids(data, clusters, medoids)函数:基于当前的聚类分配,更新聚类中心(medoids)。对于每个聚类,计算该聚类内所有样本点两两之间的距离之和,选择距离和最小的样本点作为新的聚类中心。
- k_medoids(data, k, max_iterations=100)函数:实现K-medoids聚类算法。首先,初始化聚类中心,然后进行以下步骤:分配样本点到最近的聚类中心,更新聚类中心,直到达到最大迭代次数或聚类分配不再改变为止。函数返回最终的聚类结果和聚类中心。
- 加载Iris数据集:调用load_iris_data()函数加载Iris数据集,并将返回的数据赋值给变量data。
- 设置聚类的数量k:将聚类数量设置为3,赋值给变量k。
- 运行K-medoids聚类算法:调用k_medoids()函数,传入数据集和聚类数量k,得到最终的聚类结果和聚类中心,并分别赋值给变量clusters和medoids。
- 绘制聚类结果:使用Matplotlib绘制聚类结果的散点图。调用scatter()函数绘制数据点,使用聚类结果clusters来确定每个数据点的颜色,并使用"viridis"颜色映射。再调用scatter()函数绘制聚类中心。
- 设置x轴和y轴标签:使用xlabel()和ylabel()函数设置x轴和y轴的标签为"sepal length (cm)"和"sepal width (cm)"。
- 保存图像:使用savefig()函数将图像保存为文件。
2.4.4 DBScan算法

图2-7
运行结果

图2-8
实验代码:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 导入Iris数据集
def load_iris_data():
data = pd.read_csv("data/iris.csv") # 假设数据集保存在名为"iris.csv"的文件中
data = data.drop("Species", axis=1) # 移除类别列
return data.values
# 欧氏距离
def euclidean_distance(a, b):
return np.linalg.norm(a - b)
# 寻找在给定点半径范围内的邻域数据点
def region_query(data, point_index, epsilon):
distances = np.linalg.norm(data - data[point_index], axis=1)
return np.where(distances <= epsilon)[0]
# 扩展簇,将邻域内的点添加到同一簇中
def expand_cluster(data, cluster_labels, point_index, neighbors, cluster_id, epsilon, min_samples):
cluster_labels[point_index] = cluster_id
i = 0
while i < len(neighbors):
neighbor_index = neighbors[i]
if cluster_labels[neighbor_index] == 0: # 未分类的点
cluster_labels[neighbor_index] = cluster_id
new_neighbors = region_query(data, neighbor_index, epsilon)
if len(new_neighbors) >= min_samples:
neighbors = np.concatenate((neighbors, new_neighbors))
i += 1
# DBSCAN聚类算法
def dbscan(data, epsilon, min_samples):
cluster_labels = np.zeros(len(data), dtype=int)
cluster_id = 0
for i in range(len(data)):
if cluster_labels[i] == 0: # 未分类的点
neighbors = region_query(data, i, epsilon)
if len(neighbors) < min_samples: # 边界点
cluster_labels[i] = -1
else: # 核心点
cluster_id += 1
expand_cluster(data, cluster_labels, i, neighbors, cluster_id, epsilon, min_samples)
return cluster_labels
# 加载Iris数据集
data = load_iris_data()
epsilon = 1 # 设置半径
min_samples = 1 # 设置每个类别的最小样本量
cluster_labels = dbscan(data, epsilon, min_samples)
# 绘制聚类结果
plt.scatter(data[:, 0], data[:, 1], c=cluster_labels, cmap="viridis") # 绘制数据点,颜色根据聚类结果cluster_labels来区分,使用颜色映射"viridis"
plt.xlabel("sepal length (cm)") # 设置x轴标签
plt.ylabel("sepal width (cm)") # 设置y轴标签
plt.title("DBSCAN Clustering") # 设置图的标题为"DBSCAN Clustering"
plt.savefig("data/DBScan聚类结果") # 保存图像为文件
plt.show() # 显示图像
源码分析
- 定义函数 load_iris_data() 导入Iris数据集。该函数读取名为 "iris.csv" 的文件,并移除数据集中的类别列,然后返回数据的值部分(去除了类别信息)。
- 定义函数 euclidean_distance(a, b) 计算两个向量 a 和 b 之间的欧氏距离。该函数使用 numpy.linalg.norm() 函数来计算向量的范数,即欧氏距离。
- 定义函数 region_query(data, point_index, epsilon) 寻找在给定点的半径范围内的邻域数据点。该函数计算数据集 data 中每个点与指定点之间的欧氏距离,并返回在半径 epsilon 范围内的点的索引。
- 定义函数 expand_cluster(data, cluster_labels, point_index, neighbors, cluster_id, epsilon, min_samples),用于将邻域内的点添加到同一簇中。
- 定义函数 dbscan(data, epsilon, min_samples) 实现了DBSCAN聚类算法。该函数使用一个数组 cluster_labels 来记录每个数据点所属的簇,遍历数据集中的每个点,对未分类的点进行处理。如果一个点的邻域内的点数量小于最小样本量 min_samples,则将该点标记为边界点(簇标签为-1)。否则,将该点标记为核心点,并将其与邻域内的点扩展为同一簇。
- 加载Iris数据集并存储在变量 data 中。
- 设置参数 epsilon 和 min_samples,分别表示邻域半径和每个簇的最小样本量。
- 调用函数 dbscan(data, epsilon, min_samples) 执行DBSCAN聚类算法,并将聚类结果存储在变量 cluster_labels 中。
2.4.5 凝聚聚类算法

图2-9
运行结果
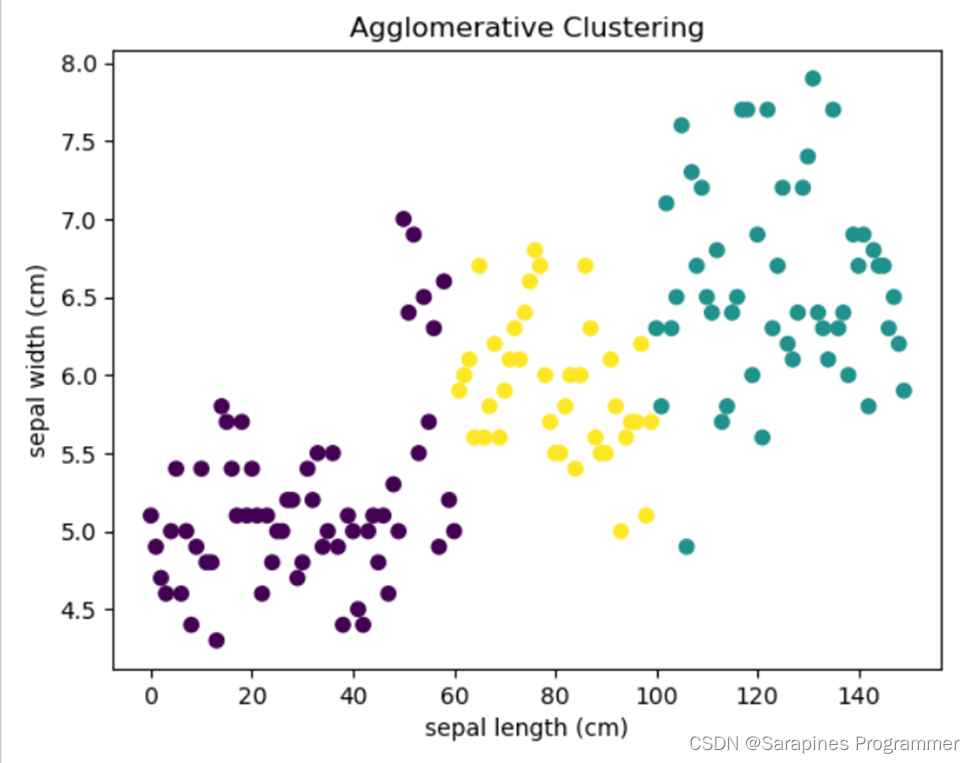
图2-10
实验代码:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.cluster import AgglomerativeClustering
# 导入Iris数据集
def load_iris_data():
data = pd.read_csv("data/iris.csv") # 假设数据集保存在名为"iris.csv"的文件中
data = data.drop("Species", axis=1) # 移除标签列
return data.values
# 加载Iris数据集
data = load_iris_data()
# 执行凝聚层次聚类
clustering = AgglomerativeClustering(n_clusters=3) # 创建AgglomerativeClustering对象并指定聚类个数为3
cluster_labels = clustering.fit_predict(data) # 对数据进行聚类并获取聚类标签
# 绘制聚类结果
plt.scatter(data[:, 0], data[:, 1], c=cluster_labels, cmap="viridis") # 绘制数据点,颜色根据聚类结果cluster_labels来区分,使用颜色映射"viridis"
plt.xlabel("sepal length (cm)") # 设置x轴标签
plt.ylabel("sepal width (cm)") # 设置y轴标签
plt.title("Agglomerative Clustering") # 设置图的标题为"Agglomerative Clustering"
plt.savefig("data/Agglomerative聚类结果.png") # 保存图像为文件
plt.show() # 显示图像
源码分析
- 定义了一个函数 load_iris_data(),用于加载 Iris 数据集。函数通过使用 pd.read_csv() 从名为 "iris.csv" 的文件中读取数据集,然后通过 drop() 方法移除标签列 "Species",最后返回数据的值。
- 调用 load_iris_data() 函数加载 Iris 数据集,并将数据赋值给变量 data。
- 创建AgglomerativeClustering对象,通过 AgglomerativeClustering(n_clusters=3) 指定聚类个数为 3。这里的参数 n_clusters 表示要聚类成的簇的数量。
- 使用 fit_predict() 方法对数据进行聚类,并将聚类标签存储在变量 cluster_labels 中。fit_predict() 方法首先拟合数据,然后根据拟合的模型对数据进行聚类并返回每个数据点的聚类标签。
- 绘制聚类结果图。使用 plt.scatter() 函数绘制数据点,其中 x 坐标为 data[:, 0],y 坐标为 data[:, 1],颜色根据聚类结果 cluster_labels 区分,使用颜色映射 "viridis"。
- 使用 plt.xlabel() 设置 x 轴标签为 "sepal length (cm)"。
- 使用 plt.ylabel() 设置 y 轴标签为 "sepal width (cm)"。
- 使用 plt.title() 设置图的标题为 "Agglomerative Clustering"。
- 使用 plt.savefig() 将绘制的聚类结果图保存为文件。文件名为 "Agglomerative聚类结果.png",保存在名为 "data" 的文件夹中。
- 最后,使用 plt.show() 显示绘制的图像。这将在图形窗口中显示聚类结果图。
2.5 实验心得
这次实验如同踏足深邃的聚类算法探索之旅,涵盖了K-means、K-medoids、DBSCAN和凝聚聚类等引人瞩目的算法。
K-means通过不懈的迭代,将样本点巧妙地划分到K个簇中,并通过持续更新聚类中心的手法,不断提炼出聚类结果的精髓。虽然其简约高效,却对初始聚类中心的选择极为敏感,于是乎,K-means++以一种独特的方式改善了这一选择过程,从而显著提升了聚类的优越性。
K-medoids则以实际样本点为聚类中心,使得整个过程更为稳健,然而在处理大规模数据时,或许会面临着计算复杂度的考验。
DBSCAN以一种独特的方式通过密度可达关系扩展簇的规模,但对参数的选择显得尤为挑剔,需要谨慎斟酌,方可发挥其优势。
而凝聚聚类算法如同绘画一般,从每个样本点作为独立簇起步,逐渐融合最为相似的簇,从而呈现出层次化的聚类结果。然而,这一过程的计算复杂度较高,尤其在处理大规模数据时可能遭遇性能上的制约。
这次实验深刻地引领我步入这些算法的深邃世界,深刻理解它们的原理、特点和应用场景。在选择聚类算法时,需全面考虑数据的独特之处、规模的宏观因素、计算资源的稀缺与充足,以及对所需聚类结果形式的渴望等多重要素。同时,算法的效果不仅仅在于其自身,更在于恰如其分的参数选择,这是一个需要精心权衡的决定性因素。
致读者
风自火出,家人;君子以言有物而行有恒