nrrd数据格式
MITK默认会将医学图像保存为格式为NRRD的图像,在这个数据格式中包含:
1、一个单个的数据头文件:为科学可视化和医学图像处理准确地表示N维度的栅格信息。
2、既能分开又能合并的图像文件。
nrrd_options输出
{u’dimension’: 3, # 维度
u’encoding’: ‘raw’, # 编码方式
u’endian’: ‘little’, #
u’keyvaluepairs’: {},
u’kinds’: [‘domain’, ‘domain’, ‘domain’], # 三个维度的类型
u’sizes’: [30, 30, 30], #三个维度的大小
u’space’: ‘left-posterior-superior’, # 空间信息
u’space directions’: [[‘1’, ‘0’, ‘0’], [‘0’, ‘1’, ‘0’], [‘0’, ‘0’, ‘1’]],
u’space origin’: [‘0’, ‘0’, ‘0’],
u’type’: ‘short’}
dcm数据格式
DICOM(DigitalImaging andCommunications inMedicine)是指医疗数字影像传输协定,是用于医学影像处理、储存、打印、传输的一组通用的标准协定。
DCM格式的文件会存储其各类重要信息。其中,常用到
(1)Patient ID : 患者ID (一般与患者的文件夹命名一致);
(2)Series Number : 属于同一三维数据的该序号应当是一样的,例如都是同一CT图像中的某张切片,那么该值是一样,通过该值判断是不是属于同一个三维数据(可能一个患者拍了多张CT图像);
(3)Image Position :该切片左上角像素点的空间位置。简而言之,利用Series Number找出患者文件夹下的所有属于同一三维图像的所有切片,然后利用Image Position对这些切片进行排序并重构成三维数据。
这里仅展示二维读取
代码实现
import nrrd
import pydicom
import numpy as np
import matplotlib.pyplot as plt
# dicom文件
path = ".dcm"
ds = pydicom.dcmread(path,force=True)
data = np.array(ds.pixel_array)
plt.figure()
plt.imshow(data,cmap='gray')
plt.axis('off')
plt.show()
#nrrd文件
nrrd_filename = '.nrrd'
nrrd_data, nrrd_options = nrrd.read(nrrd_filename)
print(nrrd_data.shape)
data =nrrd_data[:,:,50]#取切片
data = np.array(ds.pixel_array)
def WLww(image,WL,WW):
min_v = (2 * WL - WW) / 2.0
max_v = (2 * WL + WW) / 2.0
img1= (image-min_v)/WW
img1[img1>1] = 1
img1[img1<0] = 0
img1=img1*255
return img1
data = WLww(data,20,400)
plt.figure()
plt.imshow(data,cmap='gray')
plt.axis('off')
plt.show()
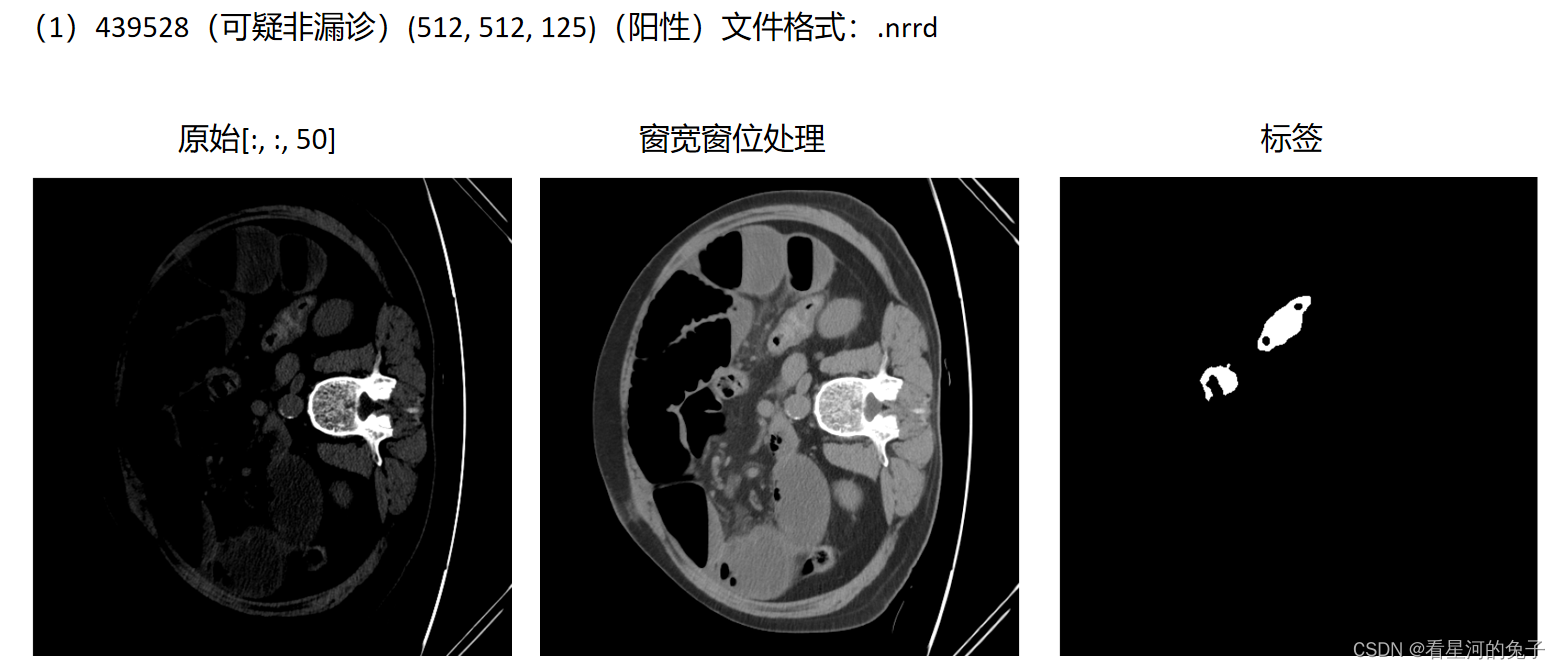
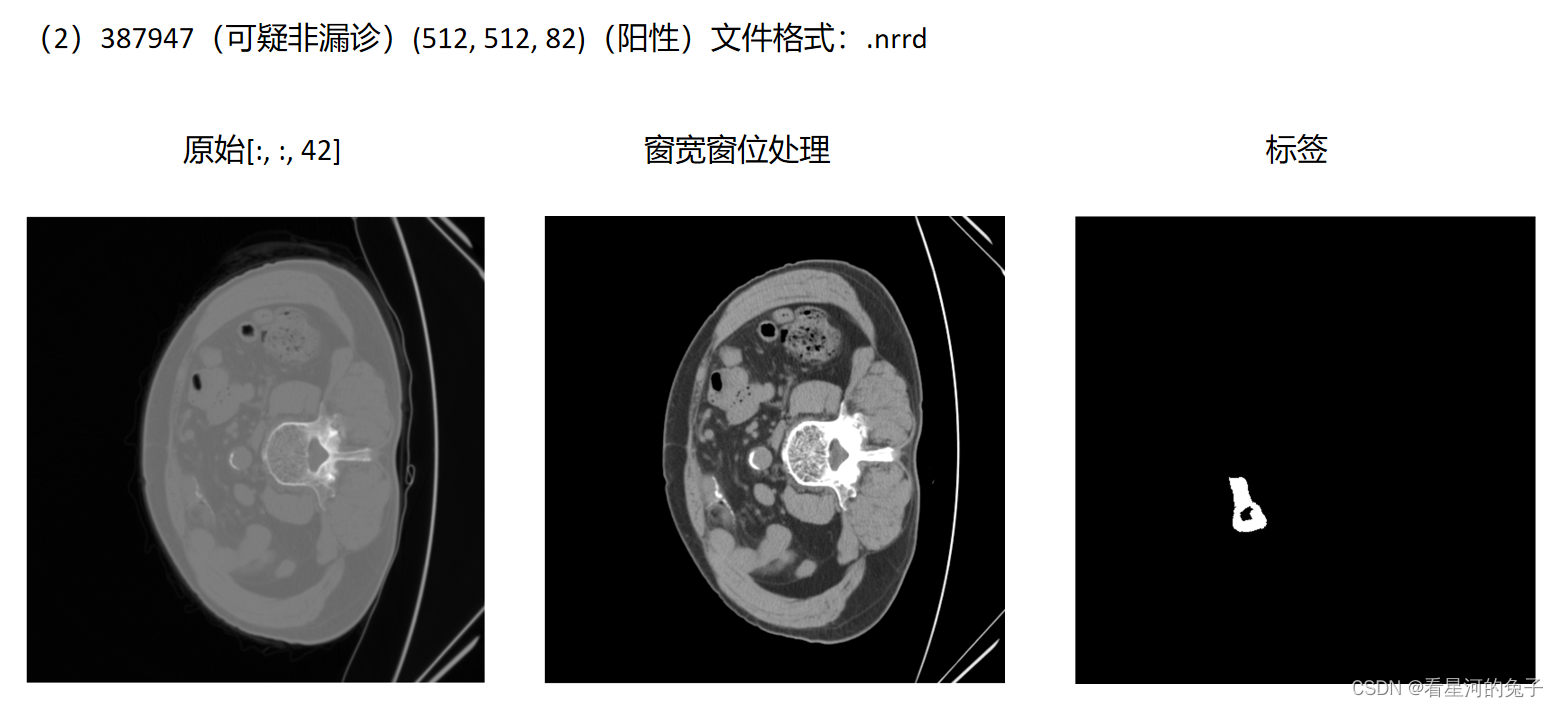
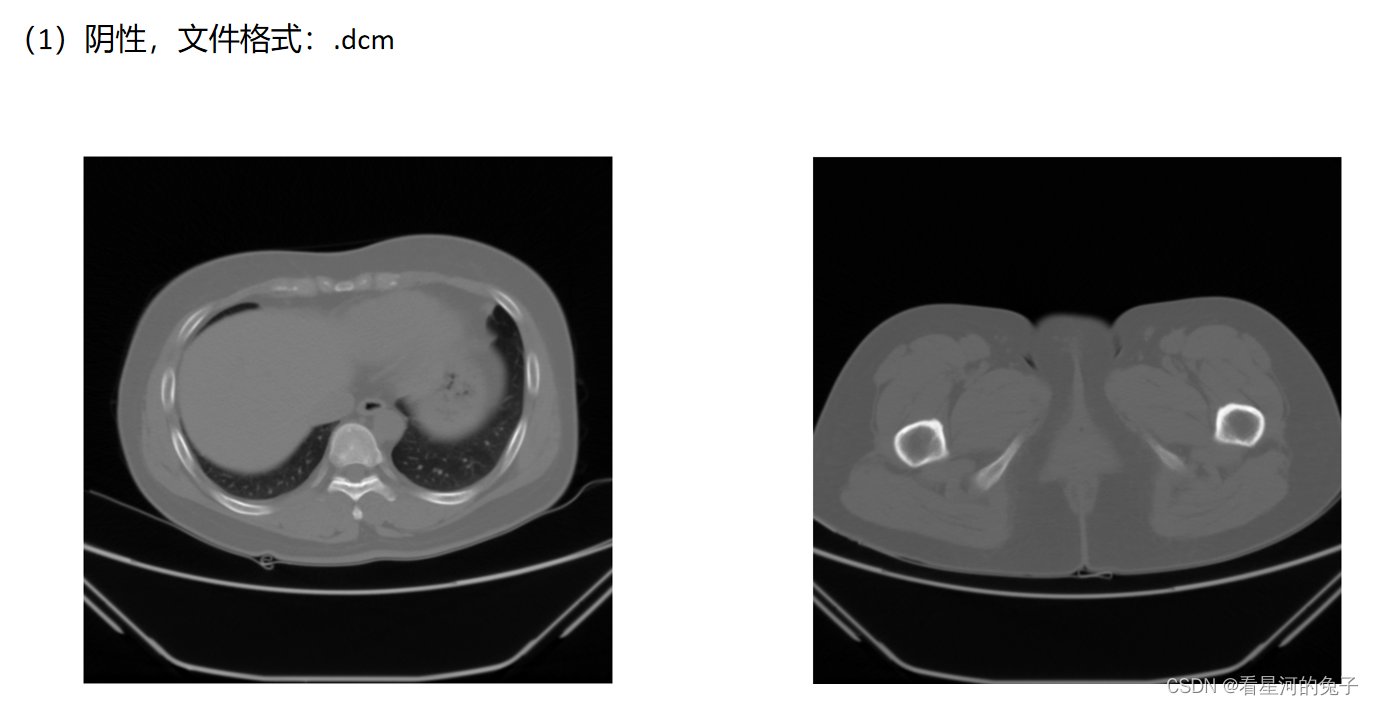
结果展示