实验篇—— 基因家族Motif 分析
文章目录
- 前言
- 一、名词解释
- 二、实操
- 1. MEME工具箱
- 2. Motif Discovery(基序发现)
- 1. 结果网页
- 2. 在TBtools中(额外)
- 2. Motif Enrichment(基序富集分析)
- 3. Motif Search(基序搜索)
- 4. Motif Comparison(基序比较)
- 总结
前言
参考文献:
https://www.jianshu.com/p/6bb6967c455d
一、名词解释
Motif分析是一种用于识别和分析DNA、RNA或蛋白质序列中重复出现的模式或序列片段的方法。这些模式被称为motif,它们可能具有特定的生物学功能,如结合到特定的转录因子、调控基因表达或参与蛋白质相互作用等。
可以帮助了解基因家族成员之间的关系,推断它们的功能和调控机制。
二、实操
1. MEME工具箱
http://meme-suite.org/index.html
MEME套件允许在未对齐的核苷酸或蛋白质序列集合中发现新的基序,并进行各种基于基序的分析

流程图:
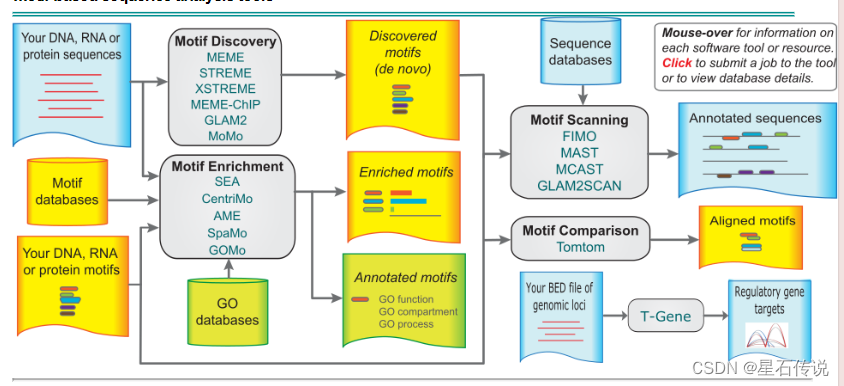
查看介绍的详细信息(点击大标题 "The MEME Suite ")
2. Motif Discovery(基序发现)
用于预测输入序列上的motif信息,支持DNA,RNA或者蛋白序列。
实现该功能的工具有许多,以MEME为例
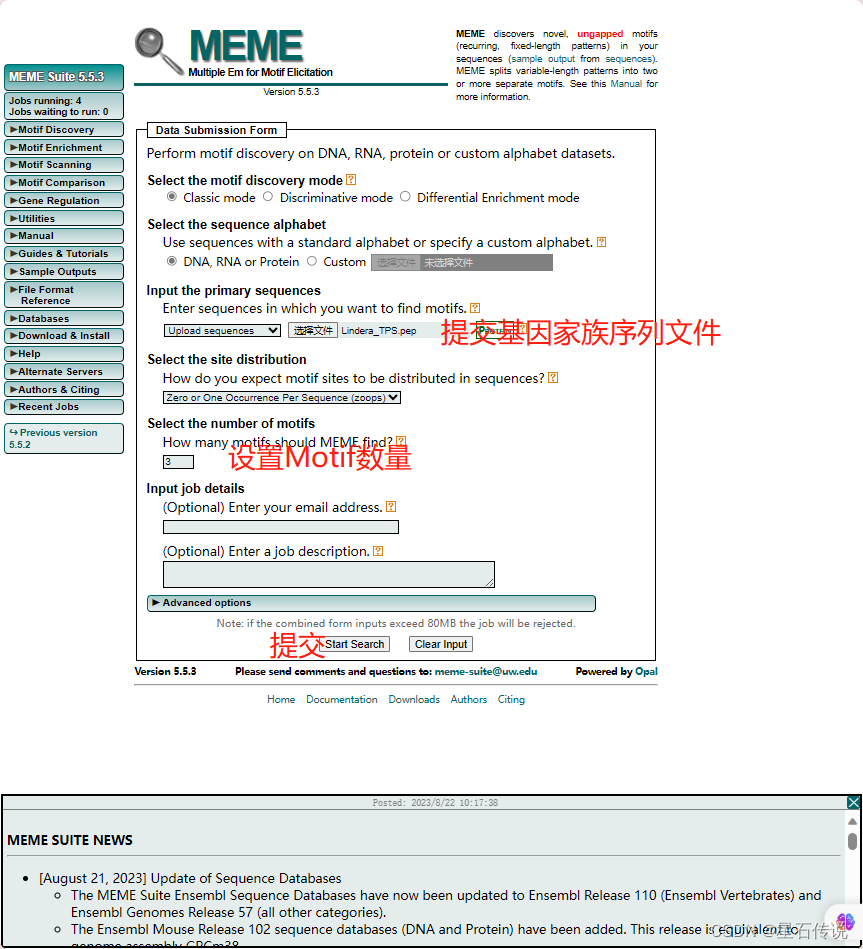
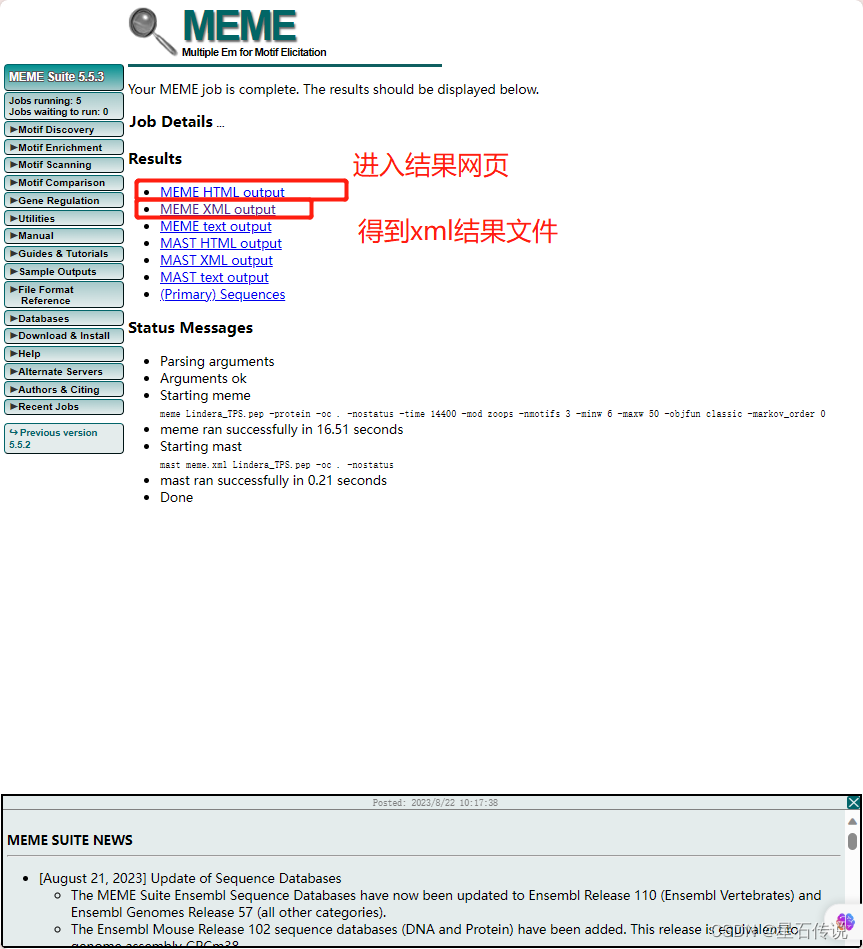
1. 结果网页
直接进入结果网页(比如可以设置Motif数量为 9)
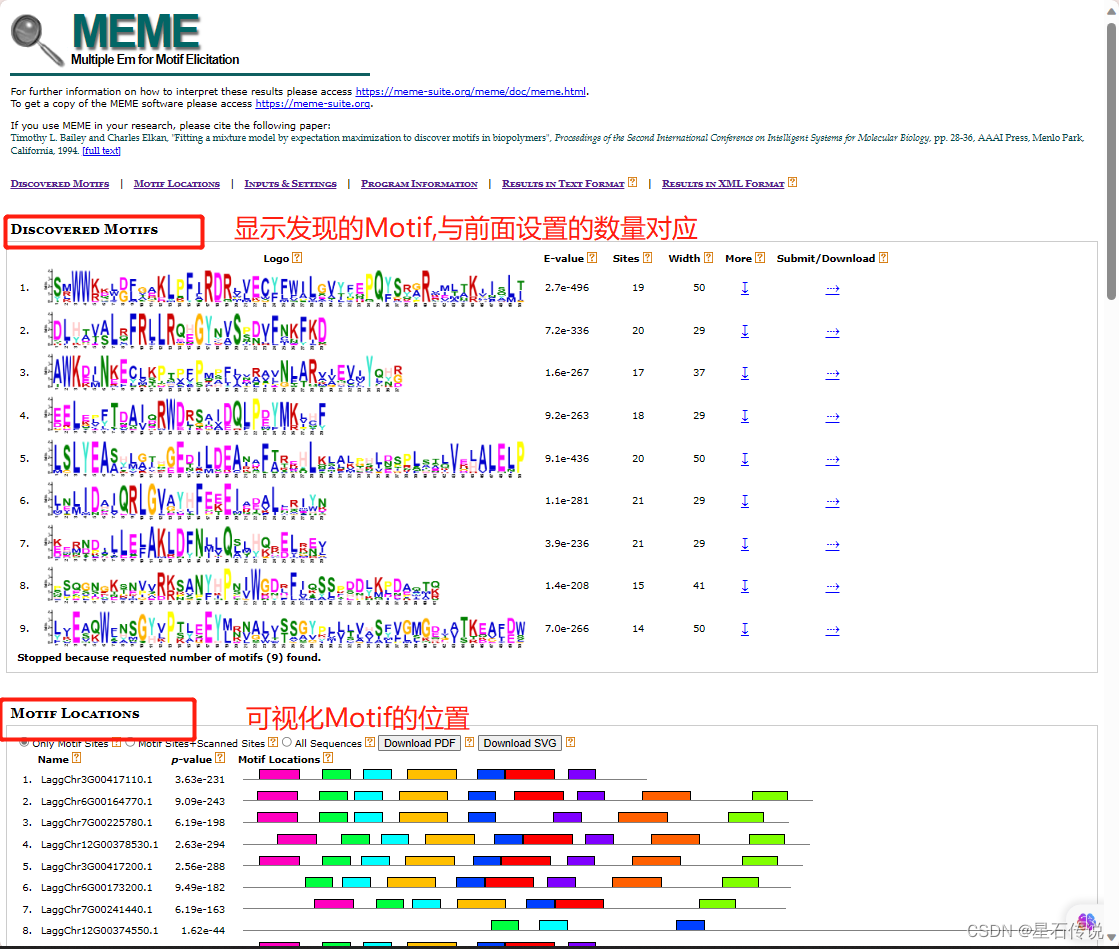
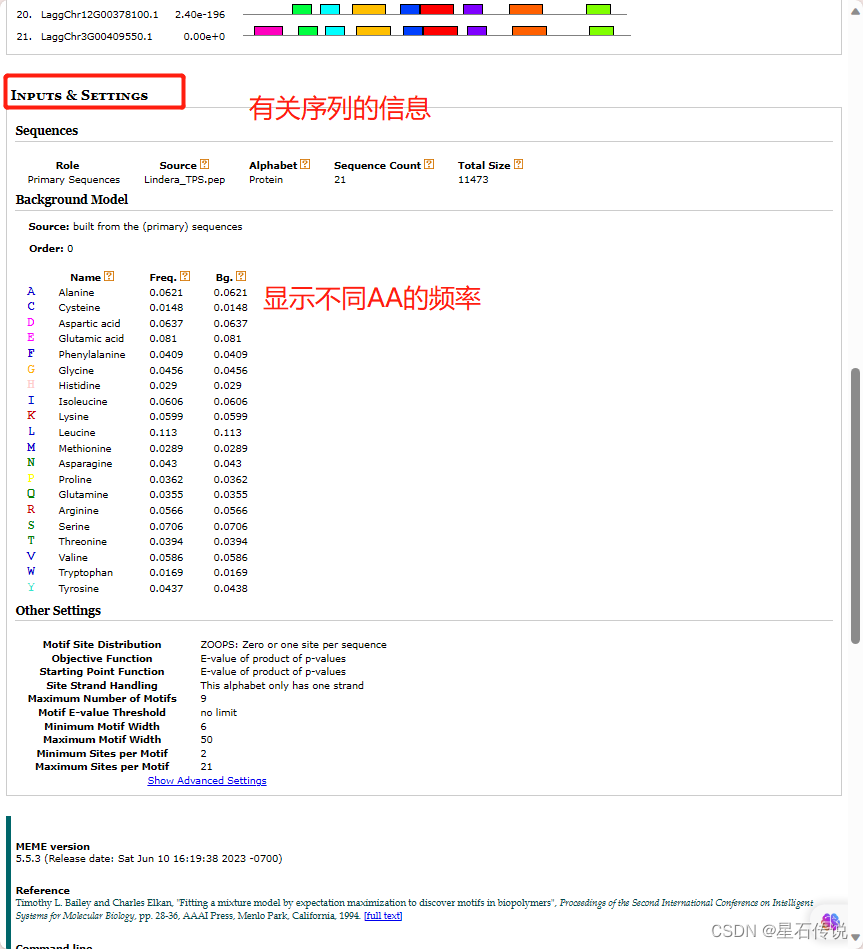

查看More
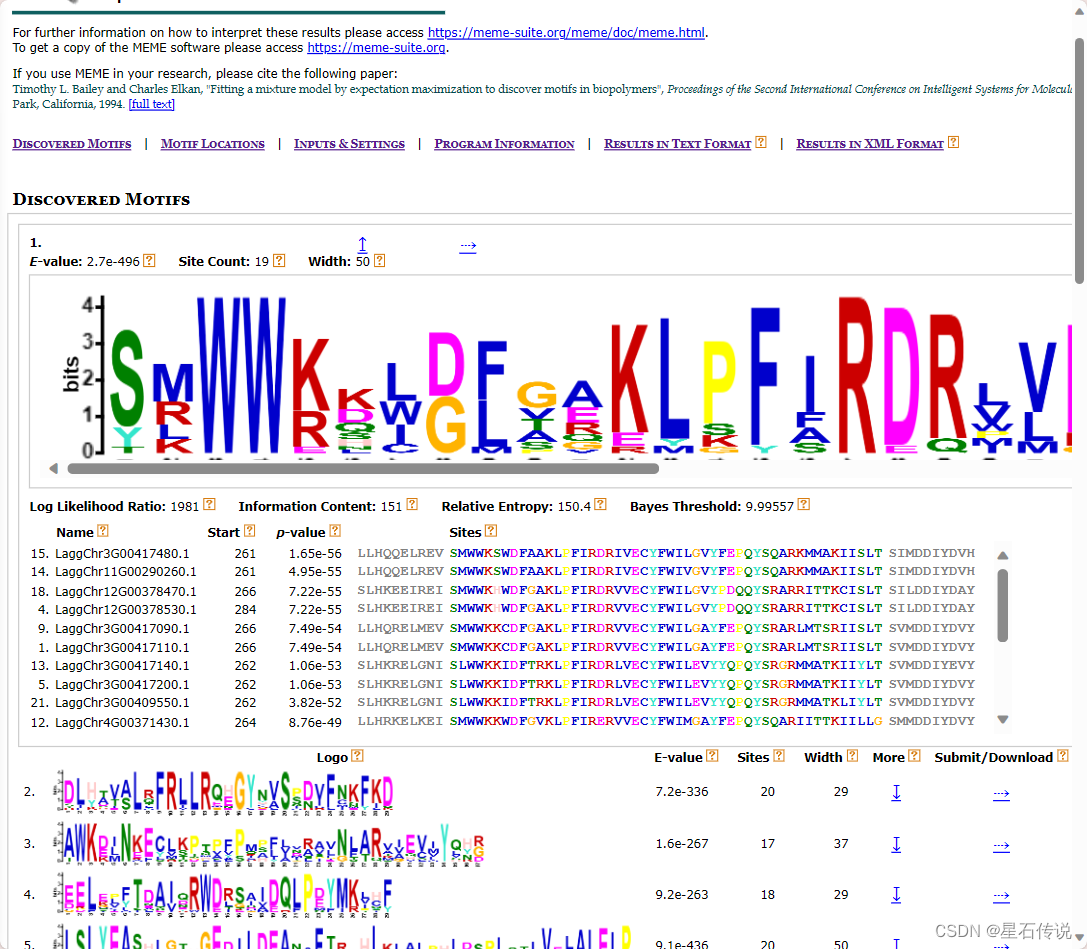
提交/下载 motif
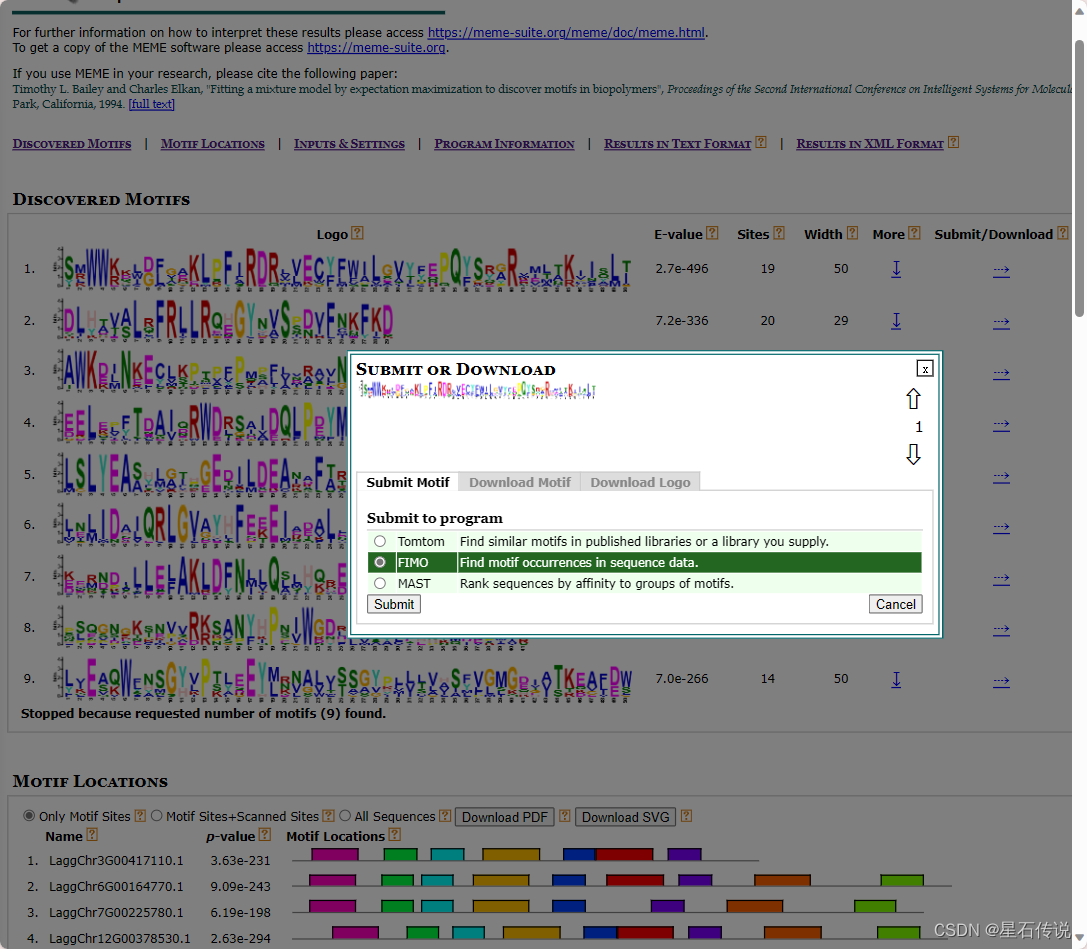
下载 Motif Locations
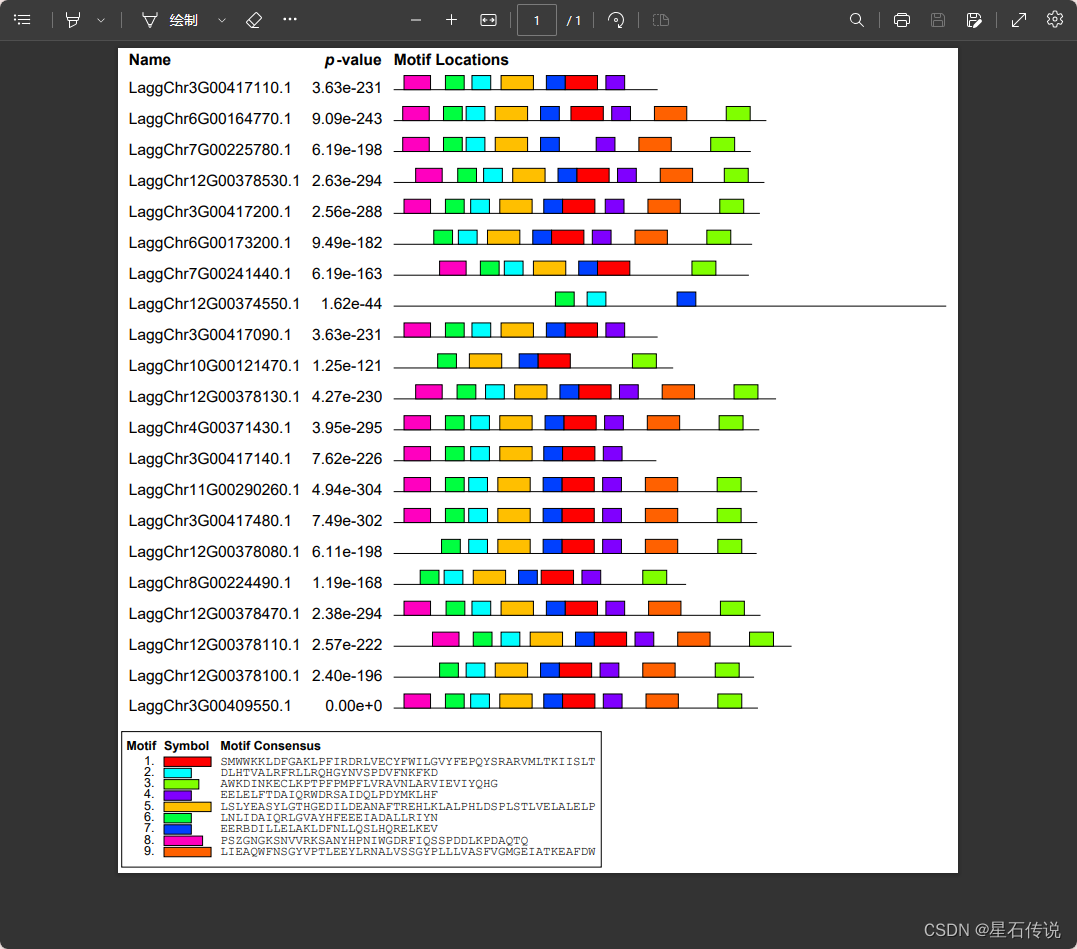
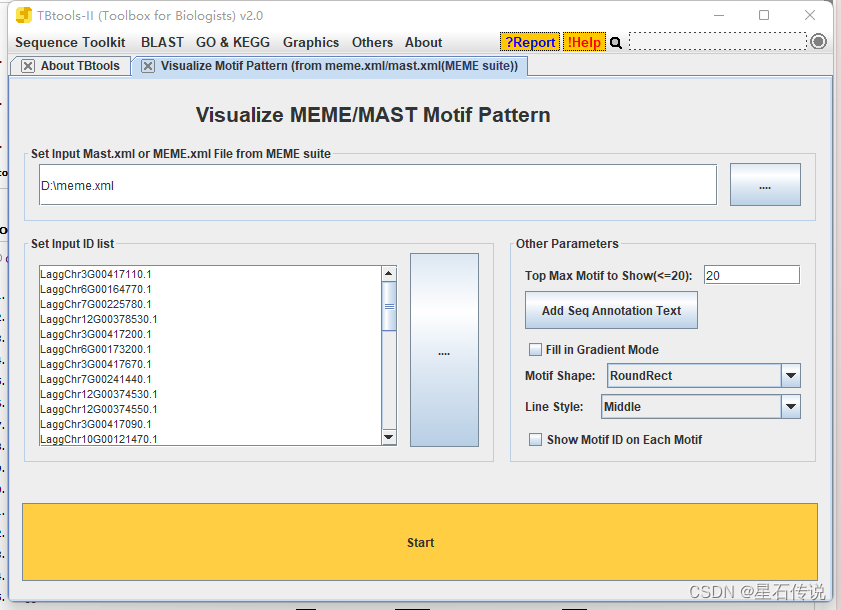
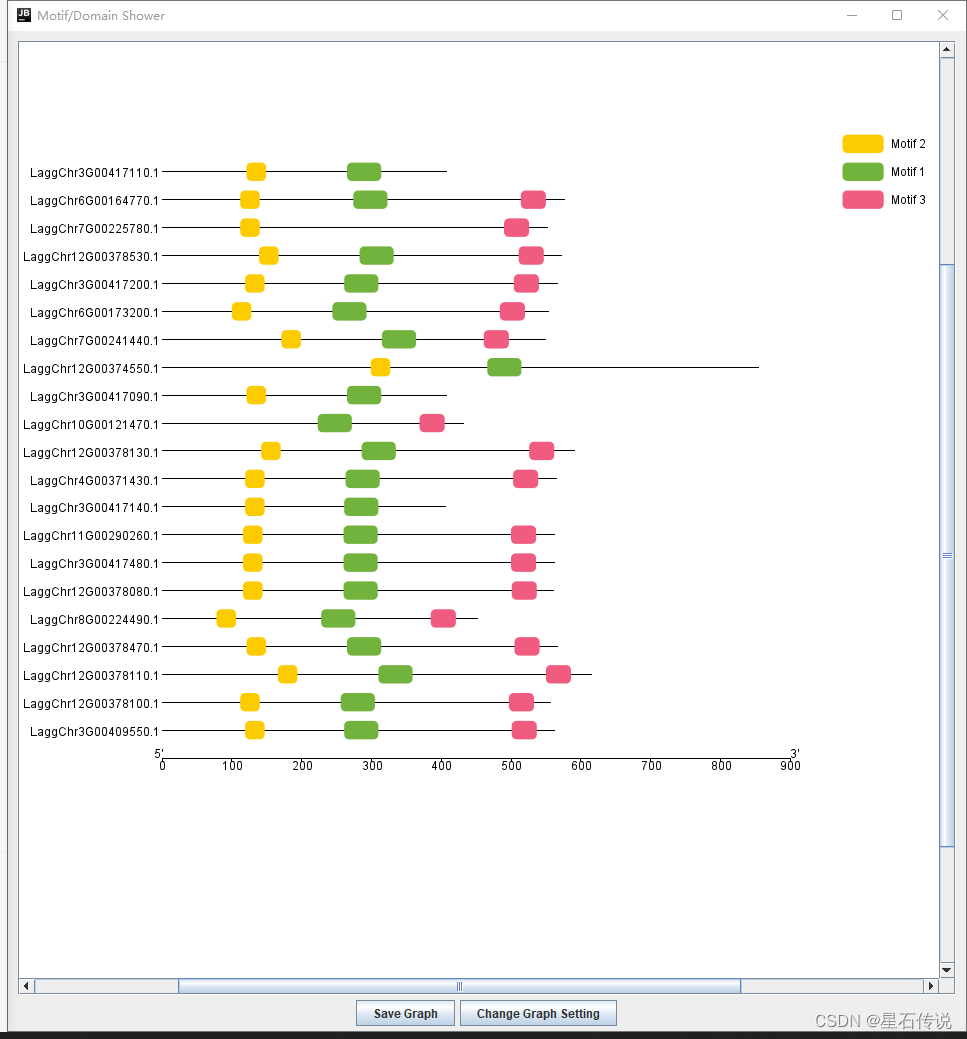
2. 在TBtools中(额外)
或者得到xml文件后,导入TBtools进行可视化
(设置Motif数量为3)
2. Motif Enrichment(基序富集分析)
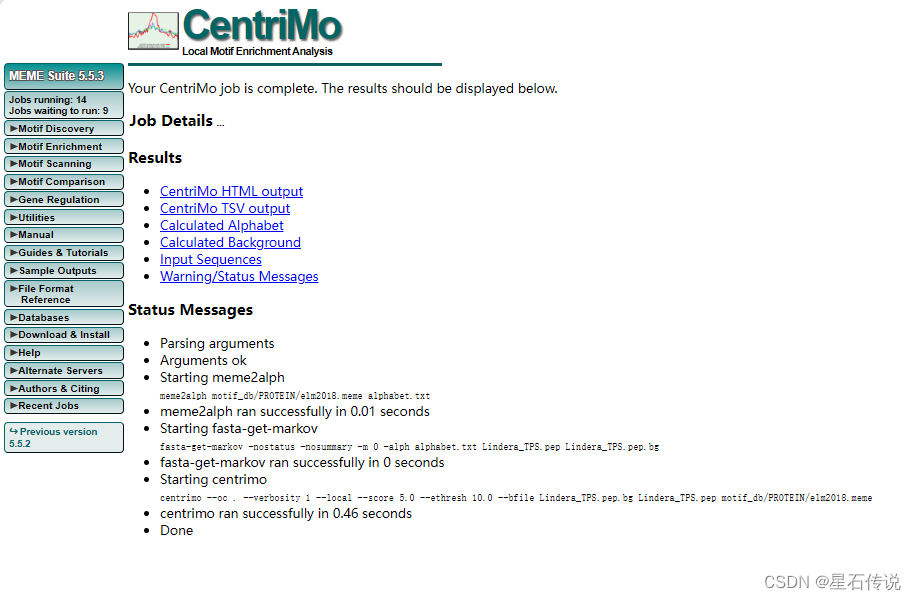
提供了5中工具,这些工具可以测量一组序列中已知基序的富集程度。基序富集可以出现在序列的任何位置(SEA,AME),也可以集中在序列的中心区域(CentriMo)。
以 CentriMo 工具为例:

3. Motif Search(基序搜索)
用于扫描序列以识别与已知基序匹配。以FIMO工具为例。
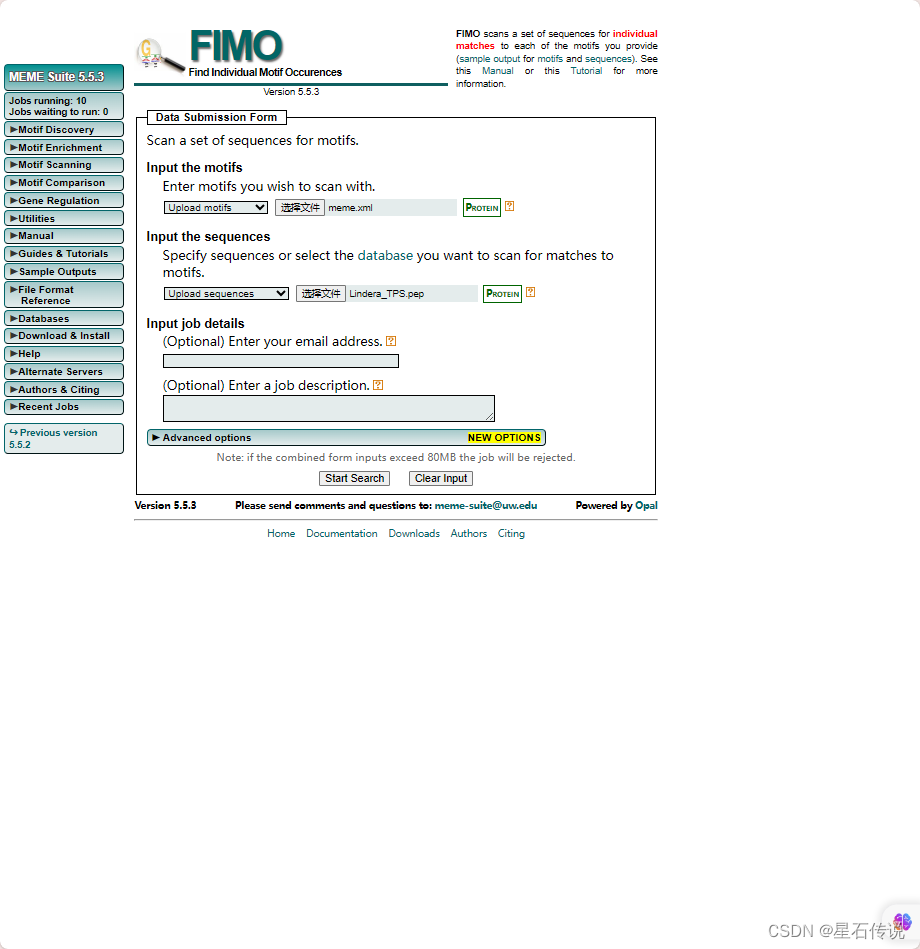
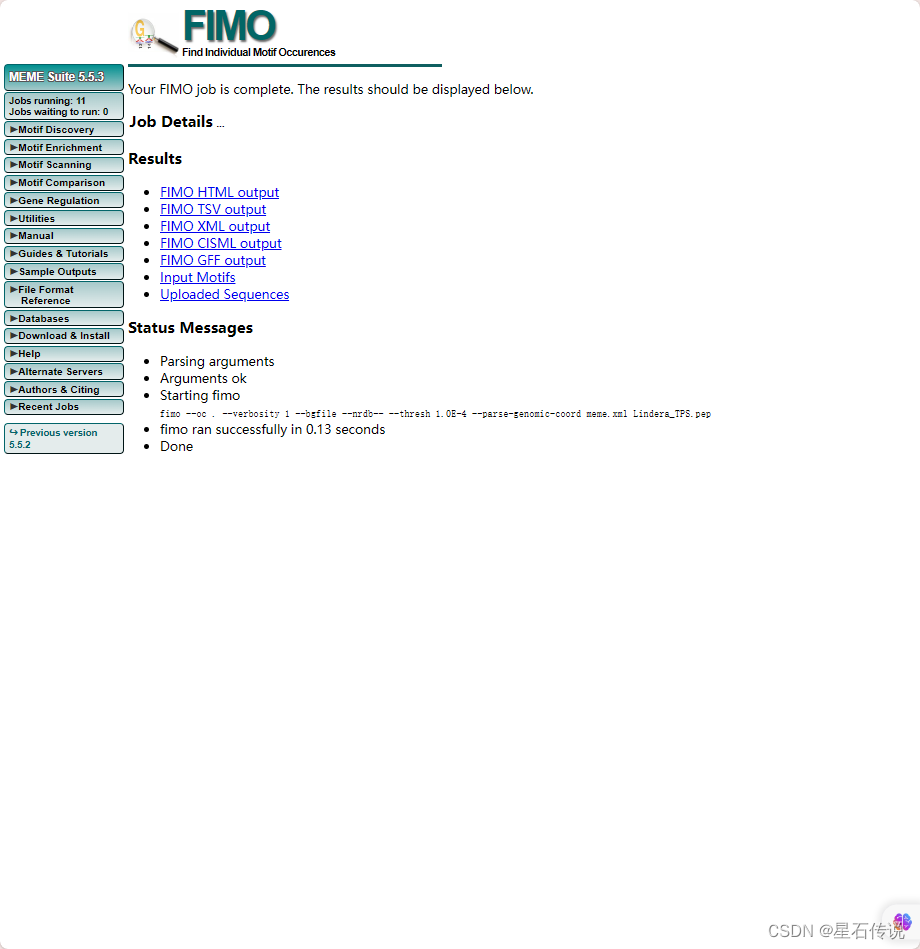
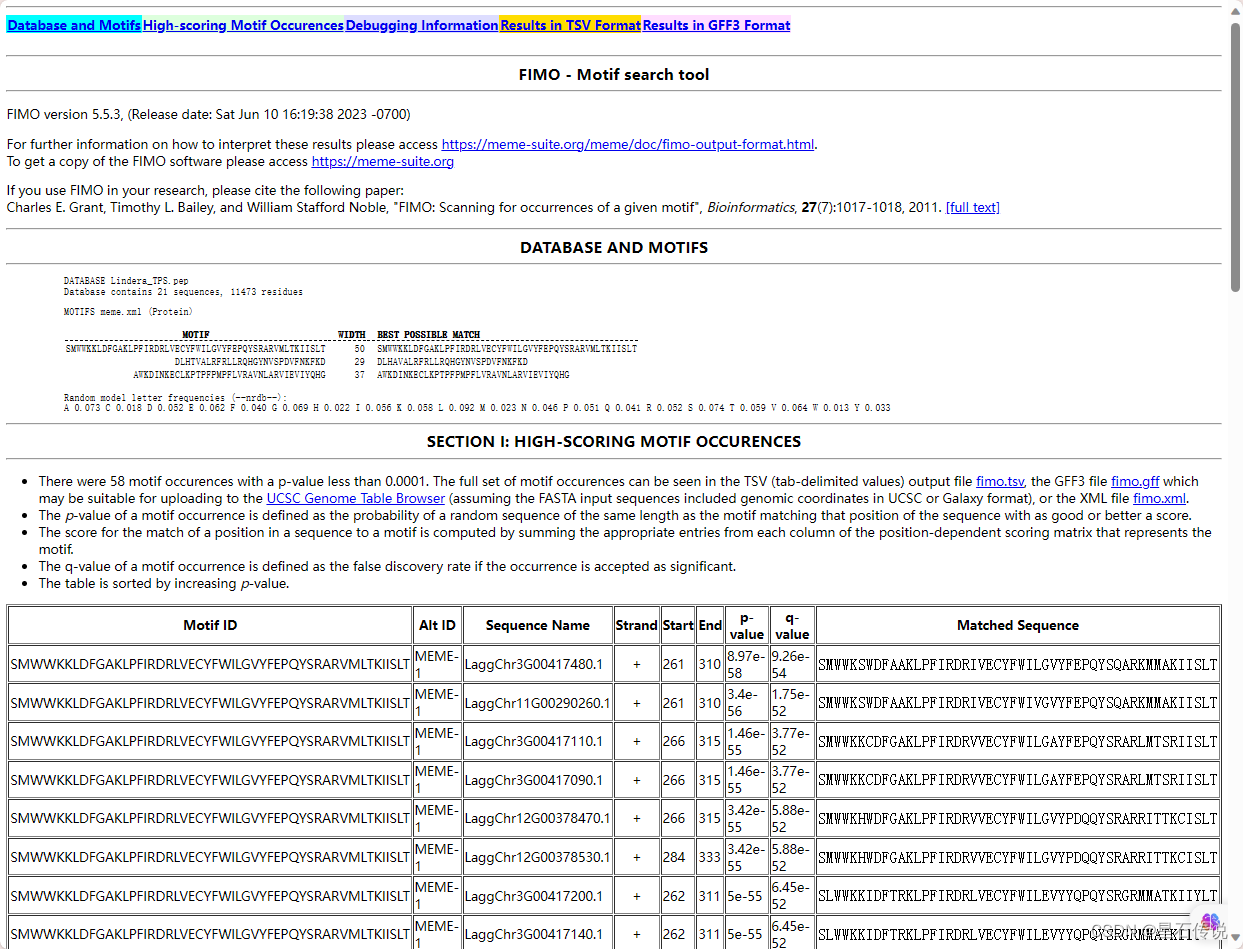
每一列的含义如下:
Motif ID:基序的唯一标识符。
Alt ID:基序的备用标识符。
Sequence Name:匹配基序的序列名称或标识符。
Strand:匹配基序的序列的方向(正向或反向)。
Start:匹配基序在序列中的起始位置。
End:匹配基序在序列中的结束位置。
p-value:基序出现的p值,表示与该位置的序列匹配的与基序相同长度的随机序列具有与或更好得分的概率。
q-value:基序出现的q值,定义为如果接受该出现为显著时的假阳率。
Matched Sequence:与基序匹配的序列片段。
4. Motif Comparison(基序比较)
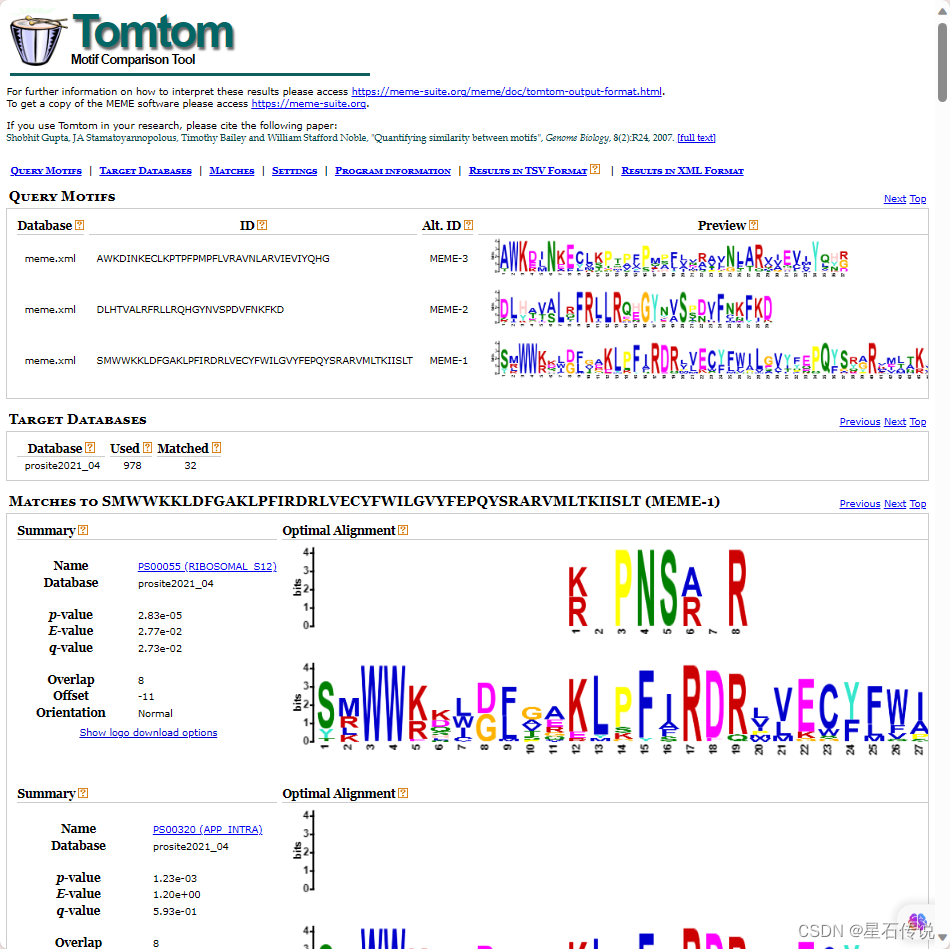
用于衡量基序之间的相似性。比较基序并识别它们之间的相似性或差异。
将分析到的de novo motif与已知的motif数据库进行分析比对,查找相似的motif。使用Tomtom工具

总结
本文主要简述了基于MEME工具箱中的工具进行Motif分析,了解到了有关Motif分析的相关知识。通过该分析可识别序列中重复出现的模式或序列片段。
感时花溅泪,恨别鸟惊心
–2023-8-27


![Maven报错 [ERROR] Malformed \uxxxx encoding.](https://img-blog.csdnimg.cn/df41c676d8a34a4b9c275985a016282d.png)