snpEff变异注释整成人生思考
- 1.介绍
- 2.安装过程以及构建物种参考数据库
- 3.坑货来了
- 4.结果文件判读
- 5.小tips
1.介绍
  SnpEff(Snp Effect)是一个用于预测基因组变异(例如单核苷酸变异、插入、缺失等)对基因功能的影响的生物信息学工具。它可以帮助研究人员和生物信息学家分析基因组变异并预测这些变异可能对基因产生的影响。
2.安装过程以及构建物种参考数据库
其实这些我感觉大佬谢大飞的博客就写的挺全面的
大佬的博客@谢大飞:SnpEff安装使用及报错解决
主要就是一个多看snpeff官网文档确实有收获比如:
data文件目录结构吧它就是创建物种名称文件夹下面:

这就够了。
3.坑货来了
特别是NCBI的数据上传之前不管是序列格式还是.gff上传之前一定检查完整性,要么直接
gunzip xxxx.fna.gz
gunzip xxxx.gff.gz
千万别在NCBI的ftp服务器中直接一步到自己的结果上,没有数据完整性就会报错:
我忘了。。。。
FATAL ERROR: Most Exons do not have sequences!
遇见这个报错头都不用回,很多人做大一点的基因组都遇见了,是因为坑货NCBI的ftp不稳定下载的压缩包不完整导致的,这个时候就考虑你的参考基因组数据的问题。
最后其他报错都在大佬的博客里体现过了,这儿我自己踩了一坑做个记录吧。
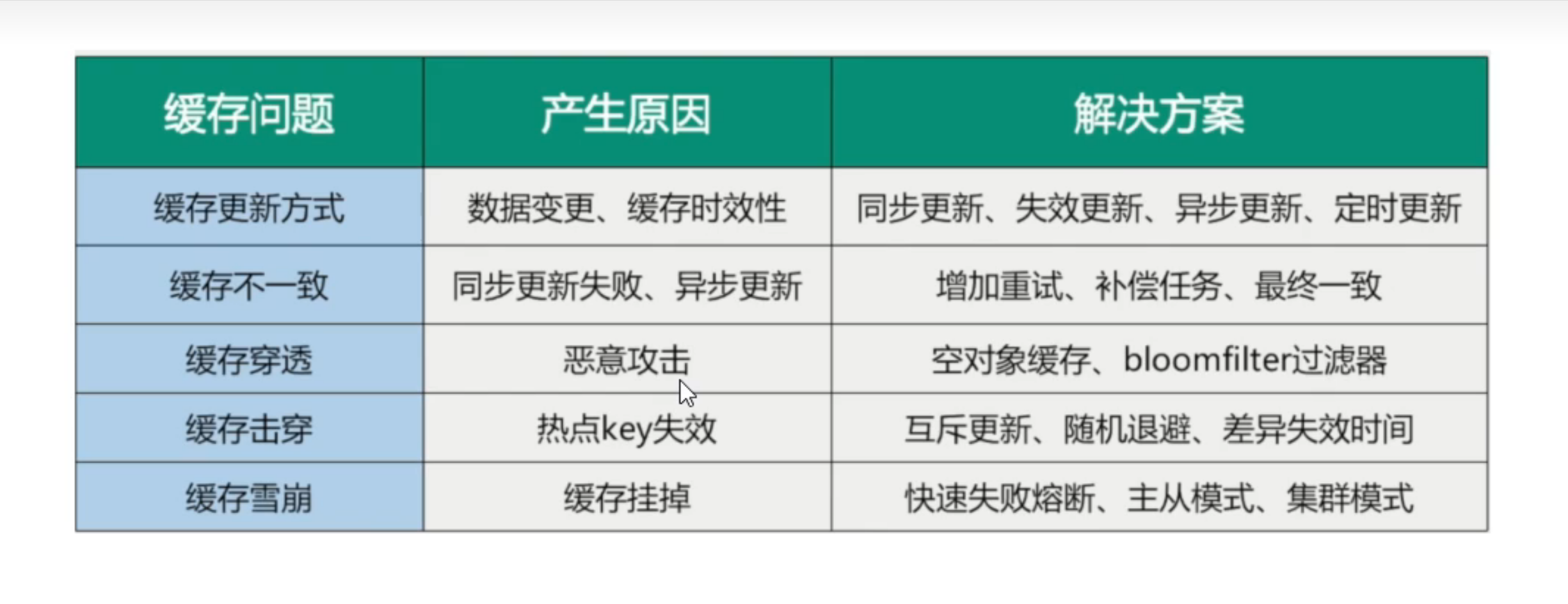
4.结果文件判读
SnpEff生成的结果文件(通常是VCF格式)的每一列都包含了不同的信息,用于描述和注释基因组变异。下面是VCF格式中常见的列及其含义:
CHROM:表示变异所在的染色体名称或编号。
POS:表示变异在染色体上的位置(1-based,即从1开始计数)。
ID:变异的唯一标识符,可以是rs号(如果变异已被记录在数据库中)或其他独特的标识符。
REF:表示参考基因组上的碱基。
ALT:表示变异的碱基替代(可能有多个,以逗号分隔)。
QUAL:表示变异的质量分数,用于衡量变异的可信度。
FILTER:表示变异是否通过了质量过滤(例如,PASS表示通过,其他值表示未通过)。
INFO:这是一个关键的列,包含了许多关于变异的附加信息,通常以键值对的形式存在。在SnpEff的结果中,常见的INFO字段可能包括:
EFF:变异效应预测结果,描述变异对基因功能的影响。
ANN:类似于EFF字段,提供变异效应预测结果,但使用不同的分隔符。
LOF_INFO:提供低功能性变异的注释信息。
其他自定义的注释字段,可以根据SnpEff的设置和用户需求添加。
FORMAT:如果VCF文件包含了多个样本的变异信息,这一列描述了每个样本的数据格式。
样本列:每一列代表一个样本,包含了该样本中每个变异的基因型信息。这些信息可以包括参考基因型、替代基因型以及可能的基因型质量等。
5.小tips
今天本想着走捷径,数据获取的途径走快速方法,结果偷鸡不成反倒蚀把米。或许人生亦是如此,有些时候真就聪明反被聪明误。结果一天时间都搭进去了,想想真是不值,不知道在内地怎么样,反正在新疆NCBI的ftp下载文件那是断了又断直接不给你活路,直到我点开了
这个小猫咪,我的问题才得以解决。。。。