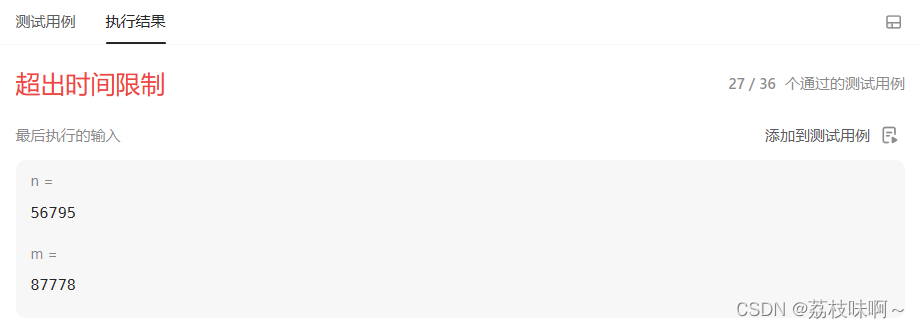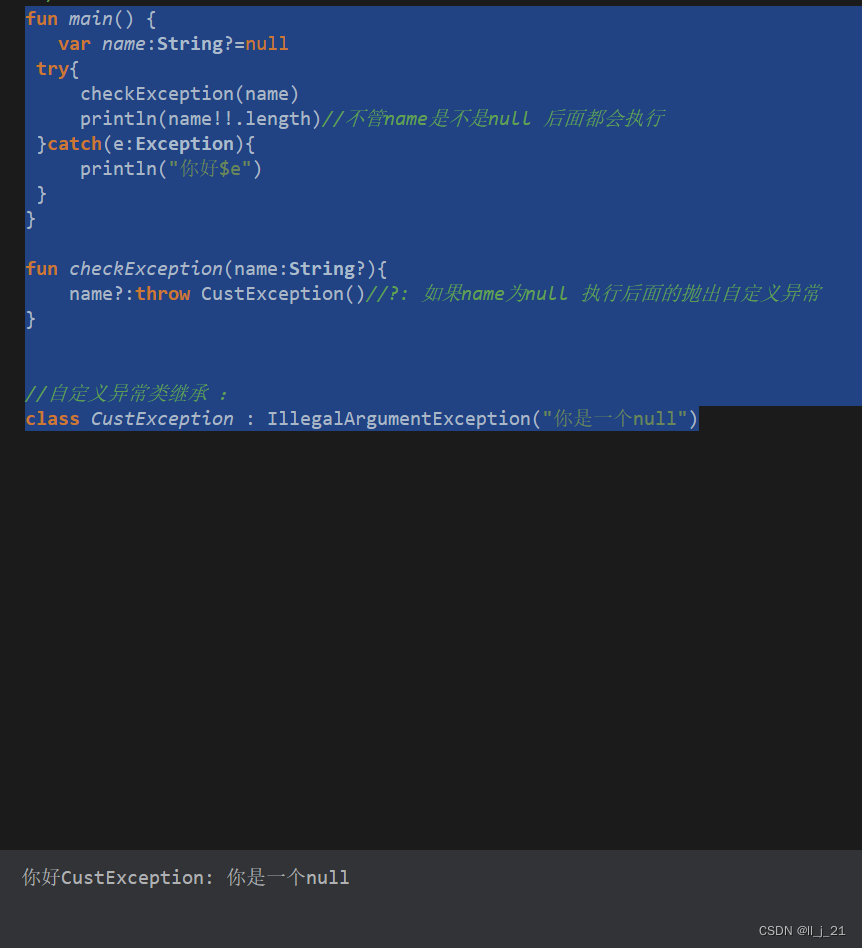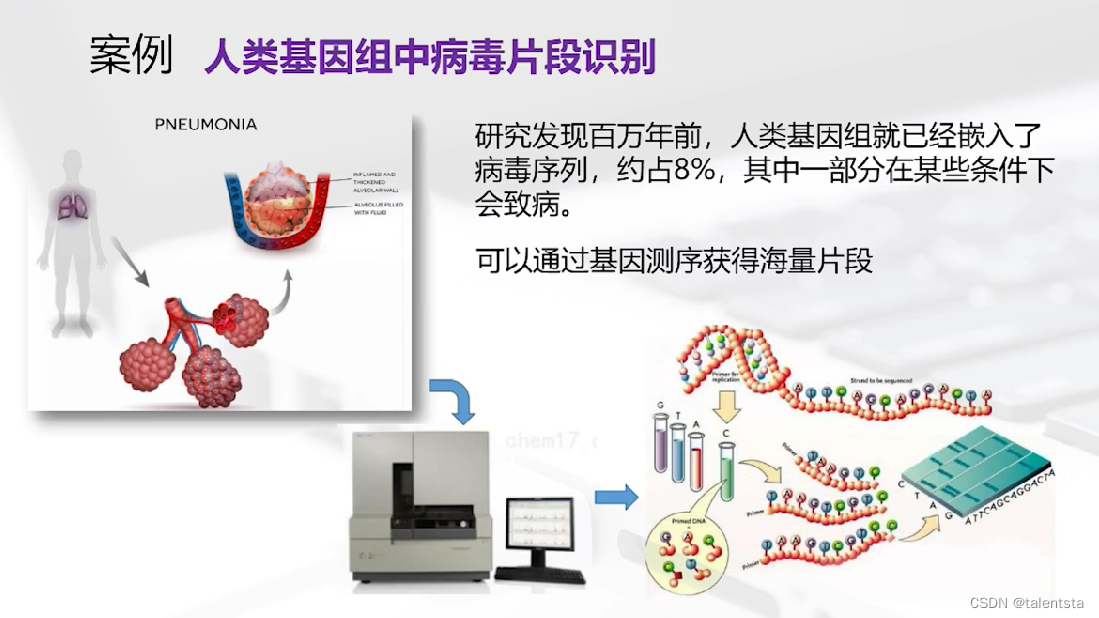
百万年前人类基因组中基因组中就已经嵌入了病毒序列,其中一部分在某些条件下会致病,通过基因测序获得海量片段之后就可以判断正常基因和病毒序列了。
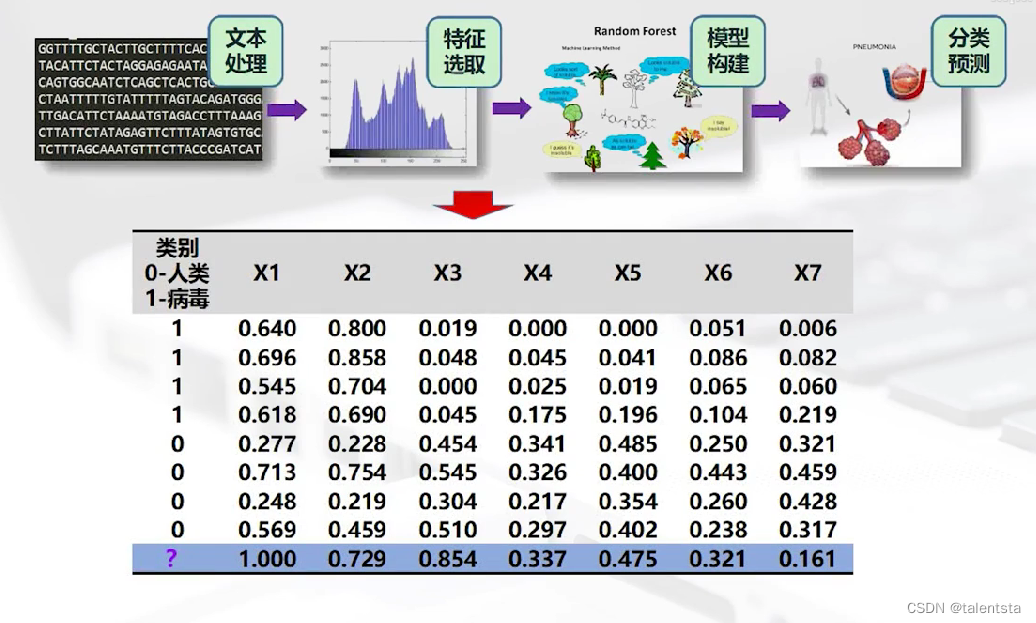
我们根据这种包含众多碱基的基因测序结果从中选取部分特征,关于特征的选取也是有好有坏的,如何能选取到更好的反映样本准确性的结果很重要,这里我们已经选取了部分特征作为构建模型的数据集。

集成学习就是通过构建并结合多个机器学习器来完成学习任务,通过多个基学习器分别产生不同的结果再从中选择结果中的众数来判定结果。
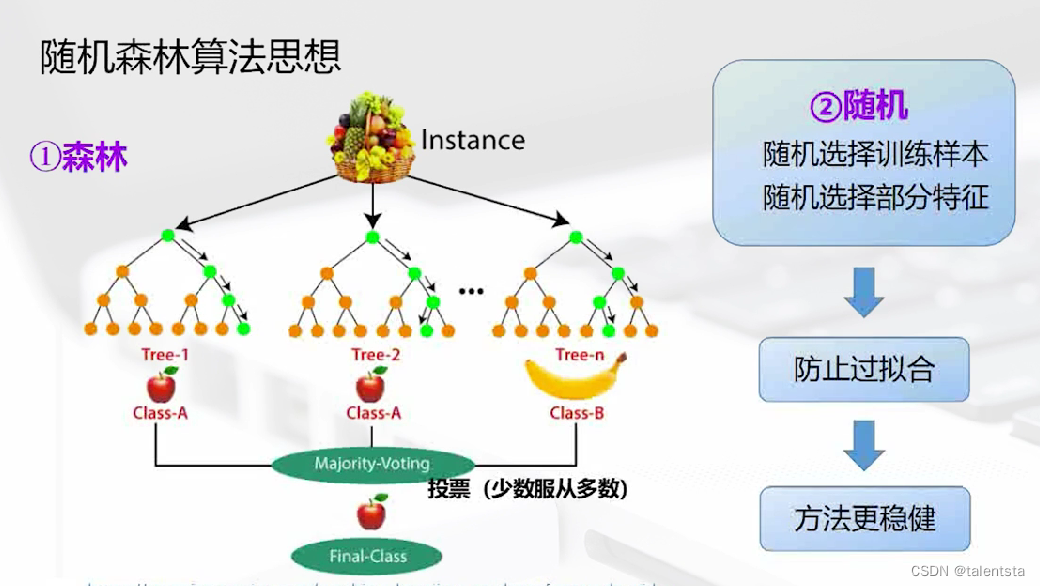
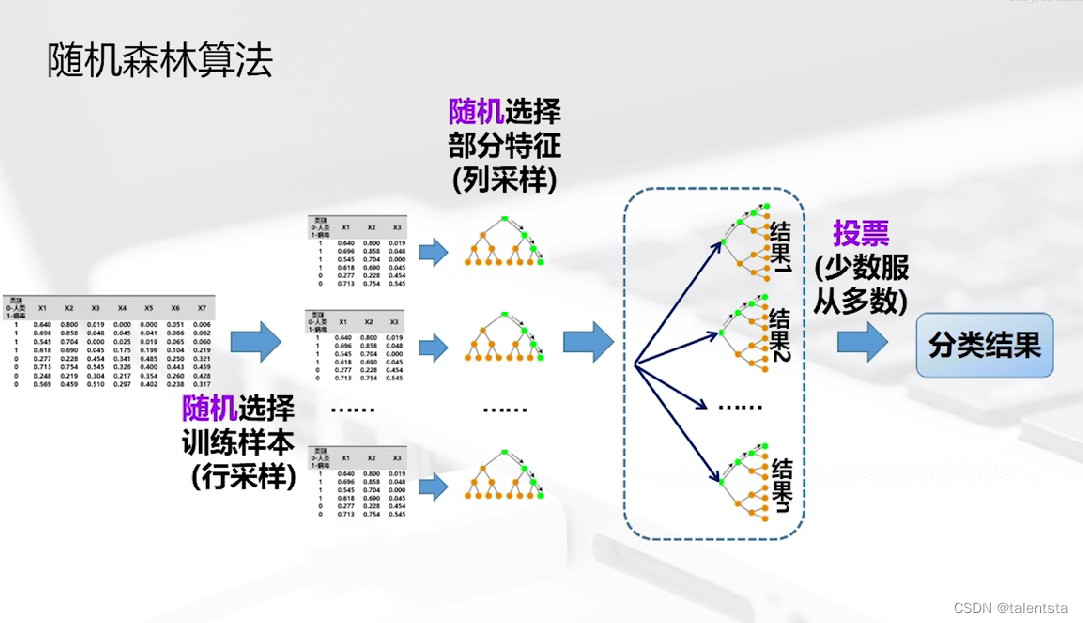
随机森林的算法思想是构建多棵决策树然后根据少数服从多数的原则选出最终的实验结果,这里的随机有两个含义,一是随机选择训练样本,训练样本并不是全部用来训练;二是随机选择部分特征,并不是选用全部的特征,选取其中一部分特征来进行训练,两个随机的目的都是为了防止过拟合,这样的结果更有稳健性。
'''step1 调用包'''
import pandas as pd
from sklearn.model_selection import train_test_split
#随机森林
from sklearn.ensemble import RandomForestClassifier
#调用准确率计算函数
from sklearn.metrics import accuracy_score
'''step2 导入数据'''
data = pd.read_excel('data_virus.xlsx')
'''step3 数据预处理'''
# 把带类标号数据(用于训练和检验)
# 和待判(最后5行)数据分开
data_used = data.iloc[:112,:]
#Python从0开始计数,故上一行代码从第0行取到第77行
#共提取了112行。
#最后5行为待判集合
data_unused = data.iloc[113:,:]
'''step4 划分数据集'''
#(将带类标号数据#划分为训练集(75%)
#和检验集(25%)
#将类别列和特征列拆分,
#便于下面调用划分函数
y_data=data_used.iloc[:,0]
x_data=data_used.iloc[:,1:]
#调用sklearn中的函数划分上述数据
x_train, x_test, y_train, y_test = train_test_split(
x_data,y_data,test_size=0.25,random_state=0)
'''step5 模型计算(训练、检验、评价)'''
#step5.1 训练模型
model_RF = RandomForestClassifier()
model_RF.fit(x_train, y_train)
#step5.2 检验模型
pred_test = model_RF.predict(x_test)
#step5.3 模型评价(准确率)
#这里y_test为真实检验集类标号
#pred_test为模型预测的检验集类标号
#比较二者即可得到准确率
acc_test = accuracy_score(y_test,pred_test)
print('检验准确率为:',acc_test)
'''step6 预测结果'''
#模型训练完,检验效果满意,即可对最后5行
#未知类别样本进行预测(分类)
x_unused = data_unused.iloc[:,1:]
pred_unused = model_RF.predict(x_unused)
print('待判样本预测类别为:0-人类/1-病毒\n',pred_unused)
我们可以看到这里调用包选择了从 sklearn 中选择了 RandomForestClassifier() 作为训练的模型,
- 导入需要的库:pandas、sklearn等
- 导入数据:读取Excel中的数据到pandas DataFrame
- 数据预处理:将带类标的数据和待预测数据分开
- 划分数据集:将带类标的数据分割为训练集和测试集
- 模型计算:
- 训练随机森林模型
- 在测试集上测试模型,计算准确率
- 输出测试集准确率
- 预测结果:
- 用训练好的模型对待预测数据进行预测
- 输出预测的类别结果
主要步骤包括:
- 导入数据
- 数据预处理
- 划分训练测试集
- 使用随机森林模型训练
- 在测试集上测试模型
- 用模型对新数据进行预测