临床数据分析方案
桓峰基因公众号推出临床数据分析方案,整理如下:
临床数据 1. 临床基因突变数据如何发高分?
临床数据 2. 基于NGS的胃癌诊疗全过程的临床应用方案
临床数据 3. 肿瘤微小残留病灶(MRD)如何发文章?
克隆进化生信分析教程
我们在桓峰基因公众号也推出克隆进化分析教程,从组织到单细胞的克隆进化分析都有介绍,供大家参考:
Clone 1. 肿瘤克隆进化之前世今生
Clone 2. 肿瘤克隆进化之不同进化模
Topic 1.克隆进化之sciClone
Topic 2.克隆进化之ClonEvol
Topic 3. 克隆进化之 fishplot
Topic 4. 克隆进化之 Pyclone
Topic 5. 克隆进化之 CITUP
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
Topic 9. 克隆进化之 TimeScape
克隆进化分析报告
准备基因组检测结果
1. CNV 检测的结果
可以通过多种软件获得,来源一般三种方式:
WGS
WES
Panel
Note: 没有也没办法
CNV 格式要求必须有这4列,如下:
1. chromosome
2. segment start position
3. segment stop position
4. copy number value for that segment.
Note: Unrepresented regions are assumed to have a copy number of 2
举例说明,如下:
Chrstartend value
11 315000 1.293159
1315001 50150000 2.269226
1 5015000151060000 2.521881
1 5106000170690000 2.231479
1 7069000170720000 1.930534
1 70720001 121185000 2.215604
2. SNV 检测的结果
可以通过多种软件获得,来源一般三种方式:
WGS
WES
Panel
Note: 没有就做不了哦!!!
SNV 格式要求必须有这5列,如下:
1. chromosome
2. position
3. reference-supporting read counts
4. variant-supporting read counts
5. variant allele fraction (between 0-100)
其他:Gene, driver gene.
举例说明,如下:
ChrPos RefDepth AltDepth VAF Gene is.dirver
chr12488153 1 352 99.44 TNFRSF14 FALSE
chr18073500 0 0 0.00 ERRFI1 FALSE
chr18073519 0 0 0.00 ERRFI1 FALSE
3. LOH检测的结果
可以通过多种软件获得,来源一般三种方式:
WGS
WES
Panel
没有也没办法
LOH格式要求必须有这3列,如下:
1. chromosome
2. window start position
3. window stop position
Note:属于这些窗口的单核苷酸变异将不包括在分析中。
举例说明,如下:
18020922 9987323
生信分析流程
克隆进化整个生信分析流程分成三部分:sciClone + clonevol + fishplot
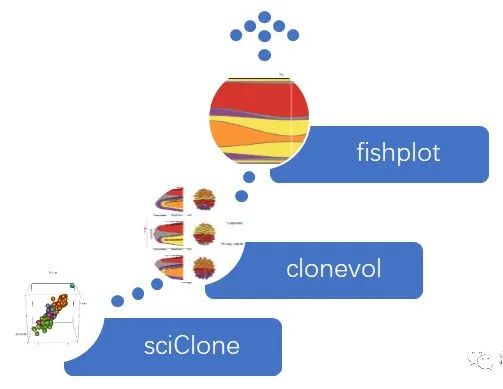
1. sciClone 产出结果解读
a) 两两样本比较聚类结果:
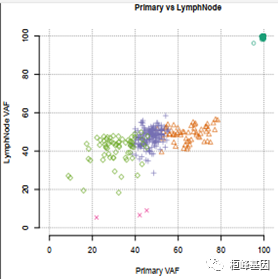
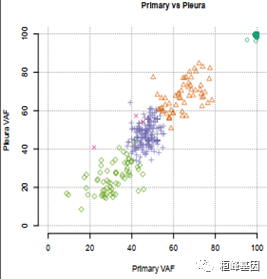
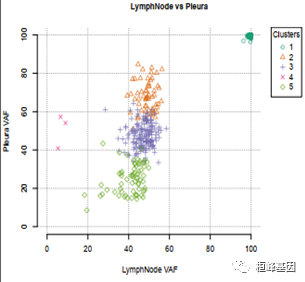
b) Table1. 聚类后信息表
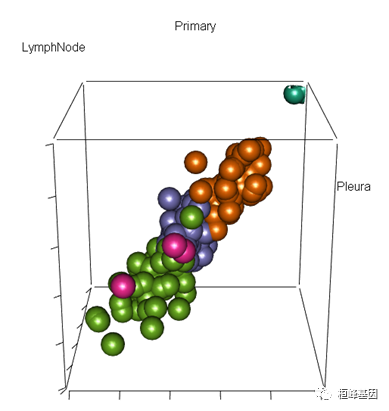
c) 三维样本结果
2. clonevol 产出结果解读
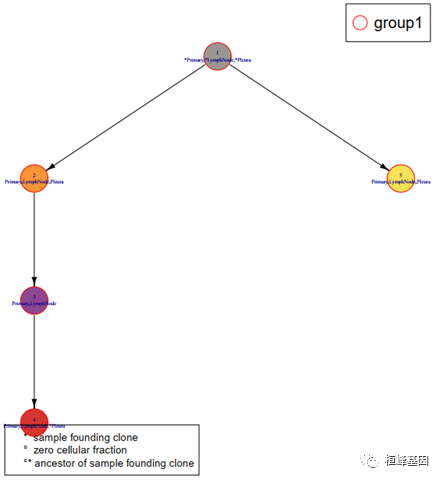
a) 绘制进化树
b) 聚类flow图
c) 样本聚类两两比较
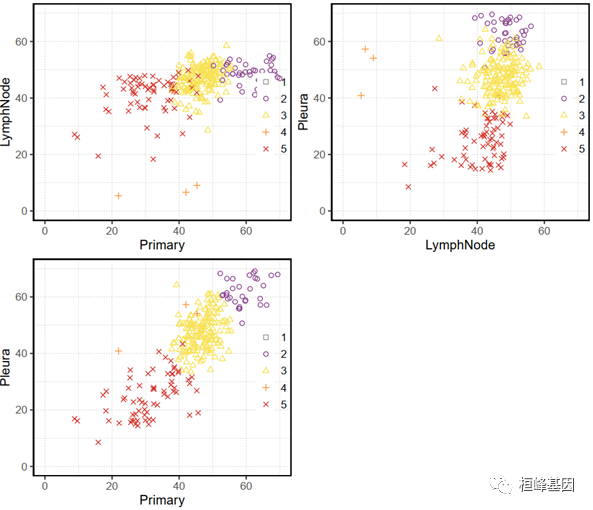
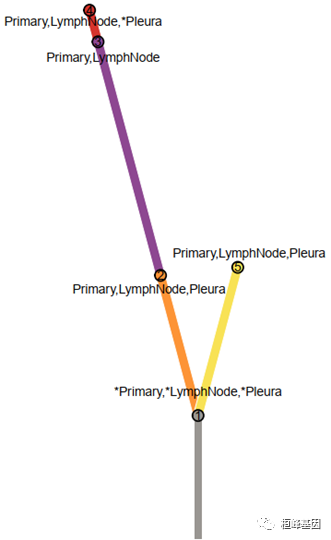
d) 所有克隆的进化树
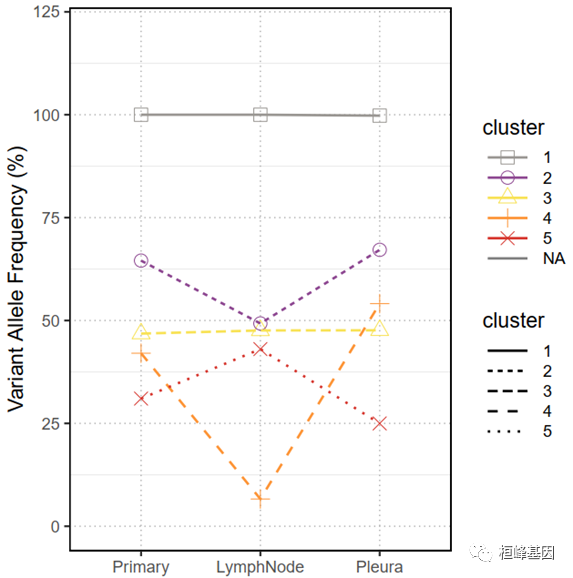
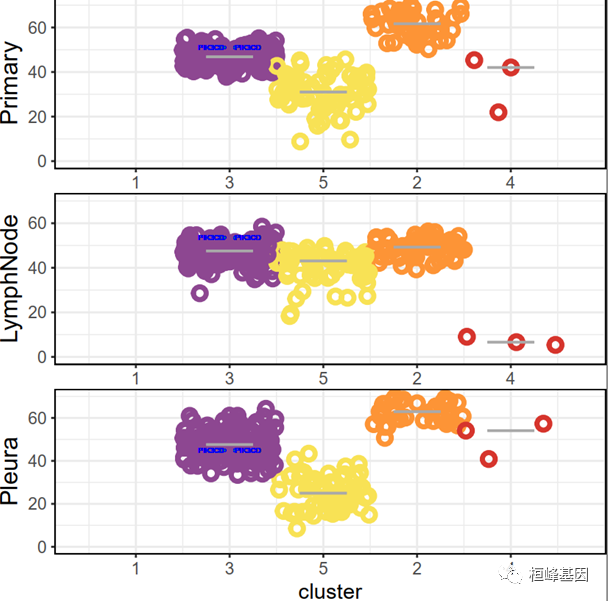
e) 样本VAF在不同cluster中的分布
f) 综合结果分五部分内容
1. 每个样本在不同cluster中的突变频率;
2. 每个样本的贝尔图;
3. 每个样本的细胞总体分布;
4. 基于节点的一致树;
5.基于分支的一致树。

3. fishplot 产出结果解读
结果显示肺癌的进化模式:转移性多克隆支型进化模式
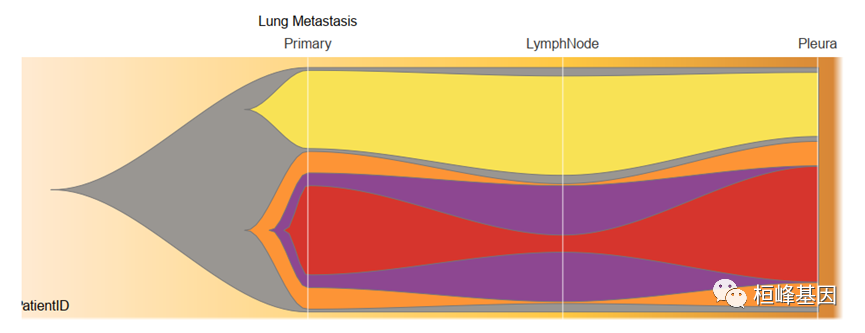
实例分析
这里我们分享一篇2012年五月发表于 Nature(IF:11.059)。作者基于全基因组测序研究血液病AML复发的克隆进化,该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!

摘 要
大多数急性髓系白血病 (AML) 患者在复发后死于进展性疾病,这与细胞遗传学水平的克隆进化有关1,2。确定突变与复发相关的谱系,我们对原发性肿瘤进行了测序以及来自八名 AML 患者的复发基因组,并经过验证使用深度测序的数百个体细胞突变;这允许我们精确地定义克隆性和克隆进化模式复发。除了发现新的、反复突变的基因(例如,WAC,SMC3,DIS3,DDX41和DAXX)在AML中,我们还发现了AML复发期间的两种主要克隆进化模式:
(1)原发性肿瘤中的创始克隆获得突变和进化为复发克隆
(2)原始的亚克隆克隆在初始治疗中幸存下来,获得了额外的突变在复发时扩大。
在所有病例中,化疗都未能根除创始克隆。复发特异性与复发特异性的比较所有八例病例的原发性肿瘤突变均显示增加在转位中,可能是由于细胞毒性引起的 DNA 损伤化学疗法。这些数据表明,AML复发与新突变和克隆进化的增加有关,这部分是由患者的化疗决定的接受以建立和维持缓解。
生信分析流程
相关数据集选择:
Patients:8例AML患者的原发和复发配对样本
测序:WGS和Capture array design
基因集选择:突变基因结果列表
生信分析方法:
我们从文章的分析流程中提取所有的分析内容,整理出来就 7个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于生信分析的文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
1. 使用 BWA 0.5.5 将 PE 比对到 NCBI build36;
2. 使用 SomaticSniper and SAMtools indel caller 检查体细胞突变位点;
3. 使用 BreakDancer 识别 SV;
4. 使用 GATK and Pindel detecting indels
5. 使用 VarScan 2 识别胚系突变;
6. 原发肿瘤和复发对亚克隆的核密度估计(sciClone)
7. 克隆进化鱼图(fishplot)
研究结果
1. 基于NGS在8个原发和复发配对的急性髓系白血病患者中捕获体细胞突变位点。
a,在八例病例中检测到的1-3级突变位点(不是包括易位)。显示的所有突变均使用捕获进行验证其次是深度测序。共享突变为灰色,原发性肿瘤特异性突变为蓝色,复发特异性突变为红色。合计每个病例的 1-3 级突变数显示在浅灰色上方矩形。
b,来自验证突变的突变等位基因频率分布原发性肿瘤的 1-3 级和病例 UPN 933124的复发(左)。突变体获得五个原发肿瘤特异性突变的等位基因频率来自 454 深度读取计数实验。鉴定出4个突变簇在原发性肿瘤中,复发时发现一个。五个低水平突变在原发性肿瘤和复发中(包括 4 例已知拷贝数量变量区域)被排除在聚类分析之外。来自 AML 中反复突变的基因的非同义突变是显示。突变的突变等位基因频率的变化来自五个突变簇状物显示在原发肿瘤和复发之间(右)。
c,在来自另外七名 AML 患者的原发性肿瘤和复发样本。这原发肿瘤簇与复发样本之间的关系由连接它们的线表示。
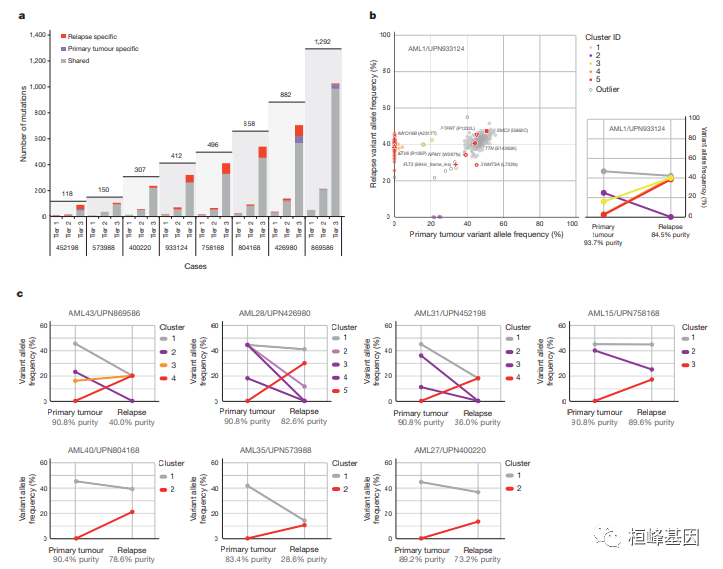
2. 在八个原发性肿瘤-复发对中鉴定出的编码突变

3. 从初级克隆进化的图形表示UPN 933124中肿瘤复发以及肿瘤演变模式
在八个原发性肿瘤和复发配对中观察到。
a,创始克隆UPN 933124的原发肿瘤包含DNMT3A、NPM1、PTPRT、SMC3 和 FLT3 在 AML 和 AML 中反复出现可能与发病机制有关;创始克隆中的一个子克隆
进化为通过获得额外的突变,包括 ETV6 和 MYO18B 的复发性突变,以及WNK1-WAC融合基因。
b, 示例AML肿瘤演变的两种主要模式。模型 1 显示占主导地位原发性肿瘤中的克隆通过获得复发特异性突变进化为复发克隆;这种模式在三个原发性肿瘤和复发对(UPN 804168、UPN 573988 和 UPN 400220)。模型 2 显示携带绝大多数原发性肿瘤突变的小克隆复发时存活并扩大。在五个主要肿瘤和复发对(UPN 933124、UPN 452198、UPN 758168、UPN426980 和 UPN 869586)。
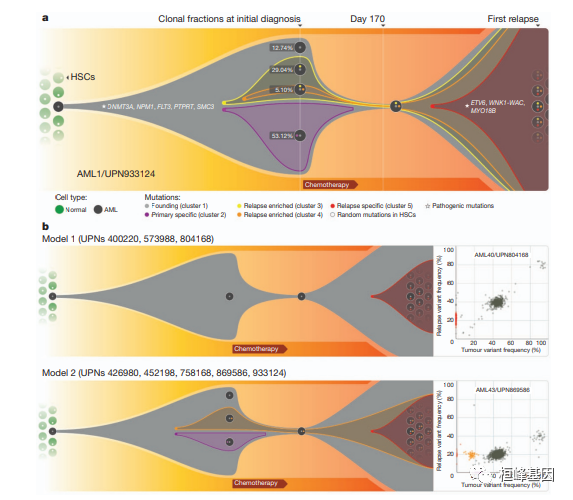
4. 原发性肿瘤和复发样本。
a,原发肿瘤的比例和复发特异性每个转换和转化类别中的突变。
b, 转换原发肿瘤的频率和复发特异性突变来自八个AML 肿瘤和复发对。456 个复发特异性突变和 3,590 个8例原发性肿瘤突变用于评估统计学使用比例检验的显著性
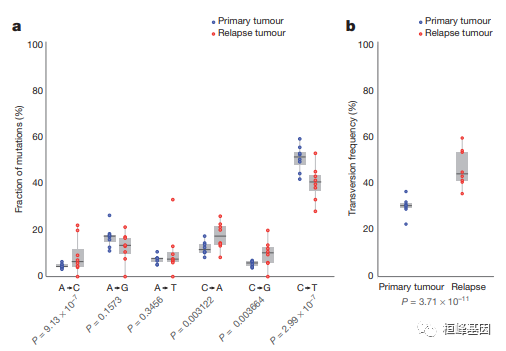
Reference:
1. Miller CA, White BS, Dees ND, et al. SciClone: inferring clonal architecture and tracking the spatial and temporal patterns of tumor evolution. PLoS Comput Biol. 2014;10(8):e1003665. Published 2014 Aug 7.
2. Dang, H. X., White, B. S., Foltz, S. M., Miller, C. A., Luo, J., Fields, R. C., & Maher, C. A. (2017). ClonEvol: clonal ordering and visualization in cancer sequencing. Annals of Oncology, 28(12), 3076-3082.
3. Miller CA, McMichael J, Dang HX, Maher CA, Ding L, Ley TJ, Mardis ER, Wilson RK. Visualizing tumor evolution with the fishplot package for R. BMC Genomics. 2016 Nov 7;17(1):880.
4. Ding, L., Ley, T., Larson, D. et al. Clonal evolution in relapsed acute myeloid leukaemia revealed by whole-genome sequencing. Nature 481, 506–510 (2012).
这篇文章利用桓峰基因公众号上的教程完全可以实现这些图表的绘制,过程需要自己细致的探究,同时也需要有一定的生信分析和结果解读的能力,有需要此类分析的老师可以联系桓峰基因!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!
http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
有想进生信交流群的老师可以扫最后一个二维码加微信
备注“单位+姓名+目的”!