玩转单细胞往期精彩系列:
玩转单细胞(2):Seurat批量做图修饰
玩转单细胞(3):堆叠柱状图添加比例
玩转单细胞(4):单细胞相关性
玩转单细胞(5):单细胞UMAP图只标记特定细胞群、圈定细胞群及坐标轴修改
玩转单细胞(6):单细胞差异基因展示之对角散点图
玩转单细胞(7):修改Seurat对象基因名称
玩转单细胞(8): 单细胞3维聚类图展示
玩转单细胞(9):单细胞Seurat对象数据操作
又来到玩转单细胞系列了,这个系列虽然很小很简单,但都是一些实用性的问题。今天这个帖子的起因是这样的。有小伙伴说自己在GEO数据库上看中了一个单细胞数据,作者提供了样本的表达矩阵,还提供了注释好celltype和坐标信息的metadata。小伙伴利用这个数据并不是想重新分析,而是想要原文作者一模一样的聚类降维,然后进行一些比较,所以说是想还原这个数据。如果不是原文作者提供了包含cell type和细胞坐标信息的metadata,那么还真不能,但是有数据就好办了。
解决这个问题很好办,那就是自己降维后替换UMAP坐标信息。为了保护粉丝数据,所以这里我们另找了一篇文章的数据集进行演示。参考文献:Deciphering human macrophage development at single-cell resolution!本文详细注释代码已上传群文件!
首先我们在GEO下载数据,读入数据预处理:
setwd('D:/KS项目/公众号文章/Seuat替换UMAp')#===============================================================================#读入数据library(ggplot2)library(Seurat)meta <- read.table('GSE133345_Annotations.txt', header = T, row.names = 1)folders=list.files('./GSE133345_RAW/',pattern='^[GSM]')foldersExp_matrix = lapply(folders,function(x){read.table(paste0('./GSE133345_RAW/',x),header = T, row.names = 1)})#批量读入names(Exp_matrix) <- substr(folders, 1, 10)scelist <- list()for (i in 1:length(Exp_matrix)) {sce <- CreateSeuratObject(counts = Exp_matrix[[i]])sce$sample <- names(Exp_matrix)[i]scelist[[i]] <- sce}#merge datasce = merge(scelist[[1]],y = c(scelist[[2]],scelist[[3]],scelist[[4]],scelist[[5]],scelist[[6]],scelist[[7]],scelist[[8]],scelist[[9]]))dim(sce)# [1] 26318 1268dim(meta)# [1] 1231 7sce1 <- sce[,rownames(meta)]dim(sce1)
走标准的seurat流程:这样我们就拥有了具有UMAP降维的数据了!
#接下来就是标准流程了,至于其中的参数,差不多就行了,我们的目的主要是为了跑UMAP降维sce1 <- NormalizeData(sce1)sce1 <- FindVariableFeatures(sce1, nfeatures = 4000)sce1 <- ScaleData(sce1,verbose = T)sce1 <- RunPCA(sce1,npcs = 50, verbose = FALSE)sce1 <- RunUMAP(sce1, dims = 1:20)sce1 <- FindNeighbors(sce1, dims = 1:20)sce1 <- FindClusters(object = sce1 , resolution = 1, verbose = FALSE)DimPlot(sce1, reduction = 'umap', label = T)+theme_bw()+theme(panel.background = element_blank(),panel.grid = element_blank())
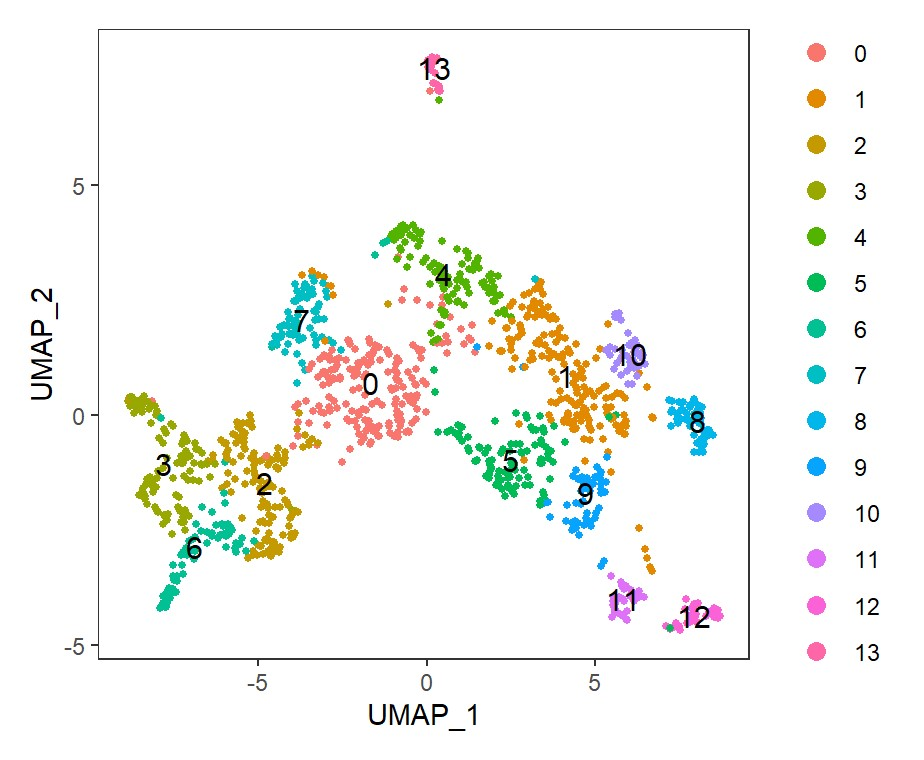
看这个图和原文还有差距比较大的,我们将原文作者的celltype信息合UMAP坐标信息替换一下。
#保证meta和seurat obj两者的barcode一致sce1$cellid <- rownames(sce1@meta.data)meta = meta[sce1$cellid,]umap1 <- meta$UMAP1names(umap1) <- rownames(meta)sce1@reductions[["umap"]]@cell.embeddings[,1] <- umap1umap2 <- meta$UMAP2names(umap2) <- rownames(meta)sce1@reductions[["umap"]]@cell.embeddings[,2] <- umap2sce1@meta.data <- cbind(sce1@meta.data, meta)Idents(sce1)='cluster'DimPlot(sce1, reduction = 'umap', label = F, repel = T)+theme_bw()+theme(panel.background = element_blank(),panel.grid = element_blank())
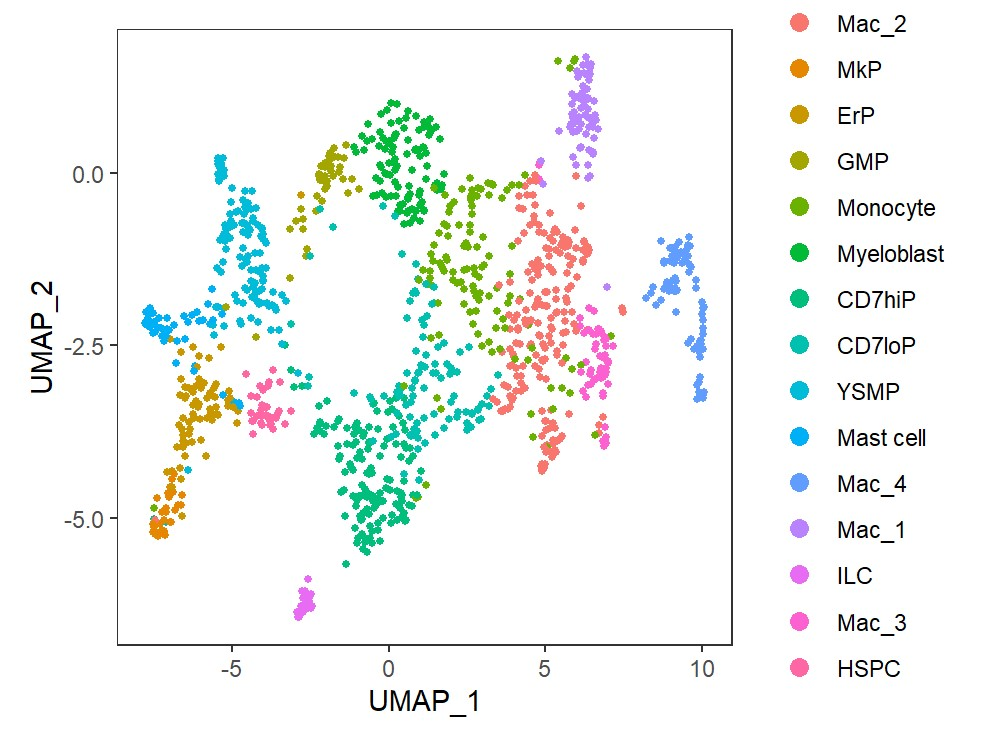
原文的UMAP降维图:

可以看到结果是一模一样,这样就可以进行后续的分析了。至于UMAP的修饰,我们在之前的帖子已经讲过了:Nature作图也出错:单细胞UMAP/TSNE图的ggplot做法与修饰。好了,这就是所有内容了,觉得分享有用的,点个赞、分享下再走呗!