文章目录
- 1. 写在前面
- 2. 手动查找和探索过程
- 从UCSC查找
- 从Ensembl查找
- 3. 代码实现
1. 写在前面
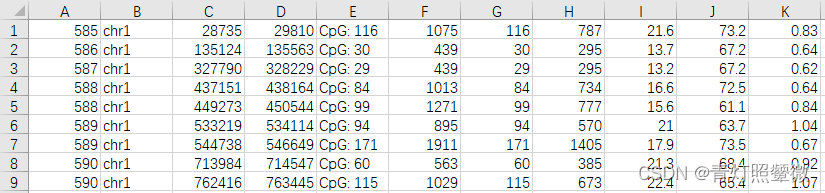
一篇专利 1 中提到多种癌种及对应的特异性CpG位点,想获取对应cg位点具体的位置或序列。专利中的一组CpG markers如下:

需求就是:将这些cg编号作为文件输入,获取对应的序列和位置信息。
2. 手动查找和探索过程
需求实现方式可直接到第3节:代码实现中直接查看实现代码,跳过本节。
从UCSC查找
由于之前未查询过cgxxx位置,直接网搜也找不到任何有效的信息,竟然在NCBI上也没有查到。
还是向ChatGPT2提问【怎么获取cgxxx(比如:cg01423964)对应的位置信息】,给出了有效信息。之所以说有效,是至少为我提供了查询的渠道(UCSC3),甚至还给出了具体的查询方式,比如下面:(步骤真详细差点信了!但是。。。)
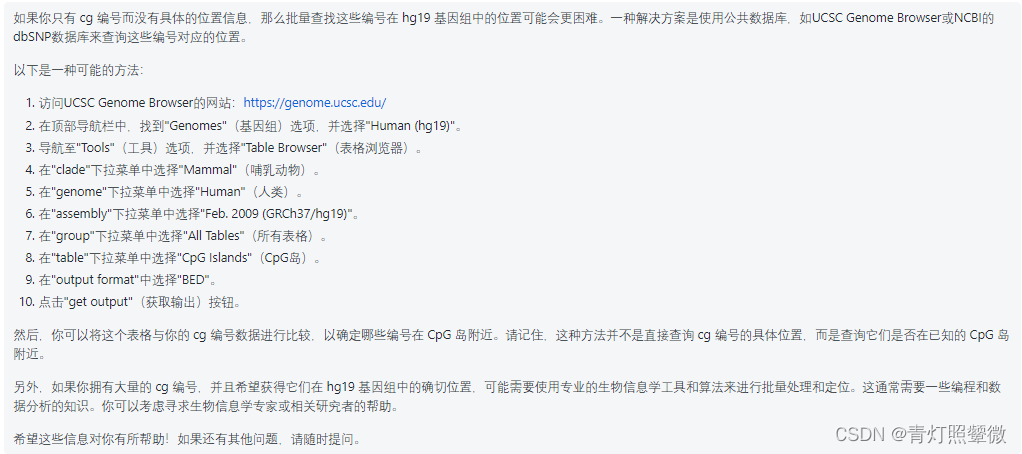
但是,请注意:上面的这个回答获取的不是cg编号,感兴趣的朋友也可以尝试上述步骤。获取的信息倒是有位置,但不是对应cgxxx。。。获取的这个文件就是这个地址:
http://hgdownload.soe.ucsc.edu/goldenPath/hg19/database/cpgIslandExt.txt.gz 。如下截图:
另外,上面得到的ChatGPT的回答也是我修改了10次以上提问才得到的答案,中间有些回答一些操作方式或者没有对应的选项问题等重新进行提问。一开始回答中有提供手动获取的方式(可参考):

如果只查询几个cgxxx的位置信息也可以手动获取,大概是这样的:
-
- 进入网址:http://genome.ucsc.edu/cgi-bin/hgSearch --> 输入cgxxx --> 点击"Search"。(注意选择参考序列版本,这里是GRCh37/hg19)
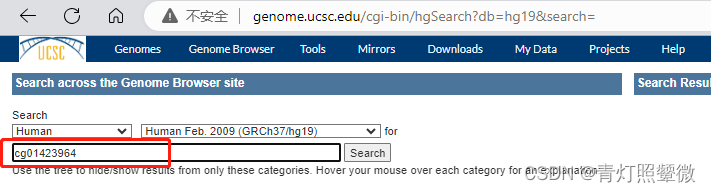
- 进入网址:http://genome.ucsc.edu/cgi-bin/hgSearch --> 输入cgxxx --> 点击"Search"。(注意选择参考序列版本,这里是GRCh37/hg19)
-
- 得到搜索结果:
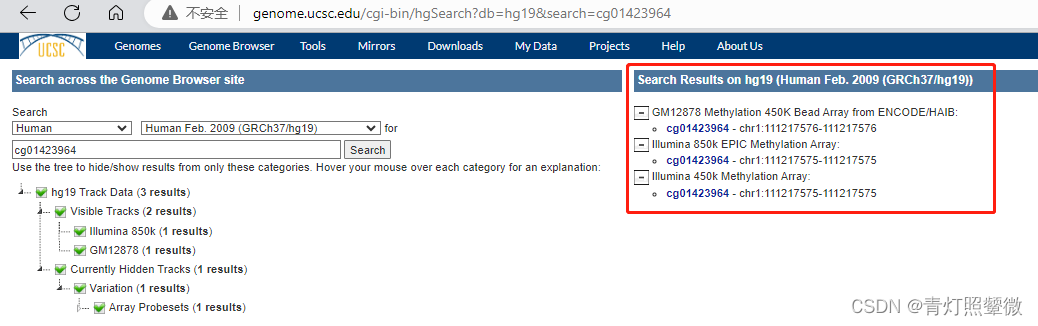
此时页面中可获取位置信息(一个CG点),也可以点击cgxxx(比如: "Illumina 450k Methylation Array"进入UCSC浏览器)查看该位置的具体信息:
- 得到搜索结果:
上述这个查询方式,可以直接在UCSC浏览器上面搜索框输入cgxxx点击"go",也可同样查询(有的会直接跳转到对应位置,有的不会。注意涉及位置时请确认参考基因组版本)
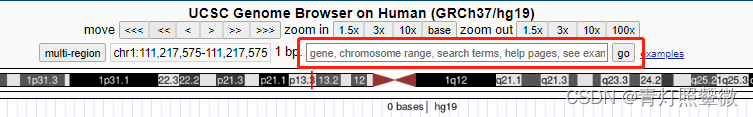
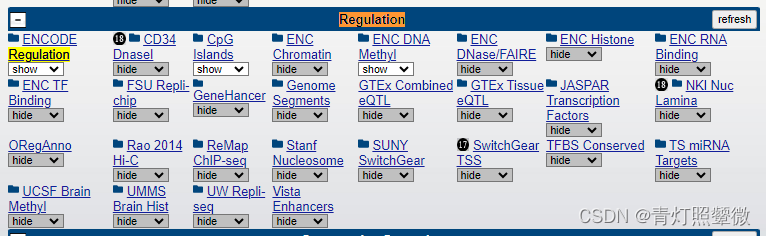
另外,如果UCSC搜索后下面显示的信息与上面截图不一致,比如没有显示cgxxx信息或其他信息,需要修改下面的Regulation或其他相关的按钮选项(hide/隐藏 或show/显示)
从Ensembl查找
在多次向ChatGPT提问后,ChatGPT建议还可以在 Ensembl4查询cgxxx对应位置。(貌似现在有问题时,问GPT比在浏览器搜的次数要多,因为ChatGPT搜寻的答案有条理且高效,虽然并不总是正确的)
Ensembl: https://grch37.ensembl.org/index.html
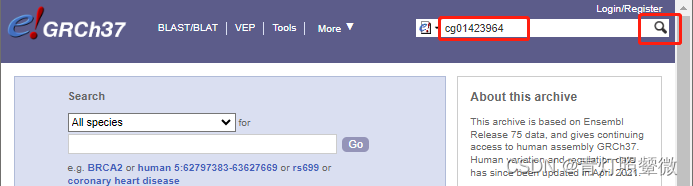
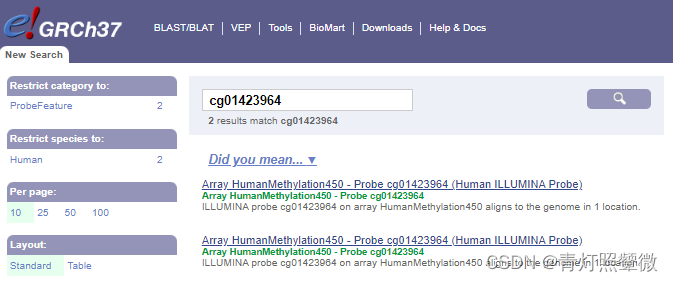
这个手动查找也算方便,搜索框输入cgxxx搜索即可(同样注意参考基因组版本),然后从搜索结果中查找具体的序列、位置信息。
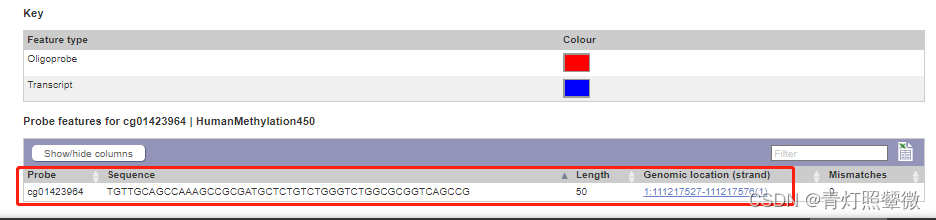
笔者没有在Ensembl上找到类似UCSC上可下载的甲基化位置信息文件,后续找到合适的方式就不用通过代码从网页获取了,从下载的所有CpG位点库中本地查找更方便。
下面代码实现是基于上面网页的搜索结果,从网页中html解析得到对应信息。
3. 代码实现
脚本命名:get_ensembl_cg_position.py
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
"""
date = '2023/07/14 0001'
author = 'QDZPW'
usage: python get_ensembl_cg_position.py ${cglist_file} ${output_file}
"""
from bs4 import BeautifulSoup
import requests
import sys
# 将获取的信息分割到具体位置
def split_loc(mystr):
# 1:111217527-111217576(1)
# 5:42994776-42994825(-1)
chrom_locs, strand = mystr.strip(')').split('(')
chrom, locs = chrom_locs.split(":")
start, end = locs.split('-')
new_strs = '\t'.join([chrom, start, end, strand])
return new_strs
# 根据html文件,获取相应信息
# eg html: https://grch37.ensembl.org/Homo_sapiens/Component/Location/Genome/genome?array=HumanMethylation450;fdb=funcgen;ftype=ProbeFeature;id=cg13788592
def extract_cginfo_from_html(html):
soup = BeautifulSoup(html, 'html.parser')
table = soup.find('table', id='ProbeFeature_table')
rows = table.find_all('tr')
result = []
for row in rows[1:]: # 跳过表头行
cells = row.find_all('td')
# cginfo_lst: found_cg_id, sequence, length, location. [found_cg_id: 网页获取的cg_id,保证与所查询的一致]
# cginfo_lst = [i.text.strip() for i in cells[:4]]
cginfo_lst = [i.text.strip() for i in cells[:4]] + [split_loc(cells[3].text.strip())]
result.append('\t'.join(cginfo_lst))
return result
# 批量查询位置信息
def get_cglist_info(cg_listfile, outfile):
# 读取包含cg编号的文件, 并写入结果到输出文件
with open(cg_listfile, 'r') as f, open(outfile, 'w') as pf:
# 输出各列:输入的cgxxx、查到的cg及对应的序列、长度、位置
# pf.write("#Input_cg\tProbe_cg\tSeuqence\tLength\tGenomic_location(strand)\n")
pf.write("#Input_cg\tProbe_cg\tSeuqence\tLength\tGenomic_location(strand)\tChrom\tStart\tEnd\tStrand\n")
for line in f:
cg_id = line.strip()
# 指定URL, 注意这里对应的是GRCH37版本的参考基因组
url = f"https://grch37.ensembl.org/Homo_sapiens/Component/Location/Genome/genome?array=HumanMethylation450;fdb=funcgen;ftype=ProbeFeature;id={cg_id}"
# 发送HTTP请求进行查询, 获取HTML内容
response = requests.get(url)
html = response.text
# 提取序列
result_info = '\t'.join(extract_cginfo_from_html(html))
pf.write(f"{cg_id}\t{result_info}\n")
if __name__ == "__main__":
cglist_file = sys.argv[1] # 每行一个cgxxx
out_file = sys.argv[2] # 输出文件
get_cglist_info(cglist_file, out_file)
使用方式:python get_ensembl_cg_position.py ${cglist_file} ${output_file}
上面代码,就是从html网页源代码中获取获取信息:
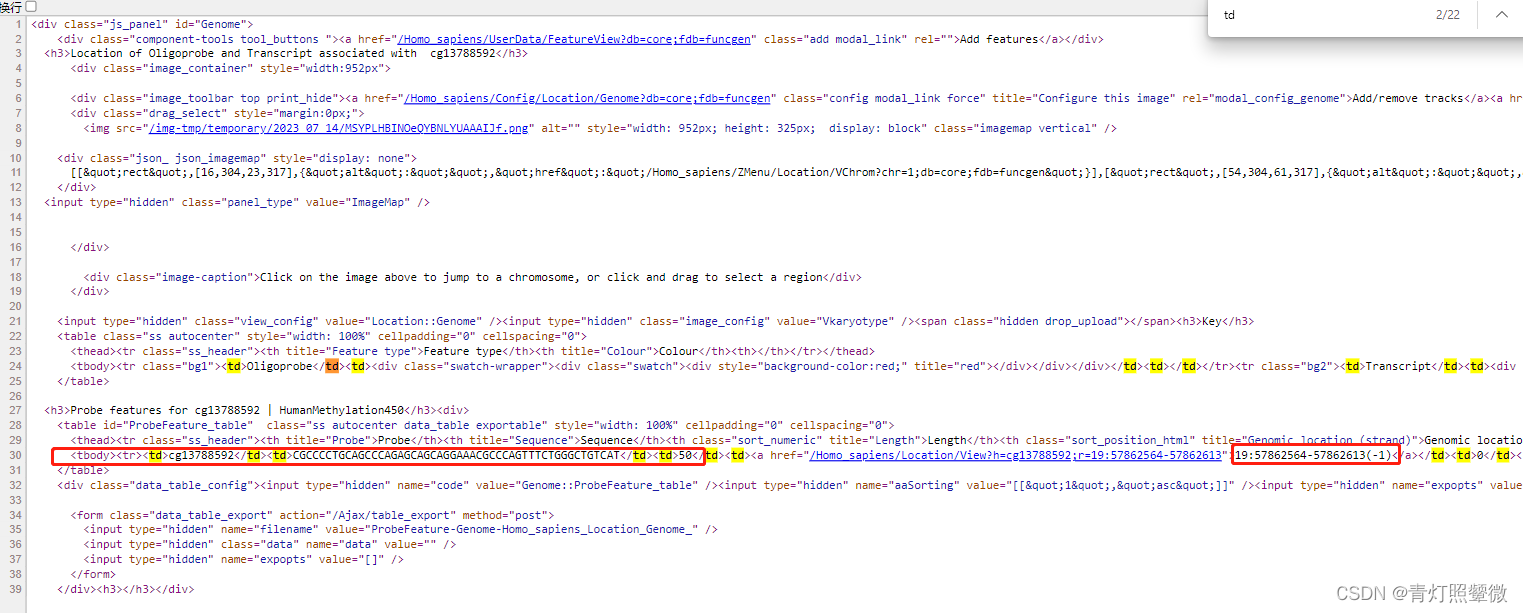
使用UCSC查找的方式,从网页源代码中也可用Ensenmbl代码类似的方式 获取cg对应的位置信息,只是少了序列信息。
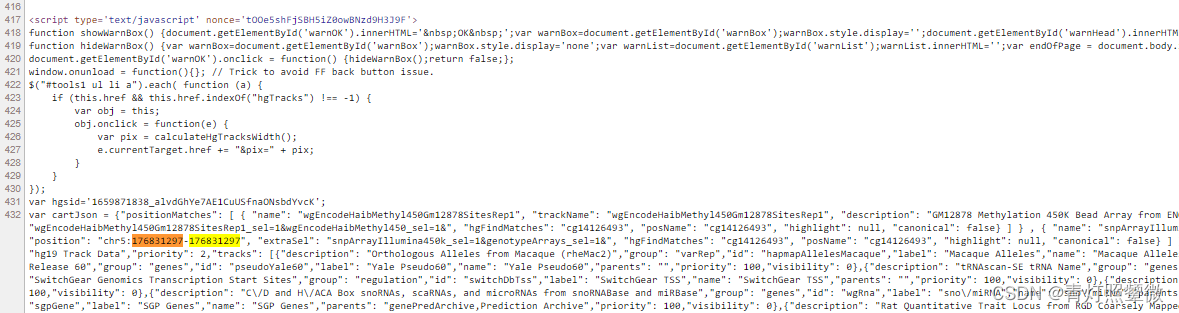
UCSC在线手动查找方式第二步获取的网页后查看其 源代码,根据html格式获取对应信息即可:
DNA METHYLATION MARKERS FOR NONINVASIVE DETECTION OF CANCER AND USES THEREOF: https://www.freepatentsonline.com/y2021/0171617.html ↩︎
ChatGPT: https://chat2.jinshutuan.com ↩︎
UCSC: http://genome.ucsc.edu/index.html ↩︎
Ensembl: https://grch37.ensembl.org/index.html ↩︎