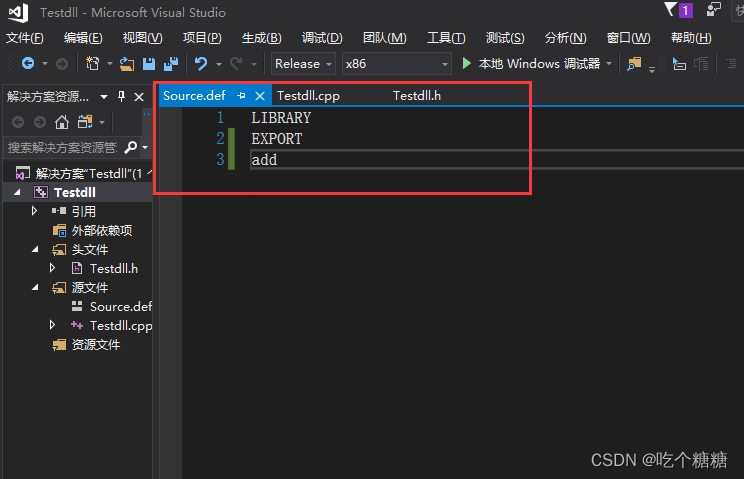
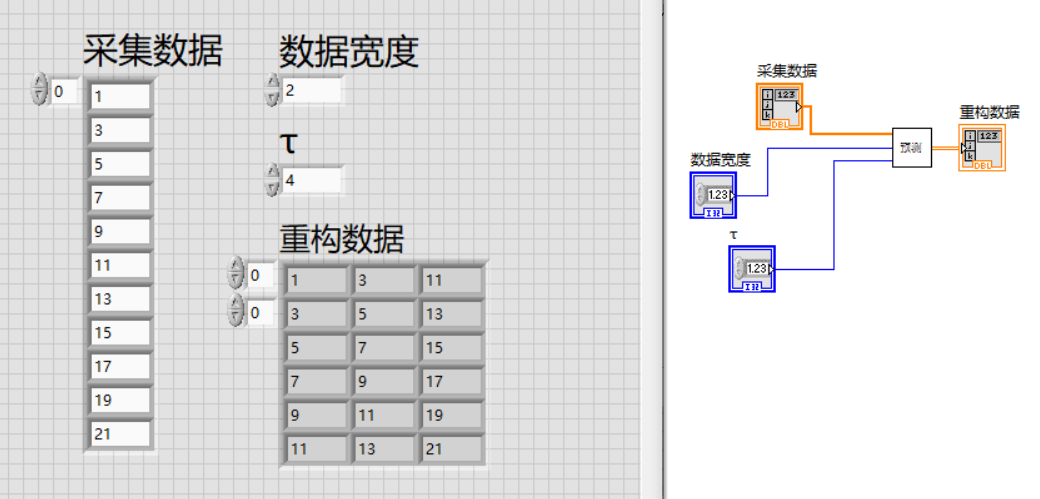
安装:
pip install simpletik
读取
目前主流的医疗图像格式是nifti格式,相比于dicom格式更加简单和更加容易读取和操作。后缀名为nii或者nii.gz(nii为原格式,gz结尾的是经过压缩格式,因为医疗图像的重复像素特别多,通过压缩空间可以节省很多存储空间)。
import Simpleitk as sitk # 通常取名为sitk 后续不再解释
path = r'自定义路径'
img = sitk.ReadImage(path)
arr = sitk.GetArrayFromImage(img)
arr是一个numpy数组,此时要注意索引顺序,在源图像中的索引顺序为x-y-z,arr中的索引顺序为z-y-x
保存
tem = sitk.GetImageFromArray(arr)
tem.SetDirection(img.GetDirection())
tem.SetOrigin(img.GetOrigin())
tem.SetSpacing(img.GetSpacing())
sitk.WriteImage(tem,r'要保存的路径和文件名')
arr经过业务处理后,保存到本地,上述代码为基础步骤,如果不进行2-4行代码,则保存的图像属性为默认值:spacinga为(1,1,1),origin为(0,0,0),具体默认值为下图所示.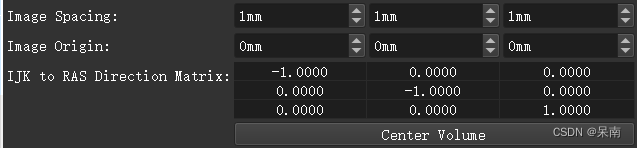
连通域
img = sitk.ReadImage(path)
cc = sitk.ConnectedComponent(img)
stats = sitk.LabelIntensityStatisticsImageFilter()
stats.Execute(cc, img)
arr = sitk.GetArrayFromImage(cc)
数组arr是经过连通域分析的numpy数组,每个连通域被赋予不同的标签.
stats.Getlabels() 是获取所有的标签,输出一个包含所有标签的列表,因此可以通过len(stats.Getlabels())获取连通域的数量.
stats.GetBoundingBox(标签值) 可以获得某个连通域的bonding box,返回一个包含六个整数值的元组,(min_x, max_x, min_y, max_y, min_z, max_z)
stats.GetCenterOfGravity(标签值) 返回某个连通域的重心
stats.GetCentroid(标签值) 返回某个连通域的质心
stats.GetPhysicalSize(标签值) 返回某个连通域的体素数量 通常来统计各个连通域大小的排名
stats.GetMaximum() 获得最大像素值 不可以是彩色图像
stats.GetMaximumIndex() 获得最大像素值的索引
stats.GetNumberOfPixels() 获取图像像素的总数量
下面是通过连通域分析处理杂质的例子
img= sitk.GetImageFromArray(path)
cc = sitk.ConnectedComponent(img)
stats = sitk.LabelIntensityStatisticsImageFilter()
stats.Execute(cc, img)
arr = sitk.GetArrayFromImage(cc)
max_dict = {i:stats.GetPhysicalSize(i) for i in stats.GetLabels()}
maxsize = max(max_dict.values())
rate = 0.2
for l in stats.GetLabels():
size = max_dict.get(l)
if size < maxsize * rate:
arr [np.where(arr == l)] = 0
tem = sitk.GetImageFromArray(arr)
tem.SetDirection(img.GetDirection())
tem.SetOrigin(img.GetOrigin())
tem.SetSpacing(img.GetSpacing())
sitk.WriteImage(tem,r'要保存的路径和文件名')
通过循环获得最大连通域的像素数量,设定阈值为0.2,将小于此数量的连通域去掉,并将结果保存
暂时写到这,以后有新想法再写,其实通过sitk.GetArrayFromImage获取numpy数组后,可以通过很多库对图像进行处理.例如sklearn nilearn skimage库等等.